Thermoproteus
| Thermoproteus | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Thermoproteus | ||||||||||||
| Zillig & Stetter 1982 |
Thermoproteus ist eine Gattung von Archaeen aus der Klasse Thermoprotei. Die Organismen dieser Gattung sind thermophil und schwefelabhängig. Sie sind mit den Gattungen Sulfolobus, Pyrodictium und Desulfurococcus verwandt.[1][2]
Beschreibung
Die stäbchenförmigen, begeißelten Mikroorganismen sind hyperthermophil: sie wachsen bei Temperaturen von bis zu 95 °C. Sie sind Schwefelwasserstoff-autotrophe Anaerobier – sie wachsen in der Natur durch die Reduktion von Schwefel; aber in Kultivierung wachsen sie per Schwefelatmung viel schneller. Ihre Zellmembran besteht aus besonderen Lipiden, die Glycerinderivate darstellen, die über Etherbindungen (nicht wie sonst üblich über Esterbindungen) an verzweigte Fettsäuren mit 20 bis 40 Kohlenstoffatomen gebunden sind. Die Doppelbindungen in diesen Molekülen sind in der Regel konjugiert. Wie bei allen Thermoproteota (früher „Crenarchaeota“ genannt) besteht die Zellmembran aus einer einfachen Lipidschicht, deren Moleküle aus zwei polaren Enden um ein aliphatisches Segment bestehen. Die Stäbchenzellen sind bis zu 100 μm lang bei einem Durchmesser bis zu ca. 4 µm und vermehren sich durch Knospung an den Zellenden.[3]
Wie andere hyperthermophile Organismen ist Thermoproteus ein lebendes Beispiel für einige der ältesten Organismen der Erde, die basal im Stammbaum der Archaeen stehen.
Thermoproteus-Vertreter wurden in sauer-heißen Quellen in Island, Italien, Nordamerika, Neuseeland, auf den Azoren und in Indonesien gefunden. Ihre optimale Entwicklungstemperatur liegt bei ca. 85 °C.[3]
Genomstruktur
Die Genom-Sequenzierung von Thermoproteus-Vertretern hat viel über die Stoffwechselvorgänge dieser Organismen verraten. Beispielsweise ist das Genom von der Typusart Thermoproteus tenax vollständig sequenziert worden. Die Gesamtlänge des Genoms des Referenzstamms Kra 1 beträgt 1,84 Mbp (Megabasenpaare), die Doppelstrang-DNA ist zirkulär. Die Gene sind in Operons (gemeinsam transkribierten Clustern) angeordnet.[4]
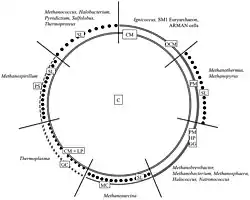
Systematik und Phylogenie
Die gegenwärtig akzeptierte Taxonomie basiert auf der List of Prokaryotic names with Standing in Nomenclature (LPSN),[2] sowie dem National Center for Biotechnology Information (NCBI),[5] mit Ergänzungen gemäß der Genome Taxonomy Database (GTDB)[6] – Stand 19. Juli 2025:
Gattung Thermoproteus Zillig & Stetter 1982(L,N,G)[3]
- Spezies Thermoproteus tenax Zillig & Stetter 1982(L,B,N,G)[3] (Typusart)
- Stamm Kra 1(L,B,N,G) alias ATCC 35583; DSM 2078; JCM 9277; NBRC 100435(L,B,N)
– Fundort: Sauere Thermalquelle/Schlammloch in einem solfatarischen Feld, Krafla, Island(B)
- Stamm Kra 1(L,B,N,G) alias ATCC 35583; DSM 2078; JCM 9277; NBRC 100435(L,B,N)
- Spezies Thermoproteus thermophilus Yim et al. 2015(L,B,N) [inkl. Thermoproteus sp. CBA1502(N)]
- Stamm ATCC BAA-2416; CBA1502; JCM 17229(L,B,N)
– Fundort: Heißes Solfatarisches Sediment, Vulkan Mayon, Philippinen(B,N)
- Stamm ATCC BAA-2416; CBA1502; JCM 17229(L,B,N)
- Spezies „Thermoproteus uzoniensis“ Bonch-Osmolovskaya et al. 1990(L,B,N,G)
- Stamm DSM 5263; Z-605(L,B,N)
– Fundort: Thermalquelle/Solfatar (Boden), Uson Caldera, Kamtschatka, Russland(N) - Stamm 768-20(N,G)
– Fundort: Thermisches Solfatarenfeld in der Nähe des Vulkans Mutnowski, Kamtschatka, Russland(N) - Stamm MAG35(N,G)
– Fundort: Arkashin Schurf[7], Uson Caldera, Kamtschatka, Russland(N)
- Stamm DSM 5263; Z-605(L,B,N)
- Spezies Thermoproteus sp000960885(G) [Thermoproteus sp. AZ2](N)
- Stamm AZ2(N) alias JZWT02000000(N,G)
– Fundort: Los Azufres National Park, Mexiko(N)
- Stamm AZ2(N) alias JZWT02000000(N,G)
- Spezies Thermoproteus sp002077075(G) [Thermoproteus sp. CP80(N), Thermoproteus sp. CIS_19(N), Thermoproteus sp. JCHS_4(N)]
- Stamm CP80(N,G)
– Fundort: Cinder Pool, Thermalquelle im Yellowstone-Nationalpark, Wyoming, USA(N)[8] - Stamm CIS_19(N) alias LOCA01000000(N,G)
– Fundort: Cistern Spring, Thermalquelle im Yellowstone-Nationalpark, USA - Stamm JCHS_4(N) alias LOBZ01000000(N,G)
– Fundort: Joseph's Coat Hot Spring,[9] Thermalquelle im Yellowstone-Nationalpark, Wyoming, USA - Stämme EvPrim.Bin4; RSEP8; RSNP7; RSNS9; 2016_B01_C_7; RBS.bin.1; RBP.bin.3(G)
- Stamm CP80(N,G)
- Spezies Thermoproteus sp002497345(G) [Thermoproteus sp. UBA157(N)]
- Stamm UBA157(N,G)
– Fundort: Shi-Huang-Ping (alias Szehuangtzeping, SHP) Thermalquelle, Datun-Vulkangruppe (大屯火山群, Tatun Volcanic Group, TVG), Nord-Taiwan(N)
- Stamm UBA157(N,G)
- Spezies Thermoproteus sp038893495(G)
- Stamm ZMQL_201912_bins_1(G)– Fundort: ZMQL-Pool, Tengchong, Yunnan, China(N)
- Stämme DRTY-9_202101_bins_3; DRTY-2_202007_bins_8; DRTY-6_202007_bins_35; DRTY-10_202101_bins_3; DRTY-9_201912_bins_3; DRTY-10_201912_bins_11; DRTY-3_202007_bins_3(G)
– Fundort: DRTY-Quellen 2, 6, 9, und 10, Tengchong, Yunnan, China(N)
- Spezies Thermoproteus sp041287225(G) [Thermoproteus sp. isolate KC_019(N)]
- Stamm KC_019(N,G)
– Fundort: Candradimuka Hot Springs,[10] Banjarnegara, Zentral-Java, Indonesien
- Stamm KC_019(N,G)
- Spezies Thermoproteus sp. GDH-1(N)
- Stamm GDH-1(N)[11][12]
– Fundort: terrestrische heiße Quelle auf der Insel Kodakara (Kodakara-jima),[13] Tokara-Inseln, Japan[12]
- Stamm GDH-1(N)[11][12]
- Spezies Thermoproteus sp. IC-031(N)
- Spezies Thermoproteus sp. IC-033(N) [Thermoproteus sp. JCM 14453(B)]
- Stamm IC-033(B,N) alias JCM 14453(B)
– Fundort: Solfatariischer Boden vom Mt. Chausu-dake,[17] Tochigi, Japan(B)
- Stamm IC-033(B,N) alias JCM 14453(B)
- Spezies Thermoproteus sp. IC-061(N) [Thermoproteus sp. JCM 14454(B)]
- Stamm IC-061(B,N) alias JCM 14454, NBRC 100794(B)
– Fundort: Heißes Quellwasser vom Sohunzan-Onsen, Hakone, Kanagawa, Japan(B)
- Stamm IC-061(B,N) alias JCM 14454, NBRC 100794(B)
- Spezies Thermoproteus sp. IC-062(N) [Thermoproteus sp. JCM 14455(B)]
- Stamm IC-062(N,B) alias JCM 14455, NBRC 100795(B)
– Fundort: Heißes Quellwasser vom Sohunzan-Onsen, Hakone, Kanagawa, Japan(B)
- Stamm IC-062(N,B) alias JCM 14455, NBRC 100795(B)
- Spezies Thermoproteus sp. IC-070(N) [Thermoproteus sp. JCM 14456(B)]
- Spezies Thermoproteus sp. JCM 14457(B)
Die Spezies
mit Stamm V24Sta wurde in die Gattung Pyrobaculum verschoben mit der neuen Bezeichnung
- (B) – BacDive – the Bacterial Diversity Metadatabase (DSMZ)[19]
- (G) – Genome Taxonomy Database[6]
- (L) – List of Prokaryotic names with Standing in Nomenclature (DSMZ)[2]
- (N) – Taxonomie des National Center for Biotechnology Information[5]
| 16S-rRNA-basiert: LTP_06_2022[20] | Basierend auf 53 Markerproteinen: GTDB 09-RS220[21] | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
|
|
Zellaufbau und Stoffwechsel
Der Stoffwechsel von Thermoproteus und anderen hyperthermophilen Organismen wurde inzwischen in großem Umfang erforscht. Thermoproteus lebt autotroph durch Schwefelreduktion, wächst aber durch Schwefelatmung in der Kultur viel schneller. Bei T. tenax ermöglichen mehrere Stoffwechselwege der Zelle, je nach ihrem Energiebedarf (abh. von den Entwicklungs- oder Wachstumsstadien) eine bestimmte Art des Stoffwechsels zu wählen. Wie alle Archaeen besitzt Thermoproteus charakteristische Membranlipide, bei denen es sich um etherverknüpfte Glycerinderivate mit Verzweigungen und 20 oder 40 Kohlenstoffatomen handelt. Die Doppelbindungen der ungesättigten Lipide ist im Allgemeinen konjugiert (im Gegensatz zu den unkonjugierten bei Bakterien und Eukaryoten). Bei Thermoproteus, wie auch bei allen anderen Thermoproteota („Crenarchaeota“, z. B. Thermosphaera) überwiegen die 40-Kohlenstoff-Lipide, die die gesamte Membran bedecken. Dies führt dazu, dass die Membran aus Monoschichten mit polaren Gruppen an jedem Ende besteht. Die Zellen sind stäbchenförmig mit einem Durchmesser von bis zu 4 Mikrometern und einer Länge von bis zu 100 Mikrometern und vermehren sich, indem sie am Ende der Zelle Verzweigungen bilden, die zu eigenen neuen Zellen heranwachsen. Sie sind durch Geißeln (‚Archaellen‘) beweglich.
Ökologie
Mitglieder der Gattung Thermoproteus kommen in sauren heißen Quellen und Solfataren vor; sie wurden in diesen Lebensräumen u. a. in Island, Italien, Nordamerika, Neuseeland, den Azoren und Indonesien isoliert. Ihre optimale Wachstumstemperatur beträgt 85 °C.[5][22]
Viren

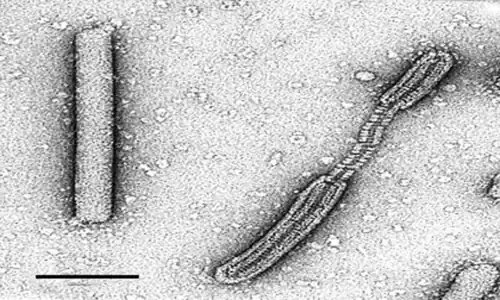
Ein Beispiel für ein Virus, das Thermoproteus-Archaeen infiziert, ist das Thermoproteus tenax virus 1 (TTV1, Spezies Betatristromavirus kraflaense) aus der Familie Tristromaviridae.[23]
Auch in der Familie Globuloviridae gibt es Vertreter, die Thermoproteus-Arten infizieren, etwa Thermoproteus tenax spherical virus 1 (TTSV1, Spezies Alphaglobulovirus cinderense) und Thermoproteus spherical piliferous virus 1 (TSPV1, Spezies Alphaglobulovirus sileriense). Globuloviren infizieren allgemein Mitglieder der in extremen geothermischen Umgebungen gedeihenden hyperthermophilen Archaeen der Gattungen Pyrobaculum und Thermoproteus. Die Infektion führt nicht zur Lyse der Wirtszellen, andererseits integriert sich das virale Genom aber auch nicht in das Wirtsgenom.[24][25]
Weiterführende Literatur
- Andrés Santos, Pablo Bruna, Jaime Martinez-Urtaza, Francisco Solis, Bernardita Valenzuela, Pedro Zamorano, Leticia Barrientos: Two Archaeal Metagenome-Assembled Genomes from El Tatio Provide New Insights into the Crenarchaeota Phylum. In: MDPI: Genes, Band 12, Nr. 3, 9. März 2021, S. 391; doi:10.3390/genes12030391 (englisch).
Quellen
Benutzte Literatur
- Judicial Commission of the International Committee on Systematics of Prokaryotes: The nomenclatural types of the orders Acholeplasmatales, Halanaerobiales, Halobacteriales, Methanobacteriales, Methanococcales, Methanomicrobiales, Planctomycetales, Prochlorales, Sulfolobales, Thermococcales, Thermoproteales and Verrucomicrobiales are the genera Acholeplasma, Halanaerobium, Halobacterium, Methanobacterium, Methanococcus, Methanomicrobium, Planctomyces, Prochloron, Sulfolobus, Thermococcus, Thermoproteus and Verrucomicrobium, respectively. Opinion 79. In: International Journal of Systematic and Evolutionary Microbiology. 55. Jahrgang, Nr. 1, 1. Januar 2005, S. 517–518, doi:10.1099/ijs.0.63548-0, PMID 15653928 (englisch).
- Siegfried Burggraf, Harald Huber, Karl O. Stetter: Reclassification of the crenarchael orders and families in accordance with 16S rRNA sequence data. In: International Journal of Systematic and Evolutionary Microbiology. 47. Jahrgang, Nr. 3, 1. Juli 1997, S. 657–660, doi:10.1099/00207713-47-3-657, PMID 9226896 (englisch).
Einzelnachweise
- ↑ Carl R. Woese, Gary J. Olsen: Archaebacterial phylogeny: perspectives on the urkingdoms. In: Systematic and Applied Microbiology. 7. Jahrgang, Nr. 2–3, Mai 1986, S. 161–177, doi:10.1016/s0723-2020(86)80001-7, PMID 11542063 (englisch).
- ↑ a b c J.P. Euzéby: Thermoproteus. List of Prokaryotic names with Standing in Nomenclature (LPSN), abgerufen am 10. Juni 2023.
- ↑ a b c d Wolfram Zillig, Karl O. Stetter, W. Schäfer, Davorin Janekovic, Simon Wunderl, Ingelore Holz, Peter Palm: Thermoproteales: a novel type of extremely thermoacidophilic anaerobic archaebacteria isolated from Icelandic solfataras. In: Zentralblatt für Bakteriologie Mikrobiologie und Hygiene: I. Abt. Originale C: Allgemeine, angewandte und ökologische Mikrobiologie. 2. Jahrgang, Oktober 1981, S. 205–227, doi:10.1016/S0721-9571(81)80001-4 (englisch).
- ↑ Bettina Siebers, Melanie Zaparty, Guenter Raddatz, Britta Tjaden, Sonja-Verena Albers, Steve D. Bell, Fabian Blombach, Arnulf Kletzin, Nikos Kyrpides, Christa Lanz, André Plagens, Markus Rampp, Andrea Rosinus, Mathias von Jan, Kira S. Makarova, Hans-Peter Klenk, Stephan C. Schuster, Reinhard Hensel: The Complete Genome Sequence of Thermoproteus tenax: A Physiologically Versatile Member of the Crenarchaeota. In: PLOS ONE. Band 6, Nr. 10, 7. Oktober 2011, S. e24222; doi:10.1371/journal.pone.0024222, bibcode:2011PLoSO...624222S, PMC 3189178 (freier Volltext), PMID 22003381 (englisch).
- ↑ a b c NCBI Taxonomy Browser: Thermoproteus, Details: Thermoproteus Zillig and Stetter 1982. Rank: genus. Graphisch: Thermoproteus. Auf: Lifemap.
- ↑ a b GTDB: Thermoproteus.
- ↑ Elizabeth A. Burgess, Jason M. Unrine, Gary L. Mills, Christopher S. Romanek, Juergen Wiegel: Comparative Geochemical and Microbiological Characterization of Two Thermal Pools in the Uzon Caldera, Kamchatka, Russia. In: Environmental Microbiology: Microbial Ecology, Band 63, Nr. 3, April 2012, S. 471–489; doi:10.1007/s00248-011-9979-4, PMID 22124570, ResearchGate:51833597, Epub 29. November 2011 (englisch). Siehe insbes. Fig. 1 Arkashin Schurf & Zavarzin Spring.
- ↑ Matthew R. Urschel, Trinity L. Hamilton, Eric E. Roden, Eric S. Boyd: Substrate preference, uptake kinetics and bioenergetics in a facultatively autotrophic, thermoacidophilic crenarchaeote. In: FEMS Microbiology Ecology, Band 92, Nr. 5, Mai 2016, S. fiw069; doi:10.1093/femsec/fiw069, PMID 27037359, Epub 31. März 2016 (englisch).
- ↑ Josephs Coat Springs, USA. Auf. GeoNames.
- ↑ Kawah Candradimuka, Indonesian. Auf: GeoNames.
- ↑ Glucose 1-dehydrogenase. Species: Thermoproteus sp. GDH-1. Auf: InterPro (ebi.ac.uk).
- ↑ a b Hiroshi Aiba, Yoshiaki Nishiya, Masayuki Azuma, Yuusuke Yokooji, Haruyuki Atomi, Tadayuki Imanaka: Characterization of a thermostable glucose dehydrogenase with strict substrate specificity from a hyperthermophilic archaeon Thermoproteus sp. GDH-1. In: Bioscience, Biotechnology, and Biochemistry, Band 79, Ne. 7, 3. Juli 2015, S. 1094–1102; doi:10.1080/09168451.2015.1018120, PMID 25746627, Epub 9. März 2015 (englisch).
- ↑ Ko-takara-jima. Auf: GeoNames.
- ↑ a b JCM:14452. Japan Collection of Microorganisms (jcm.riken.jp).
- ↑ Tochigi’s Okushiobara Onsen. 29. April 2018 (nippon.com).
- ↑ Okushiobara Kogen, mit nahen Thermalquellen 寺の湯 (Dera no yu) und 中の湯 (Chū no yu). Auf Mapcarta (de).
- ↑ a b Mt. Chausu-dake, Japan. Auf: GeoNames.
- ↑ JCM:14457. Japan Collection of Microorganisms (jcm.riken.jp).
- ↑ Thermoproteus BacDive - the Bacterial Diversity Metadatabase (dsmz.de).
- ↑
‘The All-Species Living Tree’ Project 06_2022
- About. Abgerufen am 10. Mai 2024 (englisch).
- LTP_all tree in newick format. Abgerufen am 10. Mai 2024.
- LTP_06_2022 Release Notes. Abgerufen am 10. Mai 2024 (englisch).
- ↑
Genome Taxonomy Database (GTDB): Release 09-RS220
- About. In: GTDB. Abgerufen am 10. Mai 2024 (englisch).
- ar53_r220.sp_label. In: GTDB. Abgerufen am 10. Mai 2024.
- Taxon History. In: Genome Taxonomy Database. Abgerufen am 10. Mai 2024 (englisch).
- ↑ Paul Messner, Dietmar Pum, Margit Sára, Karl O. Stetter, Uwe B. Sleytr: Ultrastructure of the cell envelope of the archaebacteria Thermoproteus tenax and Thermoproteus neutrophilus. In: Journal of Bacteriology. 166. Jahrgang, Nr. 3, 1. Juni 1986, S. 1046–1054, doi:10.1128/jb.166.3.1046-1054.1986, PMID 3086286, PMC 215230 (freier Volltext) – (englisch).
- ↑ ICTV: Taxon name: Betatristromavirus kraflaense.
- ↑ David Prangishvili, Mart Krupovic: Family: Globuloviridae. The ICTV Report on Virus Classification and Taxon Nomenclature.
- ↑ NCBI Taxonomy Browser: Globuloviridae.