Saccharolobus solfataricus
| Saccharolobus solfataricus | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
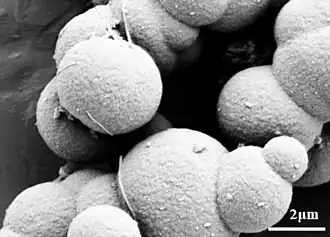
EM-Aufnahme von auf einem Substrat fixierten Zellen von S. solfataricus | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Saccharolobus solfataricus | ||||||||||||
| (Zillig et al. 1980) Sakai & Kurosawa 2018 |
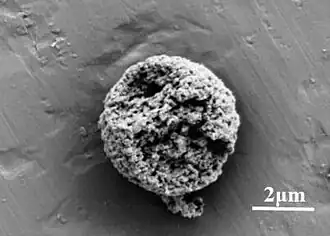
Saccharolobus solfataricus (früher Sulfolobus solfataricus) ist eine Art (Spezies) thermophiler (hitzeliebender) Archaeen aus der Klasse Thermoprotei im Reich Thermoproteati (alias TACK-Superphylum).[1][2][3]
S. solfataricus wurde 1980 von den beiden deutschen Mikrobiologen Karl Stetter und Wolfram Zillig erstmals auf dem Vulkan Solfatara (Pisciarelli, Pozzuoli, Region Kampanien, Italien) entdeckt und isoliert.[4] Diese Organismen sind jedoch nicht auf Vulkane beschränkt, sondern kommen in Umgebungen mit hoher Temperatur überall auf der Welt vor, insbesondere an heißen Quellen.
Die Art galt ursprünglich als Vertreter der Gattung Sulfolobus, wurde aber 2018 zusammen mit der Erstbeschreibung von Saccharolobus caldissimus aus dieser Gattung in die neue Gattung Saccharolobus überführt.[5]
S. solfataricus gedeiht am besten bei Temperaturen um 80 °C, einem pH-Wert zwischen 2 und 4 und mit genügend Schwefel, den diese Organismen zur Energiegewinnung verstoffwechseln können. Diese Bedingungen qualifizieren sie als extremophil, und wegen ihrer Vorliebe für hohe Temperaturen und niedrige pH-Werte werden sie speziell als thermoacidophil (hitze- und säureliebend) bezeichnet. Aufgrund ihres Stoffwechselsystems ist die Spezies außerdem aerob und heterotroph.[6] Als autotropher Organismus gewinnt sie Energie, indem sie auf Schwefel oder auch auf einer Vielzahl organischer Verbindungen wächst.[7] Sie hat in der Regel eine kugelförmige (kokkoide) Zellform und bildet häufig Lappen.
S. solfataricus ist der am meisten untersuchte Organismus innerhalb des Phylums Thermoproteota (früher „Crenarchaeota“, Stand 2006).[8] Die Spezies wurde und wird auf ihre Methoden der DNA-Replikation, des Lebenszyklus, der chromosomalen Integration, der Transkription, der RNA-Verarbeitung und der Translation untersucht. Die Daten deuten darauf hin, dass der Organismus einen hohen Prozentsatz an archaeenspezifischen Genen aufweist, was die Unterschiede zwischen den drei großen Gruppen von Mikroben zeigt: Archaeen, Bakterien und Mikroeukaryoten (Eukaryoten).
Genom

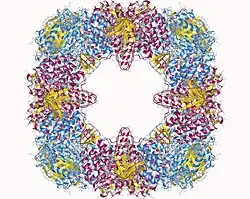

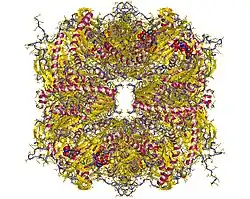
S. solfataricus ist aus molekularer, genetischer und biochemischer Sicht einer der am besten untersuchten Mikroorganismen, der in extremen Umgebungen gedeihen kann. Trotz seiner Extremophilie lässt er sich im Labor leicht vermehren, wobei die Zellen genetisches Material durch die Prozesse Transformation, Transduktion und Konjugation austauschen können. Da Proteine normalerweise bei hohen Temperaturen zerfallen (denaturieren), ist die Thermostabilität ihrer Proteine ein Hauptgrund für die Sequenzierung thermophiler Mikroorganismen wie S. solfataricus. Die von solchen Proteinen in der Biotechnologie katalysierten Reaktionen (wie beispielsweise PCR) können dann bei höheren Temperaturen und damit schneller ablaufen.
Die vollständige Sequenzierung des Genoms von S. solfataricus wurde 2001 abgeschlossen.[12] Auf einem einzigen Chromosom befinden sich 2.992.245 Basenpaare (bp), die für 2.977 Proteine und zahlreiche RNAs kodieren. Ein Drittel der kodierten Proteine von S. solfataricus hatte keine Homologe in Genomen anderer Organismen. Von den übrigen kodierten Proteinen sind 40 % spezifisch für Archaeen, 12 % kommen auch in Bakterien und 2,3 % auch in Eukaryoten vor.[13] 33 % der Proteine werden ausschließlich in Sulfolobus/Saccharolobus/Sulfurisphaera kodiert (Stand 2002). Eine große Anzahl offener Leserahmen (ORFs) ist denen der Gattung Thermoplasma aus dem Archaeen-Reich Methanobacteriati („Euryarchaeida“) sehr ähnlich.[6]
Auch „kleine nukleoläre RNAs“ (englisch small nucleolar ribonucleic acids, snoRNAs), wie sie in Eukaryoten vorkommen, wurden sowohl in Saccharolobus solfataricus als auch in Sulfolobus acidocaldarius entdeckt. Sie sind bereits für ihre Rolle bei Veränderungen an der RNA nach der Transkription (posttranskriptionellen Veränderungen) wie der Entfernung von Introns aus der ribosomalen RNA (rRNA) bei Eukaryoten bekannt. Das Genom ist durch kurze Tandemwiederholungen (Short Tandem Repeats), Insertionen und andere repetitiven Elemente gekennzeichnet. Es weist eine große Vielfalt von ca. 200 verschiedenen Insertionssequenz-Elementen (insertion sequence elements) auf.[14]
Thermophile Reverse Gyrase
Die Stabilisierung der DNA-Struktur gegen Denaturierung ist in hyperthermophilen und thermophilen Archaeen und Bakterien auf ein besonderes hitzebeständiges (thermostabiles) Enzym, die Reverse Gyrase, zurückzuführen. In S. solfataricus gibt es zwei Gene, die jeweils für eine Reverse Gyrase kodieren.[15] Es handelt sich dabei um eine atypische DNA-Topoisomerase, deren Hauptaktivität in der Herstellung von positivem Supercoiling[A. 1] in einer geschlossenen zirkulären DNA besteht. Positive Supercoils sind wichtig, um Fehlbildungen und -faltungen (wie „offener Komplexe“, open complexes) zu verhindern. Reverse Gyrasen bestehen aus zwei Domänen: die erste ist eine Helikase und die zweite die Topoisomerase I. Eine mögliche Rolle der Reversen Gyrase könnte auch darin bestehen, dass sie mit den positiven Supercoils chromatinartige Strukturen aufbaut.[16] 1997 entdeckten Wissenschaftler ein weiteres wichtiges Merkmal der nahe verwandten Spezies Saccharolobus shibatae (früher ebenfalls der Gattung Sulfolobus zugeordnet): eine Topoisomerase vom Typ II, genannt TopoVI, deren Untereinheit A homolog ist zum meiotischen Rekombinationsfaktor Spo11, der als Initiator meiotischer Doppelstrangbrüche bei allen Eukaryonten eine wichtige Rolle spielt, indem er die meiotische Rekombination einleitet.[17][18][19] S. solfataricus selbst besitzt aus drei Topoisomerasen vom Typ I: TopA und zwei Reverse Gyrasen, TopR1 und TopR2; sowie eine Topoisomerase vom Typ II, TopoVI.[20]
DNA-bindende Proteine
DNA-bindende Proteine sind Proteine, die an die DNA binden. Bei Organismen des Phylums Thermoproteota gibt es drei Proteine, die an die Nebenrille von DNA-bindenden Histonen binden, und die nach dem Übersetzungsprozess verändert werden: Alba, Cren7 und Sso7d. Diese Histone sind klein und wurden in mehreren Stämmen von Sulfolobus/Saccharolobus/Sulfurisphaera gefunden, nicht aber in anderen Genomen. Chromatinproteine wie die Histone machen bei diesen Organismen 1–5 % des gesamten Genoms aus; sie können sowohl strukturelle als auch regulatorische Funktionen haben und ähneln aufgrund ihres Einflusses auf Genom, Expression, Stabilität und epigenetische Prozesse den menschlichen HMG-Box-Proteinen (englisch high mobility group box).[21][22] Sie können auch bei Arten ohne Histone wie diese acetyliert und methyliert werden.[23][24][25][26] Stämme von S. solfataricus weisen verschiedene besondere DNA-bindende Proteine auf, wie die der Proteinfamilie Sso7d. Diese stabilisieren die DNA-Struktur, verhindern die Denaturierung bei hohen Temperaturen und fördern so den Erhalt der primär durch die Wasserstoffbrückenbindungen vermittelten DNA-Doppelhelix-Struktur auch oberhalb des „Schmelzpunkts“ des DNA-Doppelstrangs, bei dem normalerweise die Auflösung der Doppelhelix beginnt.[27] Der Hauptbestandteil des Chromatins dieser Archaeen ist das Alba genannte Protein der Sac10b-Familie,[28][29][30] mit ihren kleinen, basischen und dimeren nukleinsäurebindenden Proteinen. Dabei ist Alba in den meisten sequenzierten Archaeen-Genomen konserviert.[30][31] Der Acetylierungszustand[A. 2] von Alba beeinflusst den Zugang zum DNA-Promotor und damit die Transkription,[A. 3] während der Methylierungszustand eines anderen Chromatinproteins, Sso7D, durch die Temperatur verändert wird, bei der die Kultur gehalten wird.[32][33]
Bereits die Arbeiten der Gruppe von Wolfram Zillig lieferten frühe Beweise für die eukaryotischen Merkmale der Transkription in diesen Archaeen, was S. solfataricus seitdem zu einem idealen Modellsystem für Transkriptionsstudien gemacht hat. Neuere Studien an Arten von Saccharolobus, Sulfolobus und anderen Archaeen konzentrieren sich hauptsächlich auf die Zusammensetzung, Funktion und Regulierung der Transkriptionsmaschinerie, sowie auf grundlegende sowohl bei Eukaryonten als auch bei Archaeen konservierten Aspekten dieses Prozesses.[34]
DNA-Transfer
Die Exposition von S. solfataricus gegenüber DNA-schädigenden Agenzien wie ultravioletter (UV) Bestrahlung, Bleomycin oder Mitomycin C bewirkt, dass sich die Einzelzellen zusammenballen, induziert also zelluläre Aggregation.[35] Andere physikalische Stressfaktoren wie Änderungen des pH-Werts oder der Temperatur induzieren dagegen keine Aggregation – dies deutet darauf hin, dass die Induktion der Aggregation im Zusammenhang mit DNA-Schädigung steht oder durch sie verursacht wird. Małgorzata Ajon et al. zeigten 2011, dass die UV-induzierte zelluläre Aggregation den Austausch chromosomaler Marker mit hoher Frequenz vermittelt.[36] Die Rekombinationsraten übertrafen die von „nicht induzierten“[A. 4] Kulturen um bis zu drei Größenordnungen. Sabrina Fröls et al. sowie Małgorzata Ajon et al. stellten die Hypothese auf, dass der UV-induzierte DNA-Transferprozess und die anschließende Reparatur durch homologe Rekombination einen wichtigen Mechanismus zur Aufrechterhaltung der Chromosomenintegrität darstellen.[35][37][36] Diese Reaktion könnte eine primitive Form der sexuellen Interaktion sein, ähnlich wie die besser untersuchte bakterielle Transformation, die ebenfalls mit einem DNA-Transfer zwischen Zellen verbunden ist und zu einer Reparatur von DNA-Schäden durch homologe Rekombinativen Reparatur führt.[38]
Stoffwechsel
S. solfataricus kann in Gegenwart von Sauerstoff per Chemoorganotrophie wachsen und dabei eine Vielzahl organischer Verbindungen wie Zucker, Alkohole, Aminosäuren und aromatische Verbindungen wie Phenol aufnimmt.[39] Die Spezies nutzt einen modifizierten Entner-Doudroff-Stoffwechselweg für die Glukose-oxidation, die dabei entstehenden Pyruvatmoleküle können in einem Citratzyklus (TCA-Zyklus) vollständig mineralisiert werden.[39]
Molekularer Sauerstoff (O2) ist der einzige bekannte Elektronenakzeptor am Ende der Elektronentransportkette.[40] Neben organischen Molekülen kann diese Archaeenspezies auch Schwefelwasserstoff (H2S)[13] und elementaren Schwefel als Elektronendonatoren und die Kohlenstoffdioxid-Assimilation nutzen, möglicherweise mit Hilfe des HP/HB-Zyklus (3-Hydroxypropionat-/4-Hydroxybutyrat-Zyklus),[41][39] wodurch sie auch in der Lage ist, chemoautotroph zu leben. In neueren Studien wurde zudem eine Fähigkeit festgestellt, durch Oxidation von molekularem Wasserstoff (H2) zu wachsen, wenn auch langsam.[5]
Ferredoxin
Es wird vermutet, dass Ferredoxin bei S. solfataricus der wichtigste metabolische Elektronenträger ist. Dies würde im Gegensatz stehen zu den meisten Arten innerhalb der Domänen der Bakterien und Eukaryoten, die im Allgemeinen auf Nicotinamid-Adenin-Dinukleotid-Wasserstoff (NADH) als Hauptelektronenüberträger angewiesen sind. S. solfataricus weist darüber hinaus starke eukaryotische Merkmale auf, gepaart mit vielen ganz archaeenspezifischen Fähigkeiten. Diese Befunde sind Konsequenz ihrer vielfältigen Methoden von DNA-Mechanismen, Zellzyklen und Transitionsmechanismen. Insgesamt zeigen die Studien exemplarisch die Unterschiede zwischen Thermoproteota wie S. solfataricus und Methanobacteriati („Euryarchaeida“).[13][42]
Ökologie
Habitat

S. solfataricus ist ein extrem thermophiler Vertreter der Archaeen. Diese Spezies findet ähnlich wie andere Mitglieder ihrer eigenen Gattung sowie der verwandten Gattung Sulfolobus optimale Wachstumsbedingungen in Gebieten mit starker vulkanischer Aktivität, hohen Temperaturen und einem sehr sauren pH-Wert,[43] wie sie typisch sind Geysire oder Thermalquellen. Zu den Ländern, in denen diese Mikroorganismen gefunden wurden, gehören für vulkanische Phänomene „berüchtigte“ Gebiete in den USA (Yellowstone-Nationalpark),[44] Island und Italien (Phlegräische Felder). Im Jahr 2007 wurden Sulfolobaceae-Gensequenzen aus Neuseeland (Nordinsel: Waiotapu) gefunden.[45] Eine von einem indonesischen Wissenschaftlerteam durchgeführte Studie hat 2009 auch das Vorhandensein Gemeinschaft mit thermoacidophilen Thermoproteota (früher „Crenarchaeota“) in West-Java nachgewiesen und bestätigt, dass hohe Temperaturen, ein niedriger pH-Wert und das Vorhandensein von Schwefel notwendige Bedingungen für das Wachstum dieser Mikroben sind.[46] Eine weitere indonesische Studie bestätigte 2012 das Vorkommen von Sulfolobales bzw. Sulfolobaceae.[47]
Bodenversauerung
S. solfataricus ist gemäß seiner Stoffwechselstrategie in der Lage, Schwefel zu oxidieren. Eines der Produkte dieser Reaktionen ist H+, was eine langsame Versauerung der Umgebung zur Folge hat. Die Versauerung des Bodens nimmt dort zu, wo Schadstoffemissionen aus der Industrie vorhanden sind. Dieser Prozess verringert die Zahl der an der Zersetzung beteiligten heterotrophen Bakterien, die für den Prozess der Wiederverwertung organischer Stoffe von grundlegender Bedeutung sind und verlangsamt so letztlich die Düngung des Bodens mit diesen Zersetzungsprodukten.[48]
Anwendungen in der Biotechnology
An der Spezies S. sulfataricus als mögliche Quelle für thermostabile Enzyme besteht großes Interesse, sowohl von Seiten der Forschung und Diagnostik als auch für Industriezweige wie die Lebensmittel-, Textil-, Reinigungs- sowie die Zellstoff- und Papierindustrie. Darüber hinaus sind solche Enzyme aufgrund ihrer katalytischen Vielfalt, der hohen pH- und Temperaturstabilität, sowie ihrer erhöhten Widerstandsfähigkeit gegenüber organischen Lösungsmitteln und ihrer Resistenz gegenüber Proteolyse sehr gefragt.[49][50] Insbesondere gewinnen gegenwärtig Tetretherlipide, Membranvesikel mit antimikrobiellen Eigenschaften, Trehalose als Speicherstoff und neue β-Galactooligosaccharide (β-GOS) zunehmend an Bedeutung:[51]
β-Galaktosidase
Das thermostabile Enzym β-Galactosidase wurde aus dem extrem thermophilen Stamm S. solfataricus MT-4, isoliert. Dieses Enzym wird in vielen industriellen Prozessen mit laktosehaltigen Flüssigkeiten verwendet, zwecks deren Reinigung und der Charakterisierung ihrer physikochemischen Eigenschaften.[52]
Proteasen
Die Industrie ist sowohl an stabilen Proteasen als auch an vielen verschiedenen Sulfolobus- (bzw. Sulfolobaceae-)
Bereits 1989 wurde eine aktive Aminopeptidase, die mit dem Chaperonin von S. solfataricus MT4 assoziiert ist, beschrieben.[54] Sommaruga et al. verbesserten 2014 die Stabilität und Reaktionsausbeute der gut charakterisierten Carboxypeptidase aus S. solfataricus MT4 durch magnetische Nanopartikel, die das Enzym immobilisierten.[55]
Lipasen und andere Esterasen
Lipasen (Lipide auflösende, d. h. lipolytische Enzyme) sind spezielle fettspaltende Esterasen. Eine neues thermostabile extrazelluläre Lipase, die Serin-Arylesterase,[56] Sie hat eine großen Wirksamkeit bei der Hydrolyse von Organophosphaten und wurde ursprünglich bei S. solfataricus P1 entdeckt.[57]
Chaperonine
Bei Escherichia coli wurde ein einziges Hitzeschockprotein (englisch heat shock protein, HSP, auch warm stun protein), HSP20 von S. solfataricus P2 zur effektiven Verbesserung der Temperaturtoleranz (gegen einen Temperaturschock bei 50,4 °C) eingesetzt.[58]
Da das Chaperonin Ssocpn (920 kDa) von S. solfataricus enthält Adenosintriphosphat (ATP), K+ und Mg2+. Es benötigt für seine volle Aktivität keine weiteren Proteinpartner und unterstützt in vitro die Faltung mit geringer Substratspezifität, um dynamische Proteine aus denaturierten Materialien zurückzugewinnen. Dies ermöglicht die Konstruktion eines Reaktors auf Chaperoninbasis, der aus denaturiertem Material gefaltete und aktive Proteine erzeugt.[59]
Liposomen
Aufgrund ihres Tetraether-Lipidmaterials ist die Membran von extrem thermophilen Archaeen in ihrer Zusammensetzung einzigartig. Diese Archaeen-Lipide sind vielversprechende Ausgangsstoffe für die Erzeugung von Liposomen mit außergewöhnlicher Temperatur- und pH-Stabilität, die zudem eine hohe sowie Dichtigkeit gegen das Austreten von gelösten Stoffen haben. Solche „Archaeosomen“ sind mögliche Instrumente für die Verabreichung von Medikamenten, Impfstoffen und Genen.[60]
Stämme
Referenzstamm:
- P1 alias ATCC 35091, DSM 1616, IFO 15331, JCM 8930, NBRC 15331 – Fundort: Campi flegrei, Pisciarelli bei Pozzuoli/Agnano bei Neapel, Italien[1][57][57][2][61]
Weitere Stämme:
- P1-G alias DSM 5354 – Fundort: Campi flegrei, Pisciarelli bei Agnano[61]
- P2[3][13][9][58][12][40][2][61] alias DSM 1617, JCM 11322, ATCC 35092[61] – Fundort: Campi flegrei, Pisciarelli bei Agnano[61]
- MT-4 alias MT4, DSM 5833 – Fundort: Pozzuoli7Agnano bei Neapel, Italien[55][3][61]
- S441 – Troll Bridge, Lassen-Volcanic-Nationalpark, Nordkalifornien, USA[3][2]
- POZ149 – Fundort: Pozzuoli, Italien[3][2]
Viren

Als Beispiel für Viren, die S. solfataricus infizieren, sei hier nur spindelförmige SSVRH aus der Familie Fuselloviridae erwähnt:[62][64]
- Spezies: Alphafusellovirus yellowstonense
- Virus: Sulfolobus spindle-shaped virus 8 (SSV8) mit Sulfolobus virus Ragged Hills (SSVRH)
Für eine ausführlichere Darstellung siehe Sulfolobaceae §Viren.
Anmerkungen
- ↑ Positives Supercoiling ist eine Form des DNA-Supercoilings (DNA-Superverdrillung), bei der die gesamte Doppelhelix zusätzlich in die gleiche Richtung verdrillt wird, in der sie bereits selbst vorliegt, d. h. rechtsgängig.
- ↑ Acetylierung senkt die Bindungsaffinität
- ↑ nachgewiesen in vitro
- ↑ durch geeigneten stress zur Aggregation veranlassten
- ↑ β-Galactooligosaccharide sind die wichtigsten Oligosaccharide in der menschlichen Muttermilch und können in der Lebensmittelindustrie als Nahrungsergänzungsmittel verwendet werden.
Weblinks
- BacDive: Saccharolobus solfataricus P1, a thermophilic archaeon that was isolated from volcanic hot spring. Referenzstamm von S. solfataricus. Bacterial Diversity Metadatabase (bacdive.dsmz.de).
- Chu-yu Fu, Kang Wang, Lu Gan, Jason Lanman, Reza Khayat, Mark J. Young, Grant J. Jensen: Sulfolobus solfataricus Strain 2-1-12. Caltech Data, 2010 (data.caltech.edu).
Weiterführende Literatur
- Benham Zolghadr, Andreas Klingl, Reinard Rachel, Arnold J. M. Driessen, Sonja-Verena Albers: The bindosome is a structural component of the Sulfolobus solfataricus cell envelope. In: Extremophiles, Band 15, März 2011, S. 235–244; doi:10.1007/s00792-010-0353-0, Epub 14. Januar 2011 (englisch).
Einzelnachweise
- ↑ a b LPSN: Species Saccharolobus solfataricus (Zillig et al. 1980) Sakai and Kurosawa 2018.
- ↑ a b c d e f g h i j k l m n o p GTDB: Saccharolobus solfataricus.
- ↑ a b c d e f g h i j k l m n o NCBI Taxonomy Browser: Saccharolobus solfataricus. Details: Saccharolobus solfataricus (Zillig et al. 1980) Sakai and Kurosawa 2018; basionym: Sulfolobus solfataricus Zillig et al. 1980. Rank: species. Grapgisch: Saccharolobus solfataricus. Auf: Lifemap.
- ↑ Frédéric Pecorari et al. (CNRS): Where was Sulfolobus solfataricus first found? In: www.intercept.cnrs.fr. 15. Januar 2019 (englisch).
- ↑ a b Hiroyuki D. Sakai, Norio Kurosawa: Saccharolobus caldissimus gen. nov., sp. nov., a facultatively anaerobic iron-reducing hyperthermophilic archaeon isolated from an acidic terrestrial hot spring, and reclassification of Sulfolobus solfataricus as Saccharolobus solfataricus comb. nov. and Sulfolobus shibatae as Saccharolobus shibatae comb. nov. In: International Journal of Systematic and Evolutionary Microbiology, Band 68., Nr. 4, April 2018, S. 1271–1278; doi:10.1099/ijsem.0.002665, PMID 29485400 (englisch).
- ↑ a b Maria Ciaramella, Francesca M. Pisani, Mosé Rossi: Molecular biology of extremophiles: recent progress on the hyperthermophilic archaeon Sulfolobus. In: Antonie van Leeuwenhoek, Band 81, Nr. 1–4, August 2002, S. 85–97; doi:10.1023/A:1020577510469, PMID 12448708 (englisch).
- ↑ Thomas D. Brock, Katherine M. Brock, Robert T. Belly & Richard L. Weiss: Sulfolobus: a new genus of sulfur-oxidizing bacteria living at low pH and high temperature. In: Archiv für Mikrobiologie, Band 84, Nr. 1, März 1972, S. 54–68; doi:10.1007/bf00408082, PMID 4559703, bibcode:1972ArMic..84...54B (englisch).
- ↑ a b Santina Prato, Raffaele Cannio, Hans-Peter Klenk, Patrizia Contursi, Mosè Rossi, Simonetta Bartolucci: pIT3, a cryptic plasmid isolated from the hyperthermophilic crenarchaeon Sulfolobus solfataricus IT3. In: Plasmid, Band 56, Nr. 1, Juli 2006, S. 35–45; doi:10.1016/j.plasmid.2006.02.002 (englisch).
- ↑ a b c 4XCG. Crystal structure of a hexadecameric TF55 complex from S. solfataricus. Classification: CHAPERONE. Organism(s): Saccharolobus solfataricus P2 (rcsb.org).
- ↑ 2C37 RNase. Classification: Hydrolase. Organism(s): Saccharolobus solfataricus (rcsb.org).
- ↑ N-Octyl-beta-D-Glucopyranosid (thermofisher.com), n-Octylglucosid (Merck/Roche, www.sigmaaldrich.com).
- ↑ a b
Robert L. Charlebois, Terry Gaasterland, Mark A. Ragan, W. Ford Doolittle, Christoph W. Sensen: The Sulfolobus solfataricus P2 genome project. In: FEBS Letters, Band 389, Nr. 1, 24. Juni 1996, S. 88–91; doi:10.1016/0014-5793(96)00525-X, Epub 8. März 1999 (englisch). Dazu:
- Erratum (Korrektur). In: FEBS Letters, Band 398, Nr. 2–3, 2. Dezember 1996, S. 343; doi:10.1016/S0014-5793(97)81281-1.
- ↑ a b c d Qunxin She, Rama K. Singh, Fabrice Confalonieri, Yvan Zivanovic, Ghislaine Allard, Mariana J. Awayez, Christina C.-Y. Chan-Weiher, Ib Groth Clausen, Bruce A. Curtis, Anick De Moors, Gael Erauso, Cynthia Fletcher, Paul M. K. Gordon, Ineke Heikamp-de Jong, Alex C. Jeffries, Catherine J. Kozera, Nadine Medina, Xu Peng, Hoa Phan Thi-Ngoc, Peter Redder, Margaret E. Schenk, Cynthia Theriault, Niels Tolstrup, Robert L. Charlebois, W. Ford Doolittle, Michel Duguet, Terry Gaasterland, Roger A. Garrett, Mark A. Ragan, Christoph W. Sensen, John Van der Oost: The complete genome of the crenarchaeon Sulfolobus solfataricus P2. In: PNAS, Band 98, Nr. 14, Juli 2001, S. 7835–7840; doi:10.1073/pnas.141222098, PMC 35428 (freier Volltext), PMID 11427726, bibcode:2001PNAS...98.7835S (englisch).
- ↑ Arina D. Omer, Todd M. Lowe, Anthony G. Russell, Holger Ebhardt, Sean R. Eddy, Patrick P. Dennis: Homologs of small nucleolar RNAs in Archaea. In: Science. 288. Jahrgang, Nr. 5465, April 2000, S. 517–22, doi:10.1126/science.288.5465.517, PMID 10775111, bibcode:2000Sci...288..517O (englisch).
- ↑ Mohea Couturier, Anna H. Bizard, Florence Garnier, Marc Nadal: Insight into the cellular involvement of the two reverse gyrases from the hyperthermophilic archaeon Sulfolobus solfataricus. In: BMC Molecular Biology, Band 15, Nr. 1, September 2014, S. 18; doi:10.1186/1471-2199-15-18, PMC 4183072 (freier Volltext), PMID 25200003 (englisch).
- ↑ Anne-Cécile Déclais, Janine Marsault, Fabrice Confalonieri, Claire Bouthier de La Tour, Michel Duguet: Reverse gyrase, the two domains intimately cooperate to promote positive supercoiling. In: The Journal of Biological Chemistry. 275. Jahrgang, Nr. 26, Juni 2000, S. 19498–19504, doi:10.1074/jbc.m910091199, PMID 10748189 (englisch).
- ↑ SPO11. Initiator Of Meiotic Double Strand Breaks. Auf: GeneCards (genecards.org).
- ↑ Agnès Bergerat, Bernard de Massy, Danielle Gadelle, Paul-Christophe Varoutas, Alain Nicolas, Patrick Forterre: An atypical topoisomerase II from Archaea with implications for meiotic recombination. In: Nature. 386. Jahrgang, Nr. 6623, März 1997, S. 414–417, doi:10.1038/386414a0, PMID 9121560, bibcode:1997Natur.386..414B (englisch).
- ↑ Patrick Forterre, Agnes Bergerat, Purificacion Lopex-Garcia: The unique DNA topology and DNA topoisomerases of hyperthermophilic archaea. In: FEMS Microbiology Reviews. 18. Jahrgang, Nr. 2–3, Mai 1996, S. 237–248, doi:10.1111/j.1574-6976.1996.tb00240.x, PMID 8639331 (englisch).
- ↑ Mohea Couturier, Danièle Gadelle, Patrick Forterre, Marc Nadal, Florence Garnier: The reverse gyrase TopR1 is responsible for the homeostatic control of DNA supercoiling in the hyperthermophilic archaeon Sulfolobus solfataricus. In: Molecular Microbiology, Band 113, Nr. 2, November 2019, S. 356–368; doi:10.1111/mmi.14424, PMID 31713907 (englisch).
- ↑ M. Stros, D. Launholt, K. D. Grasser: The HMG-box: a versatile protein domain occurring in a wide variety of DNA-binding proteins. In: Cell. Mol. Life Sci. 64. Jahrgang, Nr. 19–20, Oktober 2007, S. 2590–2606, doi:10.1007/s00018-007-7162-3, PMID 17599239, PMC 11136187 (freier Volltext) – (englisch).
- ↑ Christopher S. Malarkey, Mair E.A. Churchill: The high mobility group box: the ultimate utility player of a cell. In: Trends in Biochemical Sciences. 37. Jahrgang, Nr. 12, Dezember 2012, S. 553–562, doi:10.1016/j.tibs.2012.09.003, PMID 23153957, PMC 4437563 (freier Volltext) – (englisch).
- ↑ Sophie Payne, Samuel McCarthy, Tyler Johnson, Erica North, Paul Blum: Sulfolobus solfataricus. In: PNAS! Band 115, Nr. 48, November 2018, S. 12271–12276; doi:10.1073/pnas.1808221115, PMC 6275508 (freier Volltext), PMID 30425171 (englisch).
- ↑ Annamaria Guagliardi, Laura Cerchia, Marco Moracci, Mosè Rossi: The chromosomal protein sso7d of the crenarchaeon Sulfolobus solfataricus rescues aggregated proteins in an ATP hydrolysis-dependent manner. In: The Journal of Biological Chemistry, Band 275, Nr. 41, Oktober 2000, S. 31813–31818; doi:10.1074/jbc.m002122200, PMID 10908560 (englisch).
- ↑ Erlet Shehi, Vincenzo Granata, Pompea Del Vecchio, Guido Barone, Paola Fusi, Paolo Tortora, Giuseppe Graziano: Thermal stability and DNA binding activity of a variant form of the Sso7d protein from the archeon Sulfolobus solfataricus truncated at leucine 54. In: Biochemistry. 42. Jahrgang, Nr. 27, Juli 2003, S. 8362–8368, doi:10.1021/bi034520t, PMID 12846585 (englisch).
- ↑ Herbert Baumann, Stefan Knapp, Andrej Karshikoff, Rudolf Ladenstein, Torleif Härd: DNA-binding surface of the Sso7d protein from Sulfolobus solfataricus. In: Journal of Molecular Biology, Band 247, Nr. 5, April 1995, S. 840–846; doi:10.1006/jmbi.1995.0184, PMID 7723036 (englisch).
- ↑ Annamaria Guagliardi, Alessandra Napoli, Mosé Rossi, Maria Ciaramella: Annealing of complementary DNA strands above the melting point of the duplex promoted by an archaeal protein. In: Journal of Molecular Biology. 267. Jahrgang, Nr. 4, April 1997, S. 841–848, doi:10.1006/jmbi.1996.0873, PMID 9135116 (englisch).
- ↑ Patrick Forterre, Fabrice Confalonieri, Stefan Knapp: Identification of the gene encoding archeal-specific DNA-binding proteins of the Sac10b family. In: Molecular Microbiology. 32. Jahrgang, Nr. 3, Mai 1999, S. 669–70, doi:10.1046/j.1365-2958.1999.01366.x, PMID 10320587 (englisch).
- ↑ Hong Xue, Rong Guo, Yunfei Wen, Danxu Liu, Li Huang: An abundant DNA binding protein from the hyperthermophilic archaeon Sulfolobus shibatae affects DNA supercoiling in a temperature-dependent fashion. In: Journal of Bacteriology, Band 182, Nr. 14, Juli 2000, S. 3929–3933; doi:10.1128/JB.182.14.3929-3933.2000, PMC 94576 (freier Volltext), PMID 10869069 (englisch).
- ↑ a b Manish Goyal, Chinmoy Banerjee, Shiladitya Nag, Uday Bandyopadhyay: The Alba protein family: Structure and function. In: Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1864. Jahrgang, Nr. 5, Mai 2016, S. 570–583, doi:10.1016/j.bbapap.2016.02.015, PMID 26900088 (englisch).
- ↑ Ben N. Wardleworth, R. J. M. Russell, Stephen D. Bell, Garry L. Taylor, Malcom F. White: Structure of Alba: an archaeal chromatin protein modulated by acetylation. In: The EMBO Journal. 21. Jahrgang, Nr. 17, September 2002, ResearchGate:11190521, S. 4654–4662, doi:10.1093/emboj/cdf465, PMID 12198167, PMC 125410 (freier Volltext) – (englisch).
- ↑ Stephen D. Bell, Catherine H. Botting, Benjamin N. Wardleworth, Stephen P. Jackson, Malcolm F. White: The interaction of Alba, a conserved archaeal chromatin protein, with Sir2 and its regulation by acetylation. In: Science. 296. Jahrgang, Nr. 5565, April 2002, S. 148–151, doi:10.1126/science.1070506, PMID 11935028, bibcode:2002Sci...296..148B (englisch).
- ↑ Herbert Baumann, Stefan Knapp, Thomas Lundbäck, Rudolf Ladenstein, Torleif Härd: Solution structure and DNA-binding properties of a thermostable protein from the archaeon Sulfolobus solfataricus. In: Nature Structural Biology, Band 1, Nr. 11, November 1994, S. 808–819; doi:10.1038/nsb1194-808, PMID 7634092 (englisch).
- ↑ Wolfram Zillig, Karl O. Stetter, Davorin Janeković: DNA-dependent RNA polymerase from the archaebacterium Sulfolobus acidocaldarius. In: European Journal of Biochemistry, Band 96, Nr. 3, Juni 1979, S. 597–604; doi:10.1111/j.1432-1033.1979.tb13074.x, PMID 380989 (englisch).
- ↑ a b Sabrina Fröls, Malgorzata Ajon, Michaela Wagner, Daniela Teichmann, Behnam Zolghadr, Mihaela Folea, Egbert J. Boekema, Arnold J. M. Driessen, Christa Schleper, Sonja-Verena Albers: UV-inducible cellular aggregation of the hyperthermophilic archaeon Sulfolobus solfataricus s mediated by pili formation. In: Molecular Microbiology, Band 70, Nr. 4, November 2008, S. 938–952; doi:10.1111/j.1365-2958.2008.06459.x, PMID 18990182 (englisch).
- ↑ a b Małgorzata Ajon, Sabrina Fröls, Marleen van Wolferen, Kilian Stoecker, Daniela Teichmann, Arnold J. M. Driessen, Dennis W. Grogan, Sonja-Verena Albers, Christa Schleper: UV-inducible DNA exchange in hyperthermophilic archaea mediated by type IV pili. In: Molecular Microbiology. 82. Jahrgang, Nr. 4, November 2011, S. 807–817, doi:10.1111/j.1365-2958.2011.07861.x, PMID 21999488 (englisch).
- ↑ Sabrina Fröls, Malcolm F. White, Christa Schleper: Reactions to UV damage in the model archaeon Sulfolobus solfataricus. In: Biochemical Society Transactions, Band 37, Nr. 1, Februar 2009, S. 36–41, doi:10.1042/BST0370036, PMID 19143598 (englisch).
- ↑ Harris Bernstein, Carol Bernstein: Evolutionary origin of recombination during meiosis. In: BioScience, Band 60, Nr. 7, 1. August 2010, S. 498–505; doi:10.1525/bio.2010.60.7.5 (englisch).
- ↑ a b c Thomas Ulas, S. Alexander Riemer, Melanie Zaparty, Bettina Siebers, Dietmar Schomburg: Genome-scale reconstruction and analysis of the metabolic network in the hyperthermophilic archaeon Sulfolobus solfataricus. In: PLOS ONE. 7. Jahrgang, Nr. 8, 31. August 2012, S. e43401, doi:10.1371/journal.pone.0043401, PMID 22952675, PMC 3432047 (freier Volltext), bibcode:2012PLoSO...743401U (englisch).
- ↑ a b Gwénola Simon, Jasper Walther, Nathalie Zabeti, Yannick Combet-Blanc, Richard Auria, John Van Der Oost, Laurence Casalot: Effect of O2 concentrations on Sulfolobus solfataricus P2. In: FEMS Microbiology Letters, Band 299, Nr. 2, Oktober 2009, S. 255–260; doi:10.1111/j.1574-6968.2009.01759.x, PMID 19735462 (englisch).
- ↑ Andrew J. Loder, Yejun Han, Aaron B. Hawkins, Hong Lian, Gina L. Lipscomb, Gerrit J. Schut, Matthew W. Keller, Michael W. W. Adams, Robert M. Kelly: Reaction kinetic analysis of the 3-hydroxypropionate/4-hydroxybutyrate CO2 fixation cycle in extremely thermoacidophilic archaea. In: Metabolic Engineering. 38. Jahrgang, 1. November 2016, ISSN 1096-7176, S. 446–463, doi:10.1016/j.ymben.2016.10.009, PMID 27771364, PMC 5433351 (freier Volltext) – (englisch).
- ↑ Wolfram Zillig, Karl O. Stetter, Simon Wunderl, Wolfgang Schulz, Harro Priess, Ingrid Scholz: The Sulfolobus-"Caldariella" group: Taxonomy on the basis of the structure of DNA-dependent RNA polymerases. In: Archives of Microbiology, Band 125, Nr. 3, April 1980, S. 259–269; doi:10.1007/BF00446886, bibcode:1980ArMic.125..259Z (englisch).
- ↑ Dennis W. Grogan: Phenotypic characterization of the archaebacterial genus Sulfolobus: comparison of five wild-type strains. In: Journal of Bacteriology. 171. Jahrgang, Nr. 12, Dezember 1989, S. 6710–6719, doi:10.1128/jb.171.12.6710-6719.1989, PMID 2512283, PMC 210567 (freier Volltext) – (englisch).
- ↑ Sulfolobus. Auf: MicrobeWiki. Stand: 6. August 2010. Kenyon College, Department of Biology (englisch).
- ↑ Adrian Hetzer, Hugh W. Morgan, Ian R. McDonald, Christopher J. Daughney: Microbial life in Champagne Pool, a geothermal spring in Waiotapu, New Zealand. In: Extremophiles. 11. Jahrgang, Nr. 4, Juli 2007, S. 605–614, doi:10.1007/s00792-007-0073-2, PMID 17426919 (englisch). Siehe insbes. Fig. 4A.
- ↑ Pingkan Aditiawati, Heni Yohandini, Fida Madayanti Warganegara, Akhmaloka Akhmaloka: Microbial Diversity of Acidic Hot Spring (Kawah Hujan B) in Geothermal Field of Kamojang Area, West Java-Indonesia. In: The Open Microbiology Journal. 3. Jahrgang, 24. April 2009, S. 58–66, doi:10.2174/1874285800903010058, PMID 19440252, PMC 2681175 (freier Volltext) – (englisch).
- ↑ Sri Handayani, Iman Santoso, Hans-Joachim Freisleben, Harald Huber, Andi, Fery Ardiansyah, Cenmi Mulyanto, Zessinda Luthfa, Rosari Saleh, Seruni Kusuma Udyaningsih Freisleben, Septelia Inawati Wanandi, Michael Thomm: Archaeal Life on Tangkuban Perahu-Sampling and Culture Growth in Indonesian Laboratories. In: Hayati Journal of Biosciences, Band 19, Nr. 3, September 2012, S. 150–154; doi:10.4308/hjb.19.3.150 (englisch).
- ↑ Richard D. Bryant, E. A. Gordy, Edward J. Laishley: Effect of soil acidification on the soil microflora. In: Water, Air, and Soil Pollution. 11. Jahrgang, Nr. 4, Mai 1979, S. 437, doi:10.1007/BF00283435, bibcode:1979WASP...11..437B (englisch).
- ↑ Veronika Stepankova, Sarka Bidmanova, Tana Koudelakova, Zbynek Prokop, Radka Chaloupkova, Jiri Damborsky: Strategies for Stabilization of Enzymes in Organic Solvents. In: ACS Catalysis. 3. Jahrgang, Nr. 12, 14. Oktober 2013, S. 2823–2836, doi:10.1021/cs400684x (englisch).
- ↑ R. M. Daniel, Donald A. Cowan, Hugh W. Morgan, Monique P. Curran: A correlation between protein thermostability and resistance to proteolysis. In: Biochemical Journal. 207. Jahrgang, Nr. 3, 1. Dezember 1982, ResearchGate:284124100, S. 641–644, doi:10.1042/bj2070641, PMID 6819862, PMC 1153914 (freier Volltext) – (englisch).
- ↑ Julian Quehenberger, Lu ShenLu Shen, Sonja-Verena Albers, Bettina Siebers, Oliver Spadiut: Sulfolobus – A Potential Key Organism in Future Biotechnology. In: Frontiers in Microbiology, Band 8, 12. Dezember 2017, S. 2474; doi:10.3389/fmicb.2017.02474, PMC 5733018 (freier Volltext), PMID 29312184 (englisch).
- ↑ Francesca M. Pisani, Rocco Rella, Carlo A Raia, Carla Rozzo, Roberto Nucci, Agata Gambacorta, Mario De Rosa, Mosè Rossi: Thermostable β-galactosidase from the archaebacterium Sulfolobus solfataricus Purification and properties. In: European Journal of Biochemistry, Band 187, Nr. 2, Januar 1990, S. 321–328; doi:10.1111/j.1432-1033.1990.tb15308.x, PMID 2105216 (englisch).
- ↑ Markus Hanner, Bernhard Redl, Geor Stöffle: Isolation and characterization of an intracellular aminopeptidase from the extreme thermophilic archaebacterium Sulfolobus solfataricus. In: Biochimica et Biophysica Acta (BBA) - General Subjects, Band 1033, Nr. 2, Februar 1990, ISBN 0-11-753612-1, S. 148–153; doi:10.1016/0304-4165(90)90005-H, PMID 2106344 (englisch).
- ↑ Ivano Condò, Davide Ruggero, Richard Reinhardt, Paola Londei: A novel aminopeptidase associated with the 60 kDa chaperonin in the thermophilic archaeon Sulfolobus solfataricus. In: Molecular Microbiology, Band 29, Nr. 3, August 1998, S. 775–785; doi:10.1046/j.1365-2958.1998.00971.x, PMID 9723917, Epub 1. Mai 2002 (englisch).
- ↑ a b Silvia Sommaruga, Elisabetta Galbiati, Jesus Peñaranda-Avila, Chiara Brambilla, Paolo Tortora, Miriam Colombo, Davide Prosperi: Immobilization of carboxypeptidase from Sulfolobus solfataricus on magnetic nanoparticles improves enzyme stability and functionality in organic media. In: BMC Biotechnology, Band 14, Nr. 1, 5. September 2014, S. 82; doi:10.1186/1472-6750-14-82, PMC 4177664 (freier Volltext), PMID 25193105 (englisch).
- ↑ 5L2P. Structure of arylesterase; Classification: HYDROLASE; Organism(s): Saccharolobus solfataricus (rcsb.org).
- ↑ a b c Yun-Ho Choi, Young-Jun Park, Sung-Jin Yoon, Hee-Bong Lee: Purification and characterization of a new inducible thermostable extracellular lipolytic enzyme from the thermoacidophilic archaeon Sulfolobus solfataricus P1. In: Journal of Molecular Catalysis B: Enzymatic, Band 124, 2016, Februar S. 11–19; doi:10.1016/j.molcatb.2015.11.023 (englisch).
- ↑ a b Dong-Chol Li, Fan Yang, Bo Lu, Dian-Fu Chen, Wei-Jun Yang: Thermotolerance and molecular chaperone function of the small heat shock protein HSP20 from hyperthermophilic archaeon, Sulfolobus solfataricus P2. In: Cell Stress and Chaperones, Band 17, Nr. 1, August 2011, S. 103–108; doi:10.1007/s12192-011-0289-z, PMC 3227843 (freier Volltext), PMID 21853411 (englisch).
- ↑ Laura Cerchia, Mosè Rossi, Annamaria Guagliardi: An archaeal chaperonin-based reactor for renaturation of denatured proteins. In: Extremophiles. 4. Jahrgang, Nr. 1, 7. August 1999, S. 1–7, doi:10.1007/s007920050001, PMID 10741831 (englisch).
- ↑ Girishchandra B. Patel, G. Dennis Sprott: Archaeobacterial Ether Lipid Liposomes (Archaeosomes) as Novel Vaccine and Drug Delivery Systems. In: Critical Reviews in Biotechnology. 19. Jahrgang, Nr. 4, 1999, Epub 29. September 2008, S. 317–357, doi:10.1080/0738-859991229170, PMID 10723627 (englisch).
- ↑ a b c d e f g BacDive: Search: "Saccharolobus solfataricus". Bacterial Diversity Metadatabase (bacdive.dsmz.de).
- ↑ a b Ruben M. Ceballos, Caleb D. Marceau, Joshua O. Marceau, Steven Morris, Adam J. Clore, Kenneth M. Stedman: Differential virus host-ranges of the Fuselloviridae of hyperthermophilic Archaea: implications for evolution in extreme environments. In: Frontiers in Microbiology, Sec. Extreme Microbiology, Band 3, 24. August 2012; doi:10.3389/fmicb.2012.00295 (englisch).
- ↑ Chu-yu Fu, Kang Wang, Lu Gan, Jason Lanman, Reza Khayat, Mark J. Young, Grant J. Jensen: Sulfolobus solfataricus Strain 2-1-12. Caltech Data, 2010 (data.caltech.edu).
- ↑ Blake Wiedenheft, Kenneth Stedman, Francisco Roberto, Deborah Willits, Anne-Kathrin Gleske, Luisa Zoeller, Jamie Snyder, Trevor Douglas, Mark Young: Comparative Genomic Analysis of Hyperthermophilic Archaeal Fuselloviridae Viruses. In: Journal of Virology, Band 58, Nr. 4, 15 Februar 2004; doi:10.1128/jvi.78.4.1954-1961.2004, PMC 369504 (freier Volltext), PMID 14747560 (englisch)
Weiterführende Literatur
- Gabriella Fiorentino, Immacolata Del Giudice, Luigi Petraccone, Simonetta Bartolucci, Pompea Del Vecchio: Conformational stability and ligand binding properties of BldR, a member of the MarR family, from Sulfolobus solfataricus. In: Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, Band 1844, Nr. 6, Juni 2014, S. 1167–1172; doi:10.1016/j.bbapap.2014.03.011, PMID 24704039 (englisch).
- Matteo de Rosa, Francesco Bemporad, Sara Pellegrino, Fabrizio Chiti, Martino Bolognesi, Stefano Ricagno: Edge strand engineering prevents native-like aggregation in Sulfolobus solfataricus acylphosphatase. In: The FEBS Journal, Band 281, Nr. 18, September 2014, S. 4072–4084; doi:10.1111/febs.12861, PMID 24893801 (englisch).
- Roland Gamsjaeger, Ruvini Kariawasam, Christine Touma, Ann H. Kwan, Malcolm F. White, Liza Cubeddu: Backbone and side-chain 1H, 13C and 15N resonance assignments of the OB domain of the single stranded DNA binding protein from Sulfolobus solfataricus and chemical shift mapping of the DNA-binding interface. In: Biomolecular NMR Assignments, Band 8, Nr. 2, Oktober 2014, S. 243–246; doi:10.1007/s12104-013-9492-4, PMID 23749431 (englisch).
- Jianjun Wang, Junge Zhu, Cong Min, Sheng Wu: CBD binding domain fused γ-lactamase from Sulfolobus solfataricus is an efficient catalyst for (-) γ-lactam production. In: BMC Biotechnology, Band 14, Mai 2014, S. 40; doi:10.1186/1472-6750-14-40, PMC 4041915 (freier Volltext), PMID 24884655 (englisch).