Pachyviridae
| Pachyviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
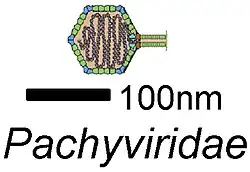
Schemazeichnung | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Pachyviridae | ||||||||||||||
| Links | ||||||||||||||
|
Pachyviridae ist eine im März 2022 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp C (Podoviren). Ihre Wirte sind Bakterien, was sie als Bakteriophagen klassifiziert.[1][2] Ihre Wirte sind Bakterien der Familie Flavobacteriaceae (Gattungen Cellulophaga und Tenacibaculum[3]).
Beschreibung
Die Familie Duneviridae wurde (zusammen mit anderen wie den Duneviridae) aufgrund eines Vorschlags eingerichtet, der darauf abzielte, einigen zu diesem Zeitpunkt neuen bzw. noch nicht klassifizierten Viren in die offizielle taxonomische Systematik aufzunehmen, die als sog. Flavophagen Mitglieder der Bakterien-Klasse Flavobacteriia infizieren.
Die Isolierung und Charakterisierung dieser neuen Flavophagen sowie die Suche nach verwandten Phagen aus Sequenzdatenbanken wird in Nina Bartlau et al. (2021/2022) beschrieben.[4]
Die meisten der Phagen wurden aus Oberflächenmeerwasser isoliert, das während der Phytoplanktonblüten im Frühjahr von der ökologischen Langzeitforschungsstation Helgoland Reede (englisch Helgoland Roads) beprobt wurde.[5][6][7]
Die innere Systematik der Familie basiert auf der Analyse nach fünf verschiedenen Methoden:
- Nukleotid-basierte Ähnlichkeiten zwischen der verschiedenen Genomen, berechnet per VIRIDIC Web-Service[8]
- Protein-basiertes hierarchische Cluster-Analyse, berechnet mit VirClust[9]
- Aminosäure-basierte phylogenetische Bäume basierend auf dem Ganzgenom, berechnet mit Viral Proteomic Tree (ViPTree)
- Aminosäure-basierte phylogenetische Bäume basierend auf dem Ganzgenom, berechnet mit der Virus Classification and Tree Building Online Resource (VICTOR)
- Kernprotein-basierte Bäume mit IQ-Trees
Die innerfamiliäre Struktur und die Beziehungen wurden wurde mit VirClust berechnet, ein Alignement mit MUSCLE (v3.8.425) vorgenommen.
Die zur Familie Pachyviridae zusammengefassten Gründungsmitglieder bilden in VirClust und VICTOR eine monophyletische Gruppe (Klade) mit 11 gemeinsamen Kerngenen, darunter ein Portalprotein, Schwanzhülle (englisch sheath protein) und Strukturproteine.
Gattung Gundelvirus
Die Mitglieder der Gattung Gundelvirus zeigen – per Vorgabe – eine Ähnlichkeit in ihrer DNA-Sequenz von (mindestens) 70 %. Kriterium für die Abgrenzung der Art Gundelvirus Gundel ist 95 % intergenomische Nukleotid-Ähnlichkeit. Die Art Gundelvirus Gundel wurde bisher (Stand Mai 2021) zweimal isoliert, vor und nach dem Phytoplankton-Hoch im Jahr 2018, mit Wirten Tenacibaculum sp. AHE14PA (DSM 111040) und Tenacibaculum sp. AHE15PA (DSM 111039), wie Cellulophaga aus der Familie Flavobacteriaceae.[10] Der Phage hat den Morphotyp der Podoviren mit einem Kapsiddurchmesser von 60,5 ± 5,2 nm, die Schwanzlänge beträgt 22,7 ± 3,2 nm. Es wurden insgesamt zehn Isolate gewonnen, die alle eine Genomgröße von 78.511 bp und einen GC-Gehalt von 30,4 % aufwiesen. Das Genom hat an den Enden kurze direkte terminale Wiederholungen (englisch direct terminal repeats, DTRs). Es kodiert Gene für Schwanzfaser, Portal, L-Alanin-D-Glutamin-Peptidase und ein Phagen-Antirepressorprotein. Darüber hinaus hat Gundel 10 tRNAs. Gundel kann nur seine beiden Isolationswirte AHE14PA und AHE15PA infizieren, die zu 99,87 % ähnliche 16S rRNA aufweisen.[5]
Gattung Baltivirus
Auch die Mitglieder der Gattung Baltivirus zeigen – per Vorgabe – eine Ähnlichkeit in ihrer DNA-Sequenz von (mindestens) 70 %. Kriterium für die Abgrenzung der Gründungs-Arten Baltivirus phi19tres, Baltivirus phi13duo und Baltivirus phi18tres ist jeweils analog ebenfalls 95 % intergenomische Nukleotid-Ähnlichkeit innerhalb der Art. Ihre Virionen (Phagenpartikel) sind alle vom Morphotyp der Podoviren.[5]
Die Art Baltivirus phi19tres wurde erstmals im Jahr 2005 mit ihrem Wirt Cellulophaga sp. #19 (nach anderer Taxonomie Cellulophaga baltica #19) aus dem Öresund: Cellulophaga-Phage phi19:3 (ϕ19:3). Die Genomgröße des Phagen beträgt 75.991 bp.[5]
Die Art Baltivirus phi13duo wurde ebenfalls erstmals im Jahr 2005 aus dem Öresund isoliert, ihr Wirt ist Cellulophaga sp. #13 [Cellulophaga baltica #13]: Cellulophaga-Phage phi13:2 (ϕ13:2). Die Genomgröße von ϕ13:2 ist 72.369 bp.[5]
Auch die Art Baltivirus phi18tres wurde erstmals im Jahr 2005 vom Öresund isoliert. Ihr Wirt ist Cellulophaga sp. #18 [Cellulophaga baltica #18]. Die Genomgröße beträgt hier 71.442 bp.[5]
Gattung Bacelvirus
Auch für die dritte Gründungs-Gattung der Familie, Bacelvirus, wird eine Ähnlichkeit der DNA-Sequenzen ihrer Mitglieder von (mindestens) 70 % vorgegeben. Auch ihre Art Bacelvirus phi46tres wurde erstmals 2005 aus dem Öresu8nd isoliert. Ihr exemplarischer Vertreter, Cellulophaga-Phage phi46:3 (ϕ46:3) hat eine Genomgröße von 72.960 bp und den Podoviren-Morphotyp. Per Vorgabe ist beträgt die intergenomische Nukleotid-Ähnlichkeit innerhalb dieser Art wie üblich ebenfalls %.[5]
Systematik
Die Systematik der Pachyviridae ist nach International Committee on Taxonomy of Viruses (ICTV) – (MSL#40v1) mit Stand 3. Mai 2025 wie folgt:[1][11][12]
Familie Pachyviridae
- ohne zugewiesene Unterfamilie
- Gattung Bacelvirus
- Gattung Baltivirus (früher Cba183likevirus)
- Spezies Baltivirus phi13duo, mit
- Cellulophaga-Phage phi13:2 (ϕ13:2)[15]
- – Wirt: Cellulophaga sp. #13 [Cellulophaga baltica #13][16]
- – Fundort: Öresund, südl. Kattegat (Dänemark), in 1 m Tiefe, am 13. Mai 2005
- Spezies Baltivirus phi18tres, mit
- Cellulophaga-Phage phi18:3 (ϕ18:3)[13]
- – Wirt: Cellulophaga sp. #18 [Cellulophaga baltica #18][17]
- – Fundort: Öresund, südl. Kattegat (Dänemark), in 1 m Tiefe, am 13. Mai 2005
- Spezies Baltivirus phi19tres, mit
- Cellulophaga-Phage phi19:3 (ϕ19:3)[18]
- – Wirt: Cellulophaga sp. #19 [Cellulophaga baltica #19][19]
- – Fundort: Öresund, südl. Kattegat (Dänemark), in 1 m Tiefe, am 13. Mai 2005
- Spezies Baltivirus phi13duo, mit
- Gattung Gundelvirus
- Spezies Gundelvirus Gundel [Tenacibaculum-Phage Gundel[20]], mit
- Tenacibaculum-Phage Gundel_1 (DSM 111235)[3]
- – Wirte: Tenacibaculum sp. AHE14PA (DSM 111040) und T. sp. AHE15PA (DSM 111039), Flavobacteriaceae.[3]
- – Fundort: SW vom Dünenhafen, Insel Düne, Helgoland, Nordsee; 19. April 2018
- Spezies Gundelvirus Gundel [Tenacibaculum-Phage Gundel[20]], mit
Namensherkunft (Etymologie)
Der Name der Familie Pachyviridae kommt von altgriechisch παχύς pakhús, deutsch ‚dick‘ und bezieht sich auf die Morphologie der Familienmitglieder, die pummelig sind und als Podoviren keinen langen „Schwanz“ haben; der Suffix ‚-viridae‘ kennzeichnet Virusfamilien.[2][5]
- Die Gattung Baltivirus ist nach dem Fundort ihrer Gründungsmitglieder benannt, der Ostsee (alias Baltisches Meer, englisch Baltic Sea).[5]
- Der Name der Gattung Bacelvirus ist ein Kofferwort, zusammengezogen aus Baltic [Sea] (für Ostsee) und dem Namen der Wirtsgattung des exemplarischen Vertreters, Cellulophage.[5]
- Die Gattung Gundelvirus ist nach ihrem exemplarischen Vertreter, Tenacibaculum-Phage Gundel_1, benannt.[5] Gundel ist ein von althochdeutsch gund ‚Kampf‘ herrührender weiblicher friesischer Vorname, der Sprache, die auf Helgoland gesprochen wird, wo die Gründungsmitglieder der Gattung im umgebenden Meereswasser entdeckt wurden.
Einzelnachweise
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Family: Pachyviridae .
- ↑ a b c Tenacibaculum phage Gundel_1 (Gundelvirus Gundel). DSM 111235. Auf: DSMZ (dsmz.de)
- ↑ Nina Bartlau, Antje Wichels, Georg Krohne, Evelien M. Adriaenssens, Anneke Heins, Bernhard M Fuchs, Rudolf Amann, Cristina Moraru: Highly diverse flavobacterial phages isolated from North Sea spring blooms. In: Oxford Academic: The ISME Journal, Band 16, Nr. 2, Februar 2022, S. 555–568; doi:10.1038/s41396-021-01097-4, PMC 8776804 (freier Volltext), PMID 34475519, Epub 2. September 2021 (englisch).
- ↑ a b c d e f g h i j k Nina Bartlau, Liliana Cristina Moraru, Antje Wichels, Karin Holmfeldt, Evelien M. Adriaenssens, Rudolf Amann: Create nine new families (Pachyviridae, Pervagoviridae, Duneviridae, Helgolandviridae, Duneviridae, Molycolviridae, Winoviridae, Forsetiviridae, and Aggregaviridae) including 13 new genera and 18 new species (Caudoviricetes). Vorschlag 2021.029B (zip:docx), Filelist. Mai 2021.
- ↑ Hanno Teeling et al.: Helgoland Roads. Spring blooms at Helgoland. Max-Planck-Institut für Marine Mikrobiologie, Abteilung Molekulare Ökologie (mpi-bremen.de).
- ↑ Inga Kirstein et al.: AG Langzeitbeobachtungen (LTO). Alfred-Wegener-Institut (awi.de).
- ↑ VIRIDIC Web-Service (viridic.icbm.de).
- ↑ VirClust (virclust.icbm.de).
- ↑ LPSN: Genus Tenacibaculum Suzuki et al. 2001.
- ↑ ICTV: Master Species Lists (MSL).
- ↑ NCBI Taxonomy Browser: Pachyviridae, Details: Pachyviridae (family).
- ↑ a b Karin Holmfeldt, Natalie Solonenko, Manesh Shah, Kristen Corrier, Lasse Riemann, Nathan C. Verberkmoes, Matthew B. Sullivan: Twelve previously unknown phage genera are ubiquitous in global oceans, In: PNAS, Band 110, Nr. 31, 30. Juli 2013, S. 12798–12803; doi:10.1073/pnas.1305956110, PMC 3732932 (freier Volltext), PMID 23858439, ResearchGate:249649168, Epub 15. Juli 2013 (englisch).
- ↑ NCBI Taxonomy Browser: Cellulophaga phage phi46:3; Nucleotide: Cellulophaga phage phi46:3, complete genome. Accession: KC821622.
- ↑ Oliver W. Bayfield, Andrey N. Shkoporov, Natalya Yutin, Ekaterina V. Khokhlova, Jake L. R. Smith, Dorothy E. D. P. Hawkins, Eugene V. Koonin, Colin Hill, Alfred A. Antson: Structural atlas of a human gut crassvirus. In: Nature, Band 617, 3. Mai 2023, S. 409–416; doi:10.1038/s41586-023-06019-2 (englisch).
- ↑ NCBI Taxonomy Browser: Cellulophaga sp. #13.
- ↑ NCBI Taxonomy Browser: Cellulophaga sp. #18.
- ↑ Karin Holmfeldt, Mathias Middelboe, Ole Nybroe, Lasse Riemann: Large Variabilities in Host Strain Susceptibility and Phage Host Range Govern Interactions between Lytic Marine Phages and Their Flavobacterium Hosts. In: Applied and Environmental Microbiology, Nand 73, Nr. 22, 31. August 2007, S. 6730–6739; doi:10.1128/AEM.01399-07, PMC 2074958 (freier Volltext), PMID 17766444, (englisch).
- ↑ NCBI Taxonomy Browser: Cellulophaga sp. #19.
- ↑ NCBI Taxonomy Browser: Tenacibaculum phage Gundel (species) und als Spezies synonym Tenacibaculum phage Gundel_1.