Intestinimonas
| Intestinimonas | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|

Gram-Färbung von Intestinimonas massiliensis Stamm GD2T | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Intestinimonas | ||||||||||||
| Kläring et al. 2013 |
Intestinimonas ist eine Gattung von Bakterien. Die Arten kommen im Darm von Menschen und von Tieren vor.[1] Als Endprodukt der Fermentation bilden sie meist Butyrat. Diese Verbindung spielt eine wichtige Rolle innerhalb des Darms und des Immunsystems. Von daher sind einige Arten als Probiotika von Interesse.[2]
Merkmale und Stoffwechsel
Die Zellen der Arten von Intestinimonas sind stäbchen- oder spindelförmig wie z.B. der Art I. gabonensis. Einige Arten, wie I. butyriciproducens, bilden Sporen. Die Gram-Färbung verläuft meist gramnegativ, die Arten haben aber eine grampositive Zellwandstruktur.
Der aus dem menschlichen Darm stammende Stamm Intestinimonas butyriciproducens AF211 kann die Aminosäure Lysin vollständig durch die Gärung in Butyrat und Acetat umwandeln.[2] Diese Fähigkeit eines Damrmikrobiums war bei der Beschreibung eine neue Entdeckung (siehe weiter unten).
Genomische Analysen von Intestinimonas butyriciproducens Stamm AF211 Intestinimonas butyriciproducens Stamm SRB521T ergaben einen vollständigen Glykolyseweg, während der Pentosephosphatweg und der Citrat-Zyklus bei beiden Stämmen unvollständig waren. In-vitro-Tests bestätigten eine gemischte Säuregärung unter Verwendung verschiedener Kohlenhydratsubstrate, die zur Produktion von Butyrat als Hauptprodukt und Ethanol, Isobutyrat bzw. Laktat als Nebenmetaboliten führte. Die beiden Stämme wuchsen mit Hexosen schlecht, aber das Wachstum wurde durch Zugabe von Acetat deutlich verbessert.
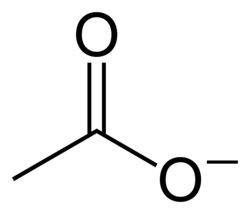
Eine ähnliche Eigenschaft wurde zuvor für Faecalibacterium prausnitzii beschrieben: Die Anwesenheit von Acetat erhöhte die Energiegewinnung aus Glukose durch diese durch Stimulation des Acetyl-CoA-Weges.[3][4][2] Dieser Stoffwechselweg verfügt über einen Butyryl-CoA-Dehydrogenase (Bcd)-Elektronen-übertragenden Flavoprotein-Komplex (Etf), der über einen membranintegrierten Rnf-Komplex eine Protonenmotorische Kraft erzeugt.[5][2]
| Intestinimonas butyriciproducens SRB521-5-1 (DSM26588T) | Intestinimonas butyriciproducens AF211 | “Intestinimonas massiliensis GD2” (DSM00417T) | |
|---|---|---|---|
| Fundort | Maus | Mensch | Mensch |
| Beweglichkeit | Negativ | Negativ | Negativ |
| Sporenbildung | Ja | Ja | Nein |
| Fermentation von Lysin | Ja | Ja | Gene vorhanden |
| Fructoselysin-Umwandlung (Fructoselysin-Kinase) | Ja | Ja | Gene vorhanden |
| Wachstum bei 45 °C | Ja | Nein | Nein |
| GC-Gehalt (Mol%) | 59,4 | 59,1 | 60,68 |
Probiotika
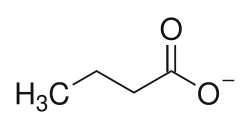
Arten von Intestinimonas bilden durch ihren Stoffwechsel u.a. Butyrat als Endprodukt. Die Verbindung Butyrat und Butyrat produzierende Bakterien haben zunehmend an Aufmerksamkeit gewonnen, da sie beide eine wichtige Rolle bei der Aufrechterhaltung der Homöostase des Darms und der Integrität des Darmepithels spielen. Butyrat ist ein wichtiges Substrat für Kolonozyten und soll gegen Krebs wirken (antikarzinogene) sowie entzündungshemmende und antioxidative Wirkungen haben.[6][7][2] Interessanterweise wurden bei entzündlichen Darmerkrankungen, Dickdarmkrebs und Typ-2-Diabetes verminderte Mengen von Butyrat bildenden (butyrogenen) Bakterien beobachtet.[2][8][9][10] Butyrat produzierende Mikroben sind von daher als Probiotika von Interesse.
Bislang wurden mehrere Butyrat-produzierende Bakterien isoliert, die hauptsächlich zu den Clostridium-Clustern XIVa und IV gehören, die viele Mitglieder der Familie Lachnospiraceae bzw. Ruminococcaceae (nun als Oscillospiraceae geführt) versammeln. Diese Bakterien produzieren Butyrat hauptsächlich über den Acetyl-CoA-Weg.[2] Wie oben beschrieben kann Intestinimonas butyriciproducens AF211 auch die Aminosäure Lysin und die Verbindung Fructoselysin vollständig durch die Gärung in Butyrat und Acetat umwandeln. Diese Eigenschaft ist einzigartig für das Darmökosystem und verbindet zwei wichtige Stoffwechselwege, die Bildung von Butyrat (Butyrogenese) und die Aminosäurefermentation im Darmtrakt. Andere Stämme von Intestinimonas butyriciproducens wurden auch aus Tieren, wie Schweinen, isoliert, vermutlich sind sie unter Säugern weit verbreitet.[2]
Intestinimonas kann des Weiteren die Belastung durch Amadori-Produkte, die sich potenziell zu krebserregenden Advanced Glycation Endproducts (AGEs) entwickeln könnten, reduzieren und in eine nützliche Verbindung (Butyrat) umwandeln.[11] Die Reaktion läuft ohne Enzyme ab, von daher spricht man von der Glykation. Amadori-Produkte entstehen z.B. beim Braten, Rösten oder Backen. Fructoselysin gehört zu den häufigsten Amadori-Produkten und ist auch ein Vorläufer von Advanced Glycation Endproducts (AGEs). Die Umwandlung von Fructoselysin in Butyrat könnte eine evolutive Anpassung des Bakteriums an den Verzehr des Wirtes von Amadori-Produkten sein, die das Ergebnis einer thermischen Behandlung von Lebensmitteln sind. Die Amadori-Produkte werden mit dem Alterungsprozess und chronischen Krankheiten in Verbindung gebracht.[12][11] Die sich daraus bildenden AGEs werden mit der Entstehung von Krebs und diabetischen Komplikationen in Verbindung gebracht, und daher könnte die Entfernung von AGEs (oder Vorläufern von AGEs) ein wichtiger Schritt zur Verringerung des Krebs- und Diabetesrisikos sein.[11]
I. butyriciproducens produziert auch das Pseudovitamin B12, das für die Darmmikroben und – direkt oder indirekt – für den Wirt von Nutzen ist.
Intestinimonas aquisgranensis wurde mit Hilfe der neuen Methode namens "Single-Cell Dispensing (SCD)" isoliert. Dies ist eine Methode zur gezielten Trennung und Sortierung einzelner, verschiedener Zellen in Mikrotiterplatten, auf Chips oder in Tröpfchen. Diese Methode wird zur Isolierung einzelner Bakterien zur genomischen Charakterisierung oder Kultivierung bislang unbekannter Mikroorganismen („culturomics“) genutzt. Bei dieser Untersuchung wurden neben Intestinimonas aquisgranensis 82 menschliche Darmbakterienarten aus fünf Phyla (Actinobacteriota, Bacteroidota, Desulfobacterota, Bacillota (Firmicutes) und Proteobacteria) und 24 Familien gewonnen, darunter befanden sich die ersten kultivierten Vertreter von 11 neuen Gattungen und 10 neuen Arten, die taxonomisch vollständig charakterisiert wurden.[13]
Systematik
Bakterien der Gattung Intestinimonas zählen zu der Ordnung Eubacteriales innerhalb der Abteilung Bacillota (früher als Firmicutes bezeichnet). Phylogenetisch nahe stehen Mitglieder der Gattungen Flavonifractor und Pseudoflavonifractor.
Es folgt eine Liste einiger Arten:[14]
- Intestinimonas aquisgranensis Afrizal et al. 2023
- Intestinimonas butyriciproducens Kläring et al. 2013
- "Intestinimonas gabonensis" Mourembou et al. 2017
- Intestinimonas massiliensis Afouda et al. 2020
- "Candidatus Intestinimonas merdavium" Gilroy et al. 2021
- "Candidatus Intestinimonas pullistercoris" Gilroy et al. 2021
- "Candidatus Intestinimonas stercoravium" Gilroy et al. 2021
- "Candidatus Intestinimonas stercorigallinarum" Gilroy et al. 2021
Einzelnachweise
- ↑ Caroline M. Plugge, Nam TP Bui: Intestinimonas (2019) In: Bergey's Manual of Systematics of Archaea and Bacteria. 1. Auflage. Wiley, 2015, ISBN 978-1-118-96060-8, doi:10.1002/9781118960608.gbm01736 (wiley.com [abgerufen am 17. April 2025]).
- ↑ a b c d e f g h Thi Phuong Nam Bui, Sudarshan Anand Shetty, Ilias Lagkouvardos, Jarmo Ritari, Bhawani Chamlagain, François P. Douillard, Lars Paulin, Vieno Piironen, Thomas Clavel, Caroline M. Plugge, Willem M. de Vos: Comparative genomics and physiology of the butyrate‐producing bacterium Intestinimonas butyriciproducens. In: Environmental Microbiology Reports. Band 8, Nr. 6, Dezember 2016, ISSN 1758-2229, S. 1024–1037, doi:10.1111/1758-2229.12483 (wiley.com [abgerufen am 20. April 2025]).
- ↑ Almut Heinken, M. Tanweer Khan, Giuseppe Paglia, Dmitry A. Rodionov, Hermie J. M. Harmsen, Ines Thiele: Functional Metabolic Map of Faecalibacterium prausnitzii, a Beneficial Human Gut Microbe. In: Journal of Bacteriology. Band 196, Nr. 18, 14. August 2014, S. 3289–3302, doi:10.1128/jb.01780-14, PMID 25002542, PMC 4135701 (freier Volltext) – (asm.org [abgerufen am 21. April 2025]).
- ↑ Sylvia H. Duncan und Harry J. Flint: Faecalibacterium In: Bergey's Manual of Systematics of Archaea and Bacteria. 1. Auflage. Wiley, 2015, ISBN 978-1-118-96060-8, doi:10.1002/9781118960608.gbm00674 (wiley.com [abgerufen am 14. November 2024]).
- ↑ Fuli Li, Julia Hinderberger, Henning Seedorf, Jin Zhang, Wolfgang Buckel, Rudolf K. Thauer: Coupled Ferredoxin and Crotonyl Coenzyme A (CoA) Reduction with NADH Catalyzed by the Butyryl-CoA Dehydrogenase/Etf Complex from Clostridium kluyveri. In: Journal of Bacteriology. Band 190, Nr. 3, Februar 2008, S. 843–850, doi:10.1128/jb.01417-07, PMID 17993531, PMC 2223550 (freier Volltext) – (asm.org [abgerufen am 21. April 2025]).
- ↑ Brian F. Hinnebusch, Shufen Meng, James T. Wu, Sonia Y. Archer, Richard A. Hodin: The Effects of Short-Chain Fatty Acids on Human Colon Cancer Cell Phenotype Are Associated with Histone Hyperacetylation. In: The Journal of Nutrition. Band 132, Nr. 5, Mai 2002, S. 1012–1017, doi:10.1093/jn/132.5.1012 (elsevier.com [abgerufen am 21. April 2025]).
- ↑ Lidija Klampfer, Jie Huang, Takehiko Sasazuki, Senji Shirasawa und Leonard Augenlicht: Inhibition of Interferon γ Signaling by the Short Chain Fatty Acid Butyrate In: Molecular Cancer Ressource (2003), 1 (11): S. 855–862.
- ↑ Harry J. Flint, Karen P. Scott, Petra Louis, Sylvia H. Duncan: The role of the gut microbiota in nutrition and health. In: Nature Reviews Gastroenterology & Hepatology. Band 9, Nr. 10, Oktober 2012, ISSN 1759-5045, S. 577–589, doi:10.1038/nrgastro.2012.156 (nature.com [abgerufen am 21. April 2025]).
- ↑ Junjie Qin, Yingrui Li, Zhiming Cai, Shenghui Li, Jianfeng Zhu, Fan Zhang, Suisha Liang, Wenwei Zhang, Yuanlin Guan, Dongqian Shen, Yangqing Peng, Dongya Zhang, Zhuye Jie, Wenxian Wu, Youwen Qin, Wenbin Xue, Junhua Li, Lingchuan Han, Donghui Lu, Peixian Wu, Yali Dai, Xiaojuan Sun, Zesong Li, Aifa Tang, Shilong Zhong, Xiaoping Li, Weineng Chen, Ran Xu, Mingbang Wang, Qiang Feng, Meihua Gong, Jing Yu, Yanyan Zhang, Ming Zhang, Torben Hansen, Gaston Sanchez, Jeroen Raes, Gwen Falony, Shujiro Okuda, Mathieu Almeida, Emmanuelle LeChatelier, Pierre Renault, Nicolas Pons, Jean-Michel Batto, Zhaoxi Zhang, Hua Chen, Ruifu Yang, Weimou Zheng, Songgang Li, Huanming Yang, Jian Wang, S. Dusko Ehrlich, Rasmus Nielsen, Oluf Pedersen, Karsten Kristiansen, Jun Wang: A metagenome-wide association study of gut microbiota in type 2 diabetes. In: Nature. Band 490, Nr. 7418, Oktober 2012, ISSN 0028-0836, S. 55–60, doi:10.1038/nature11450 (nature.com [abgerufen am 21. April 2025]).
- ↑ Willem M. de Vos, Max Nieuwdorp: A gut prediction. In: Nature. Band 498, Nr. 7452, Juni 2013, ISSN 0028-0836, S. 48–49, doi:10.1038/nature12251 (nature.com [abgerufen am 21. April 2025]).
- ↑ a b c Seegers, J.F.M.L., Bui, T.P.N., de Vos, W.M. (2021): Remarkable Metabolic Versatility of the Commensal Bacteria Eubacterium hallii and Intestinimonas butyriciproducens: Potential Next-Generation Therapeutic Microbes. In: Mojgani, N., Dadar, M. (eds): Probiotic Bacteria and Postbiotic Metabolites: Role in Animal and Human Health. Microorganisms for Sustainability Vol 2. Springer, Singapore. doi:10.1007/978-981-16-0223-8_5
- ↑ Veronika Maria Deppe, Johannes Bongaerts, Timothy O’Connell, Karl-Heinz Maurer, Friedhelm Meinhardt: Enzymatic deglycation of Amadori products in bacteria: mechanisms, occurrence and physiological functions. In: Applied Microbiology and Biotechnology. Band 90, Nr. 2, April 2011, ISSN 0175-7598, S. 399–406, doi:10.1007/s00253-010-3083-4 (springer.com [abgerufen am 21. April 2025]).
- ↑ Afrizal Afrizal, Thomas C. A. Hitch, Alina Viehof, Nicole Treichel, Thomas Riedel, Birte Abt, Eva M. Buhl, Dietrich Kohlheyer, Jörg Overmann, Thomas Clavel: Anaerobic single‐cell dispensing facilitates the cultivation of human gut bacteria. In: Environmental Microbiology. Band 24, Nr. 9, September 2022, ISSN 1462-2912, S. 3861–3881, doi:10.1111/1462-2920.15935 (wiley.com [abgerufen am 21. April 2025]).
- ↑ LPSN
Literatur
- Caroline M. Plugge, Nam TP Bui: Intestinimonas (2019) In: Bergey's Manual of Systematics of Archaea and Bacteria. 1. Auflage. Wiley, 2015, ISBN 978-1-118-96060-8, doi:10.1002/9781118960608.gbm01736 (wiley.com [abgerufen am 17. April 2025]).
- Thi Phuong Nam Bui, Sudarshan Anand Shetty, Ilias Lagkouvardos, Jarmo Ritari, Bhawani Chamlagain, François P. Douillard, Lars Paulin, Vieno Piironen, Thomas Clavel, Caroline M. Plugge, Willem M. de Vos: Comparative genomics and physiology of the butyrate‐producing bacterium Intestinimonas butyriciproducens. In: Environmental Microbiology Reports. Band 8, Nr. 6, Dezember 2016, ISSN 1758-2229, S. 1024–1037, doi:10.1111/1758-2229.12483 (wiley.com [abgerufen am 20. April 2025]).
Weiterführende Literatur
- Elizabeth A. Rettedal, Heidi Gumpert, Morten O.A. Sommer: Cultivation-based multiplex phenotyping of human gut microbiota allows targeted recovery of previously uncultured bacteria. In: Nature Communications. Band 5, Nr. 1, 28. August 2014, ISSN 2041-1723, doi:10.1038/ncomms5714 (nature.com [abgerufen am 20. April 2025]).
- Thi Phuong Nam Bui, Antonio Dario Troise, Bart Nijsse, Giovanni N Roviello, Vincenzo Fogliano, Willem M. de Vos: Intestinimonas-like bacteria are important butyrate producers that utilize Nε-fructosyllysine and lysine in formula-fed infants and adults. In: Journal of Functional Foods. Band 70, Juli 2020, S. 103974, doi:10.1016/j.jff.2020.103974 (elsevier.com [abgerufen am 22. April 2025]).
- Elena Rampanelli, Nadia Romp, Antonio Dario Troise, Jakshana Ananthasabesan, Hao Wu, Ismail Sahin Gül, Sabrina De Pascale, Andrea Scaloni, Fredrik Bäckhed, Vincenzo Fogliano, Max Nieuwdorp, Thi Phuong Nam Bui: Gut bacterium Intestinimonas butyriciproducens improves host metabolic health: evidence from cohort and animal intervention studies. In: Microbiome. Band 13, Nr. 1, 20. Januar 2025, ISSN 2049-2618, doi:10.1186/s40168-024-02002-9, PMID 39833973, PMC 11744835 (freier Volltext) – (biomedcentral.com [abgerufen am 22. April 2025]).