Vandenendeviridae
| Vandenendeviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
.png)
EM-Aufnahme von Pseudomonas-Phage PAK_P2 | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Vandenendeviridae | ||||||||||||||
| Links | ||||||||||||||
Vandenendeviridae ist eine im April 2024 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp A (Myoviren). Ihre Wirte sind Bakterien, was sie als Bakteriophagen klassifiziert.[2][3]
Beschreibung
Die Vandenendeviridae wurden mit Hilfe der Clusteranalyse-Tools VIRIDIC, ViPTree, VIRCLUST und vConTACT v.3.0 als zusammenhängende Verwandtschaftsgruppe lytischer Pseudomonas-Myoviren identifiziert. Die DNA-Sequenzähnlichkeit der Gründungsmitglieder beträgt mindestens 12,2 %; zudem haben sie 15 Proteine („Kernproteine“) gemeinsam. Als die Familie neu eingerichtet wurde, waren bereits vier Mitgliedsgattungen bekannt: Baldwinvirus, Nankokuvirus, Otagovirus, Flaumdravirus und Pakpunavirus; gleichzeitig mit der Familie wurden sieben weitere (ursprünglich) monotypische Gattungen neu eingerichtet: Weillhallvirus, Omahavirus, Torinovirus, Yunamivirus, Ventosusvirus, Uavernvirus und Chemalvirus, sowie Shenlongvirus; dazu kam die neuen Gattung Tartuvirus mit vier Arten und Kremarvirus mit zwei Arten. Bis auf Nankokuvirus und Chemalvirus wurden diese Gattungen auf zwei Unterfamilien aufgeteilt: Gorskivirinae und Skurskavirinae.[4]
Reproduktionszyklus

Genom
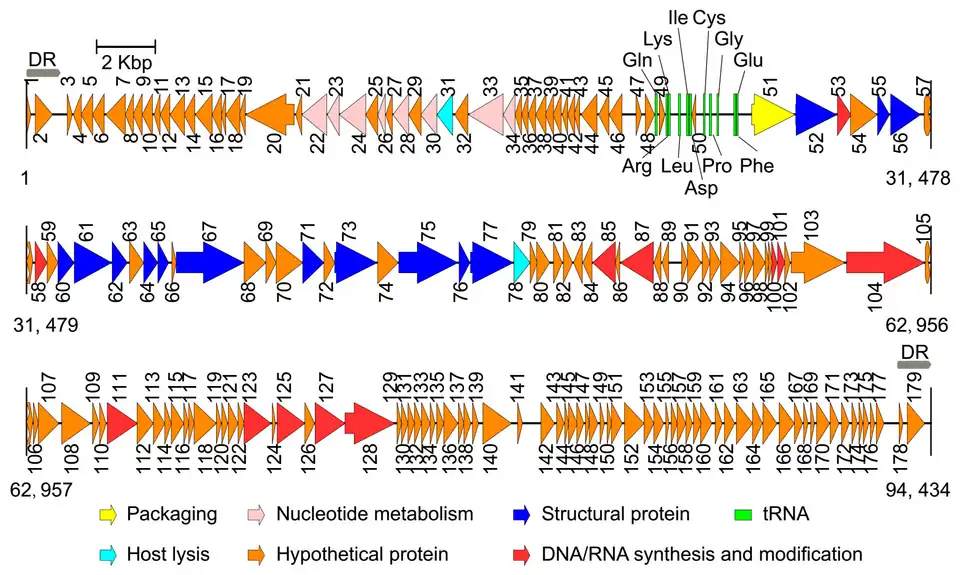
Anwendungen
Das sich unter den Bakterien der Gattung Pseudomonas etliche Erreger von Krankheiten bei Pflanzen und Tieren, insbesondere auch opportunistische Krankheitserreger des Menschen befinden, sind lytische Pseudomonas-Viren von besonderem Interesse, weil sie die Wirtszellen zum Platzen bringen. Unter den zahlreichen Veröffentlichungen zu diesem Thema seien nur einige herausgegriffen:
- Beispielsweise veröffentlichen Laurent Debarbieux et al. 2010 eine Studie, in der sie untersuchten, inwieweit Pseudomonas-Phagen, darunter PAK_P1 (Spezies Pakpunavirus PAKP1), etwa zur Behandlung oder Vorbeugung von Lungeninfektionen mit Pseudomonas aeruginosa tauglich sind.[7]
- Marine Henry et al. untersuchten 2015 in Hinblick auf Phagentherapie einige Pseudomonas-Phagen eingehender, die heute als Mitglieder der Gattungen Nankokuvirus und Pakpunavirus bestätigt sind.[1]
- Eine weitere Studie in dieser Richtung veröffentlichten Anne Chevallereau et al. 2016, unter anderem. über Pseudomonas-Phage PAK_P3 (Gattung Nankokuvirus).[5]
Wenn die Phagen sehr spezifisch ganz bestimmte Wirtsspezies oder gar -stämme infizieren, ist eine genaue Bestimmung des Pathogens Voraussetzung für eine erfolgreiche Phagentherapie. Dafür ist dann sichergestellt, dass die Phagen nicht andere („gutartige“) Bakterien, insbesondere auch von der Gattung Pseudomonas selbst, schädigen.
Systematik
Die Systematik der Vandenendeviridae ist nach International Committee on Taxonomy of Viruses (ICTV), ergänzt um einige Vorschläge nach der Taxonomie des National Center for Biotechnology Information (NCBI) in Hochkommata mit Stand 1. April 2025 (MSL#40v1) wie folgt:[2][8]
Familie Vandenendeviridae
- Gattung Dilongvirus
- Spezies Dilongvirus PHB09, mit Pseudomonas-Phage PHB09
- Gattung Dilongvirus
- Gattung Flaumdravirus[11]
- Spezies Flaumdravirus KIL2 (Pseudomonas-Virus KIL2), mit Pseudomonas-Phage vB_PsyM_KIL4 (sic!) alias Kil4
- Spezies Flaumdravirus KIL4 (Pseudomonas-Virus KIL4), mit Pseudomonas-Phage vB_PsyM_KIL1 (sic!) alias Kil1, vB_PsyM_KIL2 (sic!) alias Kil2, vB_PsyM_KIL3, vB_PsyM_KIL3b und vB_PsyM_KIL5
- Gattung Kremarvirus
- Spezies Kremarvirus kremar, mit Pseudomonas-Phage Kremar
- Spezies Kremarvirus M51, mit Pseudomonas-Phage M5.1
- Spezies Kremarvirus VCM (früher Otagovirus VCM, Pseudomonas-Virus VCM), mit Pseudomonas-Phage VCM alias Pseudomonas syringae DC3000 phage VCM.
- Gattung Omahavirus
- Spezies Omahavirus UNOG1W1, mit Pseudomonas-Phage UNO-G1W1, Wirt: Pseudomonas fluorescens Migula, isoliert aus Süßwassereis in Nebraska
- Gattung Otagovirus (11 Spezies)
- Spezies Otagovirus PE09, mit Pseudomonas-Phage PE09
- Spezies Otagovirus PN09, mit Pseudomonas-Phage PN09
- Spezies Otagovirus psa267, mit Pseudomonas-Phage phiPsa267
- Spezies Otagovirus psa300, mit Pseudomonas-Phage phiPsa300
- Spezies Otagovirus psa315, mit Pseudomonas-Phage phiPsa315
- Spezies Otagovirus psa347, mit Pseudomonas-Phage phiPsa34
- Spezies Otagovirus Psa374, mit Pseudomonas-Phage phiPsa374
- Spezies Otagovirus psa381, mit Pseudomonas-Phage phiPsa381
- Spezies Otagovirus psa397, mit Pseudomonas-Phage phiPsa397
- Spezies Otagovirus psagek4, mit Pseudomonas-Phage psageK4
- Spezies Otagovirus psagek4e, mit Pseudomonas-Phage psageK4e
- Gattung Shenlongvirus
- Spezies Shenlongvirus PPSC2, mit Pseudomonas-Phage PPSC2
- Gattung Tartuvirus
- Spezies Tartuvirus amme3, mit Pseudomonas-Phage vB_PpuM-Amme-3
- Spezies Tartuvirus kopa4, mit Pseudomonas-Phage vB_PpuM-KoPa-4
- Spezies Tartuvirus nopa, mit Pseudomonas-Phage vB_PpuM-NoPa
- Spezies Tartuvirus roomu2, mit Pseudomonas-Phage vB_PpuM-Roomu-2
- Gattung Torinovirus
- Spezies Torinovirus K7A1, mit Pseudomonas-Phage phiK7A1
- Gattung Uavernvirus
- Spezies Uavernvirus uavern, mit Pseudomonas-Phage UAVern alias USAVern
- Gattung Ventosusvirus
- Spezies Ventosusvirus ventosus, mit Pseudomonas-Phage ventosus
- Gattung Weillhallvirus
- Spezies Weillhallvirus wv16Q, mit Pseudomonas-Phage 16Q
- Gattung Yunamivirus
- Spezies Yunamivirus Y1MI, mit Pseudomonas-Phage vB_PF_Y1-MI
- Gattung Flaumdravirus[11]
.png)
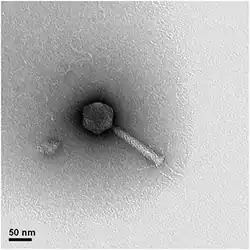
Gattung Pakpunavirus, Balken: 50 nm.[12]
- Gattung Baldwinvirus (im Vorschlag teilweise verschrieben als Balwinvirus[4])
- Spezies Baldwinvirus EM, mit Pseudomonas-Phage EM
- Gattung Pakpunavirus (früher PAK_P1likevirus[1])
- Spezies Pakpunavirus B8, mit Pseudomonas-Phage vB_PaeS_B8
- Spezies Pakpunavirus B31, mit Pseudomonas-Phage vB_PaeM_B31
- Spezies Pakpunavirus B55, mit Pseudomonas-Phage vB_PaeM_B55
- Spezies Pakpunavirus CAb1, mit Pseudomonas-Phage vB_PaeM_C2-10_Ab1
- Spezies Pakpunavirus CAb02, mit Pseudomonas-Phage vB_PaeM_C2-10_Ab02
- Spezies Pakpunavirus EPA1, mit Pseudomonas-Phage vB_PaM_EPA1
- Spezies Pakpunavirus GUMS, mit Pseudomonas-Phage vB_PA45_GUMS
- Spezies Pakpunavirus Henu5, mit Pseudomonas-Phage Henu5
- Spezies Pakpunavirus HJ01, mit Pseudomonas-Phage HJ01
- Spezies Pakpunavirus ITTPL, mit Pseudomonas-Phage ITTPL
- Spezies Pakpunavirus JG004, mit Pseudomonas-Phage JG004
- Spezies Pakpunavirus kat, mit Pseudomonas-Phage vB_Pae_Kat
- Spezies Pakpunavirus LCK69, mit Pseudomonas-Phage vB_PaeM_LCK69
- Spezies Pakpunavirus MAG1, mit Pseudomonas-Phage vB_PaeM_MAG1
- Spezies Pakpunavirus MK (Pseudomonas-Virus phiMK), mit Pseudomonas-Phage phiMK
- Spezies Pakpunavirus PA10, mit Pseudomonas-Phage PA10
- Spezies Pakpunavirus PaGz-1, mit Pseudomonas-Phage PaGz-1
- Spezies Pakpunavirus PAKP1 (Pseudomonas-Virus PAKP1), mit Pseudomonas-Phage PAK_P1 alias vB_PaeM_PAK_P1[7] – Wirt: Pseudomonas aeruginosa PAK, Fundort: Abwasser
- Spezies Pakpunavirus PAKP2 (Pseudomonas-Virus PAKP2), mit Pseudomonas-Phage PAK_P2[1]
- Spezies Pakpunavirus PAKP4 (Pseudomonas-Virus PAKP4), mit Pseudomonas-Phage PAK_P4[1]
- Spezies Pakpunavirus PaP1, mit Pseudomonas-Phage PaP1
- Spezies Pakpunavirus PaZq1, mit Pseudomonas-Phage PaZq-1
- Spezies Pakpunavirus ph0034, mit Pseudomonas-Phage PhL_UNISO_PA-DSM_ph0034
- Spezies Pakpunavirus phipa10, mit Pseudomonas-Phage phipa10
- Spezies Pakpunavirus pPA30992aT2, mit Pseudomonas-Phage pPA-3099-2aT.2
- Spezies Pakpunavirus Ps12, mit Pseudomonas-Phage vB_Paer_Ps12
- Spezies Pakpunavirus PsCh, mit Pseudomonas-Phage vB_Paer_PsCh
- Spezies Pakpunavirus PsIn, mit Pseudomonas-Phage vB_Paer_PsIn
- Spezies Pakpunavirus pv20Sep416, mit Pseudomonas-Phage 20Sep416
- Spezies Pakpunavirus SPA01, mit Pseudomonas-Phage SPA01
- Spezies Pakpunavirus SPA05, mit Pseudomonas-Phage SPA05
- Spezies Pakpunavirus vippaeumc, mit Pseudomonas-Phage vB_VIPPAEUMC01
- Spezies Pakpunavirus YS35, mit Pseudomonas-Phage YS35
- Spezies Pakpunavirus zigelbrucke (Pseudomonas-Virus Zigelbrucke), mit Pseudomonas-Phage Zigelbrucke
- Spezies „Pseudomonas phage K5“ („Pseudomonas-Phage K5“)[15] – zu unterscheiden von Klebsiella-Phage K5 (Przondovirus K5)
- Spezies „Pseudomonas phage K8“ („Pseudomonas-Phage K8“)[16][12]
- Spezies „Pseudomonas phage vB_PaeM_SCUT-S2“ („Pseudomonas-Phage vB_PaeM_SCUT-S2“) (Vorschlag, Wirt: P. aeruginosa )[6]
- Spezies „…“ (weitere Vorschläge)
- ohne zugewiesene Unterfamilie
- Gattung Chemalvirus
- Spezies Chemalvirus PseuGes254, mit Pseudomonas-Phage PseuGes_254 alias vB_PseuGesM_254
- Gattung Nankokuvirus (früher Kpp10virus, KPP10likevirus, KPP10-ähnliche Viren)[17][1]
- Spezies Nankokuvirus Ab03, mit Pseudomonas-Phage vB_PaeM_PAO1_Ab03, sowie vB_PaeM_PAO1_Ab04, vB_PaeM_PAO1_Ab06, vB_PaeM_PAO1_Ab11 und vB_PaeM_PAO1_Ab17
- Spezies Nankokuvirus G1, mit Pseudomonas-Phage vB_PaeM_G1, sowie Epa16, Epa17, Epa24 und Epa26
- Spezies Nankokuvirus KPP10, mit Pseudomonas-Phage KPP10,[7][18][19][20][A. 1] mit Pseudomonas-Phage PAP-JP, sowie PAP-JP und vB_PaeM_VL12
- Spezies Nankokuvirus PAKP3 (Pseudomonas-Virus PAKP3), mit Pseudomonas-Phage PAK_P3, sowie CHA_P1,[1] P3_CHA und PAK_P5[1]
- Spezies Nankokuvirus PS24, mit Pseudomonas-Phage vB_PaeM_PS24
- Spezies „Pseudomonas-Phage LSL4“[1]
- Spezies „…“ (etliche weitere nicht-klassifizierte Vertreter)
- Virionen von Vertretern der Gattung Nankokuvirus:[1]
- Gattung Baldwinvirus (im Vorschlag teilweise verschrieben als Balwinvirus[4])
-
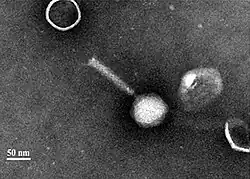
.png) EM-Aufnahme von Pseudomonas-Phage PAK_P5 (Spezies Nankokuvirus PAKP3).
EM-Aufnahme von Pseudomonas-Phage PAK_P5 (Spezies Nankokuvirus PAKP3). -
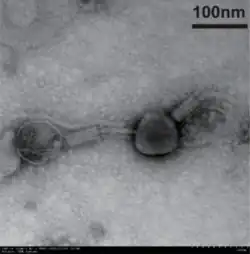
.png) EM-Aufnahme von Pseudomonas-Phage CHA_P1 (Spezies Nankokuvirus PAKP3).
EM-Aufnahme von Pseudomonas-Phage CHA_P1 (Spezies Nankokuvirus PAKP3). -
.png) EM-Aufnahme von Pseudomonas-Phage LSL4 (vorgeschlagen für Gattung Nankokuvirus).
EM-Aufnahme von Pseudomonas-Phage LSL4 (vorgeschlagen für Gattung Nankokuvirus).
Namensherkunft (Etymologie)
Mit dem Familiennamen Vandenendeviridae wird Marinus van den Ende[22] geehrt, ein südafrikanischen Mediziner und Virologe; die Endung ‚-viridae‘ kennzeichnet Virusfamilien.[3][4]
- Die Unterfamilie Gorskivirinae ist benannt zu Ehren des polnischen Immunologen Andrzej Gόrski[23] (geb. 1946 in Wrocław/Breslau, Polen).[24] Er war 2007–2015 Vizepräsident der Polnischen Akademie der Wissenschaften und 1996–1999 Rektor der Medizinischen Akademie in Warszawa/Warschau (jetzt Warschauer Medizinische Universität). Er ist Professor am Institut für Immunologie und experimentelle Therapie, Ludwik Hirszfeld PAS,[24] und war 1999–2007 Direktor dieser Abteilung. Er hat maßgeblich zur Forschung im Bereich der Phagentherapie beigetragen. Mit Stand 2019 gehört er der prestigeträchtige Gruppe der 2 % einflussreichsten Wissenschaftler an (TOP 2 % der zitierten Forscher der Welt! The World's Top 2% Scientists).[10]
- Die Unterfamilie Skurskavirinae ist zu Ehren der polnischen Virologin Zofia Skurska[25][26] (geb. 1909, Warszawa/Warschau, Polen; gest. 2007, Wrocław/Breslau, Polen) benannt. Sie war eine Schülerin des Virologen Ludwik Hirszfeld, dem langjährigen Leiter der Abteilung für Infektionskrankheiten am Institut für Immunologie und experimentelle Therapie an der Polnischen Akademie der Wissenschaften in Breslau und Ausbilderin vieler Generationen von Wissenschaftlern. Im Jahr 1942 wurde sie in den Konzentrationslagern Auschwitz und Ravensbrück inhaftiert. Am bekanntesten wurde sie durch ihre Arbeiten über Interferon. 1952 veröffentlichte sie einige bahnbrechende Studien über Pseudomonas-Phagen.[14]
Der Suffix ‚-virinae‘ kennzeichnet Virusunterfamilien.
Anmerkungen
- ↑ Wirte: P. aeruginosa strain PA20, sowie PA1-3, PA5-10, PA14, PA18, PA20, PA23, PA25, PA26, PA30, PA101-104, S10,[21] und D4[7]
Weblinks
- Virus-Host DB (genome.jp, englisch).
Einzelnachweise
- ↑ a b c d e f g h i j k Marine Henry, Louis-Marie Bobay, Anne Chevallereau, Emilie Saussereau, Pieter-Jan Ceyssens, Laurent Debarbieux: The Search for Therapeutic Bacteriophages Uncovers One New Subfamily and Two New Genera of Pseudomonas-Infecting Myoviridae. In: PLOS One, Band 10, Nr. 1, 28. Januar 2015, S. e0117163; doi:10.1371/journal.pone.0117163, ResearchGate:271597978 (englisch).
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Family: Vandenendeviridae .
- ↑ a b c Liliana Cristina Moraru, Igor Tolstoy, Andrew M. Kropinski: Create a new family, Vandenendeviridae, for a group of lytic Pseudomonas phages (Class: Caudoviricetes). Vorschlag 2024.037B (zip:docx), Filelist. 25. Mai 2024, Stand: 30. September 2024.
- ↑ a b Anne Chevallereau, Bob G. Blasdel, Jeroen De Smet, Marc Monot, Michael Zimmermann, Maria Kogadeeva, Uwe Sauer, Peter Jorth, Marvin Whiteley, Laurent Debarbieux, Rob Lavigne: Next-Generation “-omics” Approaches Reveal a Massive Alteration of Host RNA Metabolism during Bacteriophage Infection of Pseudomonas aeruginosa. In: PLOS Genetics, Band 12, Nr. 7, 5. Juni 2016, S. e1006134; doi:10.1371/journal.pgen.1006134 (englisch).
- ↑ a b Yangyijun Guo, Ping Chen, Zhanglin Lin, Tingting Wang: Characterization of Two Pseudomonas aeruginosa Viruses vB_PaeM_SCUT-S1 and vB_PaeM_SCUT-S2. In: MDPI: Viruses, Band 11, Nr. 4, 1. April 2019, S. 318; doi:10.3390/v11040318 (englisch).
- ↑ a b c d Laurent Debarbieux, Dominique Leduc, Damien Maura, Eric Morello, Alexis Criscuolo, Olivier Grossi, Viviane Balloy, Lhousseine Touqui: Bacteriophages can treat and prevent Pseudomonas aeruginosa lung infections. In: Journal of Infectious Diseases, Band 201, Nr. 7, 1. April 2010, S. 1096–1104; doi:10.1086/651135, JSTOR:27794542, PMID 20196657 (englisch).
- ↑ ICTV: Master Species Lists (MSL).
- ↑ ICTV: Subfamily: Gorskivirinae.
- ↑ a b Dann Turner, Liliana Cristina Moraru, Igor Tolstoy, Evelien M Adriaenssens, Andrew M. Kropinski: To create a new subfamily Gorskivirinae with five genera [Caudoviricetes]. Vorschlag 2023.027B (zip:docx), Filelist. April 2023.
- ↑ a b Sofie Rombouts, Anneleen Volckaert, Sofie Venneman, Bart Declercq, Dieter Vandenheuvel, Camille N. Allonsius, Cinzia Van Malderghem, Ho B. Jang, Yves Briers, Jean P. Noben, Jochen Klumpp, Johan Van Vaerenbergh, Martine Maes, Rob Lavigne: Characterization of Novel Bacteriophages for Biocontrol of Bacterial Blight in Leek Caused by Pseudomonas syringae pv. porri. In: Frontiers in Microbiology, Band 7, Sec. Antimicrobials, Resistance and Chemotherapy, Research Topic: Phage therapy: past, present and future, 15. März 2016; doi:10.3389/fmicb.2016.00279 (englisch).
- ↑ a b
Xuewei Pan, Xiaoli Cui, Fenjiao Zhang, Yang He, Lingyan Li, Hongjiang Yang: Genetic Evidence for O-Specific Antigen as Receptor of Pseudomonas aeruginosa Phage K8 and Its Genomic Analysis. In: Frontiers in Microbiology, Band 7, 2. März 2016; doi:10.3389/fmicb.2016.00252 (englisch). Dazu:
- Potential phage therapy virus massively alters RNA metabolism during infection. Auf: EurekAlert! vom 5. Juli 2016. Mememnto im Webarchiv vom 14. Februar 2021.
- ↑ ICTV: Subfamily: Skurskavirinae.
- ↑ a b Dann Turner, Liliana Cristina Moraru, Igor Tolstoy, Evelien M Adriaenssens, Andrew M. Kropinski: To create a new subfamily Skurskavirinae with two genera (Pakpunavirus and Baldwinvirus) [Caudoviricetes]. Vorschlag 2023.062B (zip:docx), Filelist. Mai 2023.
- ↑ NCBI Taxonomy Browser: Pseudomonas phage K5 (species).
- ↑ NCBI Taxonomy Browser: Pseudomonas phage K8 (species).
- ↑ NCBI Taxonomy Browser: Nankokuvirus , Details: Nankokuvirus, equivalent: Kpp10virus.
- ↑ Jumpei Uchiyama, Mohammad Rashel, Iyo Takemura, Shin-ichiro Kato, Takako Ujihara, Asako Muraoka, Shigenobu Matsuzaki, Masanori Daibata: Genetic characterization of Pseudomonas aeruginosa bacteriophage KPP10. In: Archives of Virology, Band 157, 5. Januar 2012, S. 733–738; doi:10.1007/s00705-011-1210-x, PMID 22218962, ResearchGate:51983667 (englisch).
- ↑ Xu Yang, Anwarul Haque, Shigenobu Matsuzaki, Tetsuya Matsumoto, Shigeki Nakamura: Murine Model of Pseudomonas aeruginosa Pneumonia and Sepsis. In: Frontiers in Microbiology, Band 12, 5, Juli 2021, S. 682255; doi:10.3389/fmicb.2021.682255 (englisch).
- ↑ Dhammika Leshan Wannigama, Cameron Hurst, Peter N. Monk, Paul G. Higgins, Tanittha Chatsuwan, Anthony Kicic, Parichart Hongsing, Shuichi Abe, Stephen M. Stick: Intranasal phage treatment overcomes the limitations posed by antibody-mediated neutralization in bacteriophage therapy for Pseudomonas aeruginosa lung infections. In; ,Journal of Global Antimicrobial Resistance, Band 39, Dezember 2024, S. 71; doi:10.1016/j.jgar.2024.10.229 (englisch).
- ↑ Pseudomonas phage KPP10. Auf: Virus-Host DB.
- ↑ MARINUS VAN DEN ENDE. In: Transactions of the Royal Society of South Africa, Band 35, Nr. 4, OBITUARIES, 1958, S. V-VIII; doi:10.1080/00359195809519017, Epub 22. März 2010 (englisch).
- ↑ Wikidata: Andrzej Górski (Q9152165).
- ↑ a b Prof. dr hab. n. med. Andrzej Górski wśród najbardziej wpływowych naukowców na świecie. Instytut Immunologii i Terapii Doświadczalnej im. Ludwika Hirszfelda Polskiej Akademii Nauk (hirszfeld.pl, polnisch).
- ↑ Wikidata: Zofia Skurska (Q112746424).
- ↑ Zofia Skurska. ScholarGPS (scholargps.com).