Sulfitobacter
| Sulfitobacter | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Sulfitobacter | ||||||||||||
| Sorokin et al 1998 |
Sulfitobacter ist eine Gattung von Meeresbakterien. Arten von Sulfitobacter wurde erstmals im Jahr 1995 beschrieben und sind in verschiedenen marinen Lebensräumen weit verbreitet. Die Gattung zählt zu der Familie Roseobacteraceae.
Merkmale
Arten von Sulfitobacter haben stäbchenförmige Zellen, manchmal an einem Pol spitz zulaufend. Einige teilen sich durch Knospung. Oft werden röschenartige Strukturen (Rosetten) gebildet. Mehrere Arten sind durch Geißeln beweglich.
Stoffwechsel
Arten von Sulfitobacter sind in der Regel obligat aerob, sie benötigen also Sauerstoff. Sie sind obligat chemoorganotroph, sie sind auf organische Stoffe für das Wachstum angewiesen. Energie gewinnen sie durch die Atmung. Allerdings können einige auch die Oxidation von Thiosulfat (S2O32−) und Sulfit (SO32−) zusammen mit einer organischen Kohlenstoffquelle zur Energiegewinnung nutzen.[1]
Eine Untersuchung von verschiedenen Sulfitobacter-Stämmen mit Hilfe der Genomanalyse zeigte, dass sie über den Entner-Doudoroff-Weg, den Pentosephosphatweg, den Citratzyklus und verschiedene Wege für den Aminosäurestoffwechsel verfügen.[2][3]
Ökologie
Sulfitobacter-Arten sind in marinen Lebensräumen weit verbreitet. Sie wurden in Meerwasser, Meeresboden, Meerestieren (wie Fische und Korallen) und Algen sowie Aquakulturumgebungen nachgewiesen. So wurden sie im polaren Meerwasser, im küstennahem Schlamm bei Französisch-Guayana und auch im ölverschmutztem Meerwasser gefunden. Einige Arten leben auch als Epiphyten auf Braunalgen, Kieselalgen, Rotalgen und Dinoflagellaten. Zum Beispiel ist der noch nicht näher bestimmte Stamm Sulfitobacter sp. H24 mit dem Dinoflagellaten Alexandrium fundyense vergesellschaftet.[4][5]
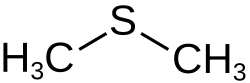
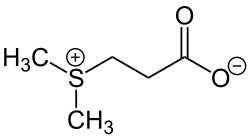
Sulfitobacter-Arten sind am Schwefelkreislauf im Meer beteiligt. Sie wirken hier an der Umwandlung von Dimethylsulfoniumpropionat (DMSP) zu Dimethylsulfid (DMS) mit. Die Verbindung kann ihnen als Schwefel- und Kohlenstoffquelle dienen. DMSP ist eine organische Schwefelverbindung, die in den Ozeanen vorkommt und eine wichtige Rolle im globalen Schwefelkreislauf spielt. Sie wird hauptsächlich von Meeresalgen gebildet. Zu der Umwandlung von DMSP zu DMS sind mehrere Gattungen der Familie Roseobacteraceae in der Lage. Hierzu zählen z. B. Arten von Roseovarius, Silicibacter, Oceanibulbus und Phaeobacter. Dies ist auch einer der Gründe für die enge Verbindung von Arten der Roseobacteraceae mit Algenblüten, da bei Algenblüten große Mengen Dimethylsulfoniopropionat gebildet werden. Das von Bakterien gebildete Dimethylsulfid (DMS) ist wichtig für die Wolkenbildung und somit für das Klima relevant.[6][7]
Sulfitobacter und Algen
Sulfitobacter-Arten vergesellschaften sich häufig mit Algen und können einen Lebensstilwechsel von der Koexistenz zur Pathogenität aufweisen. Dieser Wechsel kann durch von Algen gebildeten Stoffen ausgelöst werden. So kann eine hohe Konzentrationen von DMSP ein pathogenes Verhalten auslösen, während von Algen produziertes Benzoat das Wachstum von Sulfitobacter fördern und den DMSP-induzierten Wechsel zur Pathogenität verhindern kann, wodurch die Koexistenz erhalten bleibt.[8] Zusammen mit dem Stamm Cytophaga spec. MS6 ist Sulfitobacter spec. auch an der Steuerung der Entwicklung der Grünalge Ulva mutabilis beteiligt. Ulva mutabilis entwickelt sich nur dann vollständig, wenn sie mit bestimmten Bakterien in einer symbiotischen Gemeinschaft lebt. Cytophaga und Sulfitobacter scheinen hier die Hauptrolle zu spielen. Cytophaga spec. MS6 scheidet eine phytohormonähnliche Substanz aus, die bei Ulava für die Differenzierung von Stamm- und Rhizoidzellen sowie für die Zellwandbildung wichtig ist. Roseobacter spec. MS2 fördert die Zellteilung der Blätter und die Ausbildung der charakteristischen Blattform. Nur das Zusammenspiel der regulierenden Substanzen beider Bakterien ermöglicht die normale Entwicklung der Alge. Sulfitobacter-Stämme sind auch dafür bekannt, dass sie neben der Wachstumsförderung auch Algen vor anderen, pathogen wirkenden, Bakterien schützen.[9] So schützen Sulfitobacter die Mikroalge Emiliania huxleyi vor dem pathogenen Bakterium Phaeobacter inhibens.
Nutzung
Der mit S. pontiacus nah verwandte Stamm P1-17B ist interessant für die Bekämpfung von Krebszellen.[10] Andere Forschungsvorgänge stehen im Zusammenhang mit dem Abbau von Phthalat durch Sulfitobacter-Arten.[11] Sulfitobacter ist weiterhin für die Produktion von AMP-unabhängiger Sulfitoxidase bekannt, die bei der Herstellung von Sulfit-Biosensoren eine wichtige Rolle spielt. Diese Sulfit-Akzeptor-Oxidoreduktase kann bei der Entwicklung von Biosensorsystemen zum Nachweis von Sulfit in Lebensmitteln und Getränken genutzt werden. Biosensoren zeigen hierbei eine hohe Empfindlichkeit und die Nachfrage nach solchen Biosensorgeräten steigt.[12][13]
Systematik
Sulfitobacter zählt zur Familie der Roseobacteraceae innerhalb der Klasse der Alphaproteobakterien.[14] Sulfitobacter pontiacus ist die Typusart, sie wurde im Jahr 1996 beschrieben. Sie wurde im schwarzen Meer isoliert, aufgrund ihrer Fähigkeit, Schwefelverbindungen zu oxidieren, wurde der Gattungsname Sulfitobacter gewählt, „Sulfito“ bezieht sich auf die Verbindung Sulfit. Die Genome von mehren Sulfitobacter-Stämmen wurden sequenziert. Der Stamm Sulfitobacter NAS-14.1 hat ein Genom von 4,0 Mbp in Größe, das 3.962 kodierende Sequenzen enthält. Sulfitobacter EE-36 hat eine geschätzte Genomgröße von 3,55 Mbp mit 3.474 kodierenden Sequenzen und einem molaren G+C-Gehalt von 60,3 mol%.
Im Mai 2025 waren ungefähr 25 Arten beschrieben, es folgt eine Liste mit einigen Beispielarten:[14]
- Sulfitobacter brevis Labrenz et al. 2000
- Sulfitobacter donghicola Yoon et al. 2007
- Sulfitobacter dubius Ivanova et al. 2004
- Sulfitobacter geojensis Kwak et al. 2014
- Sulfitobacter guttiformis (Labrenz et al. 2000) Yoon et al. 2007
- Sulfitobacter litoralis Park et al. 2007
- Sulfitobacter marinus Yoon et al. 2007
- Sulfitobacter mediterraneus Pukall et al. 1999
- Sulfitobacter noctilucae Kwak et al. 2014
- Sulfitobacter noctilucicola Kwak et al. 2014
- Sulfitobacter pontiacus Sorokin 1996
- Sulfitobacter sabulilitoris Park et al. 2019
- Sulfitobacter salinus Yoon 2020
Einzelnachweise
- ↑ Dimitry Y. Sorokin, Fred A. Rainey, Richard I. Webb und John A. Fuerst: Sulfitobacter In: Bergey's Manual of Systematics of Archaea and Bacteria. 1. Auflage. Wiley, 2015, ISBN 978-1-118-96060-8, doi:10.1002/9781118960608.gbm00874 (wiley.com [abgerufen am 28. Mai 2025]).
- ↑ Han Cui, Shen Fan, Wei Ding, Weipeng Zhang: Genomic Analysis of Novel Sulfitobacter Bacterial Strains Isolated from Marine Biofilms. In: Marine Drugs. Band 22, Nr. 7, 22. Juni 2024, ISSN 1660-3397, S. 289, doi:10.3390/md22070289, PMID 39057398, PMC 11278168 (freier Volltext) – (mdpi.com [abgerufen am 29. Mai 2025]).
- ↑ Xiaokun Xu, Mengdan He, Qingjie Xue, Xiuzhen Li, Ang Liu: Genome-based taxonomic classification of the genus Sulfitobacter along with the proposal of a new genus Parasulfitobacter gen. nov. and exploring the gene clusters associated with sulfur oxidation. In: BMC Genomics. Band 25, Nr. 1, 22. April 2024, ISSN 1471-2164, doi:10.1186/s12864-024-10269-3, PMID 38649849, PMC 11034169 (freier Volltext) – (biomedcentral.com [abgerufen am 31. Mai 2025]).
- ↑ Suresh Jasti, Michael E. Sieracki, Nicole J. Poulton, Michael W. Giewat, Juliette N. Rooney-Varga: Phylogenetic Diversity and Specificity of Bacteria Closely Associated with Alexandrium spp. and Other Phytoplankton. In: Applied and Environmental Microbiology. Band 71, Nr. 7, Juli 2005, ISSN 0099-2240, S. 3483–3494, doi:10.1128/AEM.71.7.3483-3494.2005 (asm.org [abgerufen am 4. August 2025]).
- ↑ María J. Pujalte, Teresa Lucena, María A. Ruvira, David Ruiz Arahal, M. Carmen Macián: The Family Rhodobacteraceae. In: The Prokaryotes. Springer Berlin Heidelberg, Berlin, Heidelberg 2014, ISBN 978-3-642-30196-4, S. 439–512, doi:10.1007/978-3-642-30197-1_377 (springer.com [abgerufen am 7. April 2024]).
- ↑ Yinxin Zeng: Phylogenetic diversity of dimethylsulfoniopropionatedependent demethylase gene dmdA in distantly related bacteria isolated from Arctic and Antarctic marine environments. In: Acta Oceanologica Sinica. Band 38, Nr. 8, August 2019, ISSN 0253-505X, S. 64–71, doi:10.1007/s13131-019-1393-7 (springer.com [abgerufen am 27. Februar 2024]).
- ↑ Irene Wagner-Döbler, Hanno Biebl: Environmental Biology of the Marine Roseobacter Lineage. In: Annual Review of Microbiology. Band 60, Nr. 1, 1. Oktober 2006, ISSN 0066-4227, S. 255–280, doi:10.1146/annurev.micro.60.080805.142115 (annualreviews.org [abgerufen am 27. Februar 2024]).
- ↑ Noa Barak-Gavish, Bareket Dassa, Constanze Kuhlisch, Inbal Nussbaum, Alexander Brandis, Gili Rosenberg, Roi Avraham, Assaf Vardi: Bacterial lifestyle switch in response to algal metabolites. In: eLife. Band 12, 24. Januar 2023, ISSN 2050-084X, doi:10.7554/eLife.84400, PMID 36691727, PMC 9873259 (freier Volltext) – (elifesciences.org [abgerufen am 29. Mai 2025]).
- ↑ Roni Beiralas, Noy Ozer, Einat Segev: Abundant Sulfitobacter marine bacteria protect Emiliania huxleyi algae from pathogenic bacteria. In: ISME Communications. Band 3, Nr. 1, 1. Dezember 2023, ISSN 2730-6151, doi:10.1038/s43705-023-00311-y, PMID 37740057, PMC 10517135 (freier Volltext) – (oup.com [abgerufen am 31. Mai 2025]).
- ↑ Zainab Abdelghani, Nancy Hourani, Zahraa Zaidan, Ghassan Dbaibo, Marguerite Mrad, Rouba Hage-Sleiman: Therapeutic applications and biological activities of bacterial bioactive extracts. In: Archives of Microbiology. Band 203, Nr. 8, Oktober 2021, ISSN 0302-8933, S. 4755–4776, doi:10.1007/s00203-021-02505-1, PMID 34370077, PMC 8349711 (freier Volltext) – (springer.com [abgerufen am 5. August 2025]).
- ↑ Hiroaki Iwaki, Ayaka Nishimura, Yoshie Hasegawa: Isolation and characterization of marine bacteria capable of utilizing phthalate. In: World Journal of Microbiology and Biotechnology. Band 28, Nr. 3, März 2012, ISSN 0959-3993, S. 1321–1325, doi:10.1007/s11274-011-0925-x (springer.com [abgerufen am 5. August 2025]).
- ↑ Yundan Liu, Chengrui Zhu, Yantao Liang, Andrew McMinn, Kaiyang Zheng, Ziyue Wang, Hongmin Wang, Linyi Ren, Hongbing Shao, Yeong Yik Sung, Wen Jye Mok, Li Lian Wong, Min Wang: Genome analysis of vB_SupP_AX, a novel N4-like phage infecting Sulfitobacter. In: International Microbiology. Band 27, Nr. 4, 8. Januar 2024, ISSN 1618-1905, S. 1297–1306, doi:10.1007/s10123-023-00476-5 (springer.com [abgerufen am 5. August 2025]).
- ↑ Kai Muffler, Roland Ulber: Fed‐batch cultivation of the marine bacterium Sulfitobacter pontiacus using immobilized substrate and purification of sulfite oxidase by application of membrane adsorber technology. In: Biotechnology and Bioengineering. Band 99, Nr. 4, März 2008, ISSN 0006-3592, S. 870–875, doi:10.1002/bit.21631 (wiley.com [abgerufen am 5. August 2025]).
- ↑ a b LPSN - Gattung Sulfitobacter
Genutzte Literatur
- Kevin Y. H. Liang, Fabini D. Orata, Yann F. Boucher, Rebecca J. Case: Roseobacters in a Sea of Poly- and Paraphyly: Whole Genome-Based Taxonomy of the Family Rhodobacteraceae and the Proposal for the Split of the “Roseobacter Clade” Into a Novel Family, Roseobacteraceae fam. nov. In: Frontiers in Microbiology. Band 12, 25. Juni 2021, ISSN 1664-302X, doi:10.3389/fmicb.2021.683109, PMID 34248901, PMC 8267831 (freier Volltext) – (frontiersin.org [abgerufen am 3. August 2025]).
- Dimitry Y. Sorokin, Fred A. Rainey, Richard I. Webb und John A. Fuerst: Sulfitobacter In: Bergey's Manual of Systematics of Archaea and Bacteria. 1. Auflage. Wiley, 2015, ISBN 978-1-118-96060-8, doi:10.1002/9781118960608.gbm00874 (wiley.com [abgerufen am 28. Mai 2025]).
Weiterführende Literatur
- Rediat Abate, Yoong-Sin Oon, Yoong-Ling Oon, Yonghong Bi: Microalgae-bacteria nexus for environmental remediation and renewable energy resources: Advances, mechanisms and biotechnological applications. In: Heliyon. Band 10, Nr. 10, Mai 2024, S. e31170, doi:10.1016/j.heliyon.2024.e31170, PMID 38813150, PMC 11133723 (freier Volltext) – (elsevier.com [abgerufen am 5. August 2025]).
- Flora Vincent, Matti Gralka, Guy Schleyer, Daniella Schatz, Miguel Cabrera-Brufau, Constanze Kuhlisch, Andreas Sichert, Silvia Vidal-Melgosa, Kyle Mayers, Noa Barak-Gavish, J. Michel Flores, Marta Masdeu-Navarro, Jorun Karin Egge, Aud Larsen, Jan-Hendrik Hehemann, Celia Marrasé, Rafel Simó, Otto X. Cordero, Assaf Vardi: Viral infection switches the balance between bacterial and eukaryotic recyclers of organic matter during coccolithophore blooms. In: Nature Communications. Band 14, Nr. 1, 31. Januar 2023, ISSN 2041-1723, doi:10.1038/s41467-023-36049-3, PMID 36720878, PMC 9889395 (freier Volltext) – (nature.com [abgerufen am 5. August 2025]).
- Noa Barak-Gavish, Miguel José Frada, Chuan Ku, Peter A. Lee, Giacomo R. DiTullio, Sergey Malitsky, Asaph Aharoni, Stefan J. Green, Ron Rotkopf, Elena Kartvelishvily, Uri Sheyn, Daniella Schatz, Assaf Vardi: Bacterial virulence against an oceanic bloom-forming phytoplankter is mediated by algal DMSP. In: Science Advances. Band 4, Nr. 10, 5. Oktober 2018, ISSN 2375-2548, doi:10.1126/sciadv.aau5716, PMID 30397652, PMC 6200362 (freier Volltext) – (science.org [abgerufen am 5. August 2025]).