Chimalliviridae
| Chimalliviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Schemazeichnung | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Chimalliviridae | ||||||||||||||
| Links | ||||||||||||||
|
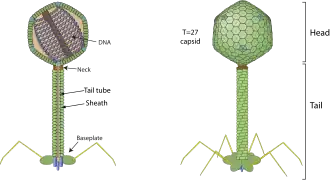
Chimalliviridae ist eine im April 2024 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp A (Myoviren). Ihre Wirte sind Bakterien, was sie als Bakteriophagen klassifiziert.[1][2][3] Charakteristisch ist für diese Viren der sog. Phagenkern.
Beschreibung
Die Entdeckung „kernbildender“ Bakteriophagen (englisch nucleus-forming bacteriophages)[3] hatte Fragen darüber aufgeworfen, wie weit deren charakteristische Replikationsstrategie unter Bakterienviren verbreitet ist und ob diese Phagen phylogenetisch verwandt sind, d. h. eine Klade (Verwandtschaftsgruppe) bilden. Die erste Entdeckung eines solchen Phagenkerns wurde bei Phagen aus der direkten Verwandtschaft von Pseudomonas-Phage phiKZ (Spezies Phikzvirus phiKZ) gemacht! seitdem werden jedes Jahr mehr und mehr Beispiele für kernbildende Phagen beschrieben; Beispiele sind:[4]
- Serratia-Phage PCH45 (nicht klassifizierter Vorschlag)[5]
- Escherichia-Phage vB_EcoM_Goslar (kurz Goslar, Spezies Goslarvirus goslar)
- Erwinia-Phage vB_EamM_RAY (kurz Ray, Spezies Agricanvirus ray)
Phylogenetische Analysen und der Vergleich der Genome dieser Gruppe bestätigten die tatsächliche Verwandtschaft. Zu ihrem Kerngenom (den allen Vertretern dieser Klade gemeinsamen Genen) gehört insbesondere das Hauptkernhüllenprotein (engl. major nuclear shell protein) das namensgebende Chimallin.[6][4]
Jumbo-Phagen und Forschungsgeschichte

Phagen mit einer Genomgröße von über 200 kb[p] (Kilobasen[paaren]) werden umgangssprachlich (d. h. micht-taxonomisch) als „Jumbo-Phagen“ bezeichnet. Jumbo-Phagen sind genetisch vielfältig sind und stellen in ihrer Gesamtheit eindeutig keine Verwandtschaftsgruppe dar.[4][7] Iyer et al. führten 2021 eine Analyse der phylogenetischen Beziehungen der Jumbo-Phagen durch. Als Ergebnis konnten sie drei große Gruppen voneinander abgrenzen, wobei die Jumbo-Phagen-Gruppe 1 aus den PhiKZ-ähnlichen Myoviren besteht, d. h. kernbildende Phagen um Pseudomonas-Phage phiKZ und seine Gattung Phikzvirus — die anderen beiden Gruppen unterteilen sich nochmals in je zwei Untergruppen (2.1, 2.2; 3.1 und 3.2).[7] Es ist also wichtig, Arbeiten über kernbildende Phagen von Arbeiten über kanonisch (herkömmlich) replizierende Phagen mit großem Genom zu unterscheiden. Auf die kernbildenden Jumbo-Phagen der Gruppe 1 hatte sich in den Jahren ab etwa 2020 eine zunehmende Zahl von Arbeiten konzentriert, wobei von einigen Teams vorgeschlagen wurde, dass diese Phagen in einer eigenen Familie zusammenzufassen. Diese Untersuchungen mündeten schließlich im Vorschlag der neuen Familie Chimalliviridae, in der die Klade der kernbildenden Myoviren zusammengefasst wird.[4]
Chimallin
Wie sich zeigte, kodieren diese kernbildenden Phagen ein Haupt-Kernhüllenprotein (englisch major nuclear shell protein), aus dem sich der Phagenkern bildet. Es konnte bei verschiedenen Phagen gezeigt werden, dass es sich während des Replikationszyklus zu ähnlichen Gitterstrukturen zusammenfügt (assembliert), die das Phagengenom umgeben. Dieses Protein wird als Chimallin (ChmA) bezeichnet. Anders als beim Tubulin PhuZ und mehrgliedrigen RNA-Polymerasen, die unter nicht miteinander verwandten Phagen weit verbreitet sind, zeigten eingehende Analysen (mit VICTOR und ViPTree), dass mit Phagen mit Chimallin-Homologen zusammengehören und unter der Gesamtheit aller Phagen eine monophyletische Klade bilden.[4]
Replikationsstrategie
Erwinia-Phage vB_EamM_RAY (kurz Ray, Spezies Agricanvirus ray) weicht genetisch von den gut untersuchten PhiKZ-ähnlichen Viren ab. Gerade deshalb wurde dieser Phage ausgewählt, um Homologe jedes Gens in den Phagengenomen zu untersuchen. Mit Hilfe von PSI-BLAST konnte gezeigt werden, dass 72 Gene von Phage RAY in 90 % der Phagen mit derselben Replikationsstrategie konserviert sind, woraus sich ein gemeinsames Kerngenom ableiten ließ. Dieses Kerngenom besteht aus 68 untereinander nicht-homologen Genen, die in sieben verschiedenen Blöcken in den Genomen der analysierten Phagen zu finden sind. Diese Blöcke werden in den Genomen der fraglichen Phagen in der gleichen Reihenfolge kodiert — was heißt, das die Mitglieder dieser Phagengruppe eine konservierte Genomorganisation aufweisen. Von diesen Genen kommen 21 nur in den Chimallin-kodierenden Phagen vor. Es kann daher vermutet werden, dass diese Gene für die Replikation unter Bildung eines Phagenkerns wichtig sind (auch wenn es aufgrund der unbekannten Funktionen dieser Gene derzeit — Stand Juni 2023 — noch unmöglich ist, mit Sicherheit vorherzusagen, ob alle Mitglieder dieser Gruppe während ihres Replikationszyklus tatsächlich einen Phagenkern bilden). Die Existenz dieser gemeinsamen Kerngene lieferte ein weiteres Argument dafür, dass die betrachteten Phagen eine gemeinsame Virusfamilie bilden.[4]
Als Kennzeichen der von ihnen zuvor definierten Jumbo-Phagen-Gruppe 1 machten Iyer et al. (2021) eine mehrgliedrige „Doppeltrommel“-RNA-Polymerase (double-barrel RNAP), eine ungewöhnliche DNA-Polymerase aus einer speziellen Gruppe der Familie B, eine spezielle Version der DnaB-Helikase und eine Hauptkapsidprotein (MCP) vom PhiKZ-Typ aus.[7]
Systematik
Die im März 2025 aufgestellten Familie Chimalliviridae umfasst als Gründungsmitglieder ausschließlich Phagen, die die zu diesem Zeitpunkt bereits Gattungen zugeordnet waren. Insgesamt wurden 19 bestehende Gattungen der Klasse Caudoviricetes ohne bisherige Familienzuordnung mit ihren Mitgliedern in die neue Familie übergeführt. Die Gattungen Aphroditevirus und Tidunavirus bilden dabei die Unterfamilie Gorgonvirinae, die als Ganzes ebenfalls in die neue Familie übergeführt wurde.[4]
Bacillus-Phage PBS1 (Spezies Takahashivirus PBS1) weist eine gewisse genetische Ähnlichkeit mit den Chimallin-kodierenden Phagen auf, hat aber weder deren konservierte Genomorganisation noch das für diese identifizierte Kerngenom. Die Gattung Takahashivirus wurde daher nicht als Mitglied der Familie zugeordnet, PBS1 bzw. Takahashivirus kann stattdessen in phylogenetischen Bäumen der Chimalliviridae als Außengruppe dienen. In diesen zeigte sich, dass die Verzweigungslänge der Chimalliviridae ähnlich groß ist wie bei den benachbarten Familien Autographiviridae und Schitoviridae.[4]
Die Systematik der Chimalliviridae ist nach International Committee on Taxonomy of Viruses (ICTV), ergänzt um einige Vorschläge nach der Taxonomie des National Center for Biotechnology Information (NCBI) in Hochkommata mit Stand 1. April 2025 (MSL#40v1) wie folgt:[1][8]
Familie Chimalliviridae (offenbar synonym zur früheren Bezeichnung Jumbo-Phagen-Gruppe 1[7])
- Unterfamilie Gorgonvirinae
- Gattung Aphroditevirus
- Spezies Aphroditevirus aphrodite1 (Vibrio-Virus Aphrodite1), mit Vibrio-Phage Aphrodite1
- Spezies Aphroditevirus av2TSL2019, mit Vibrio-Phage 2 TSL-2019
- Spezies Aphroditevirus PDCC1, mit Photobacterium-Phage PDCC-1
- Spezies Aphroditevirus USC1, mit Vibrio-Phage USC-1
- Gattung Tidunavirus
- Spezies Tidunavirus pTD1 (Vibrio-Virus pTD1), mit Vibrio-Phage pTD1
- Spezies Tidunavirus VP4B (Vibrio-Virus VP4B), mit Vibrio-Phage VP4B
- Gattung Aphroditevirus
- ohne zugewiesene Unterfamilie
- Gattung Agricanvirus (früher Agrican357virus, 6 Spezies)
- Spezies Agricanvirus deimos (Erwinia-Virus Deimos), mit Erwinia-Phage vB_EamM_Deimos-Minion
- Spezies Agricanvirus desertfox (Erwinia-Virus Desertfox), mit Erwinia-Phage vB_EamM_Desertfox
- Spezies Agricanvirus Ea3570 (Erwinia-Virus Ea35-70), mit Erwinia-Phage Ea35-70
- Spezies Agricanvirus ray (Erwinia-Virus RAY), mit Erwinia-Phage vB_EamM_RAY alias RAY,[3] Erwinia-Phage Rebecca, Erwinia-Phage vB_EamM_Bosolaphorus, Erwinia-Phage vB_EamM_Mortimer
- Spezies Agricanvirus simmy50 (Erwinia-Virus Simmy50), Erwinia-Phage vB_EamM_Simmy50
- Spezies Agricanvirus specialG (Erwinia-Virus SpecialG), Erwinia-Phage vB_EamM_Special G
- Spezies „Erwinia phage AH06“ („Erwinia-Phage AH06“)
- Gattung Branisovskavirus
- Spezies Branisovskavirus Kc263, mit Kosakonia-Phage Kc263
- Gattung Chaoshanvirus
- Spezies Chaoshanvirus ZPAH34, mit Aeromonas-Phage ZPAH34
- Gattung Chiangmaivirus (früher Rsl2virus)
- Spezies Chiangmaivirus RSF1, mit Ralstonia-Phage RSF1
- Spezies Chiangmaivirus RSL2, mit Ralstonia-Phage RSL2
- Gattung Derbicusvirus
- Spezies Derbicusvirus derbicus, mit Erwinia-Phage Derbicus
- Gattung Elvirus (früher Ellikevirus)
- Spezies Elvirus EL (Pseudomonas-Virus EL), mit Pseudomonas-Phage EL (phiEL)[9][10] und Pseudomonas-Phage ELvir (phiELvir), einer virulenten Mutante[9] – früher zu Phikzvirus
- Gattung Eowynvirus
- Spezies Eowynvirus eowyn, mit Klebsiella-Phage vB_KvM-Eowyn
- Gattung Erskinevirus (früher Eah2virus)
- Spezies Erskinevirus asesino, mit Erwinia-Phage vB_EamM_Asesino
- Spezies Erskinevirus EaH2, mit Erwinia-Phage PhiEaH2
- Gattung Ferozepurvirus
- Spezies Ferozepurvirus pAEv1810, mit Aeromonas-Phage pAEv1810
- Spezies Ferozepurvirus PS1, mit Aeromonas-Phage PS1
- Goslarvirus
- Spezies Goslarvirus goslar (Escherichia-Virus Goslar), mit Escherichia-Phage vB_EcoM_Goslar alias Goslar
- Gattung Iapetusvirus
- Spezies Iapetusvirus EaH1 (Erwinia-Virus EaH1), mit Erwinia-Phage PhiEaH1
- Gattung Ludhianavirus (Aeromonas-Phagen, 3 Spezies)
- Spezies Ludhianavirus D3, mit Aeromonas-Phage D3
- Spezies Ludhianavirus D6, mit Aeromonas-Phage D6
- Spezies Ludhianavirus LAh10, mit Aeromonas-Phage LAh10
- Gattung Maaswegvirus
- Spezies Maaswegvirus Kp24, mit Klebsiella-Phage vB_KpM_FBKp24
- Gattung Agricanvirus (früher Agrican357virus, 6 Spezies)
.png)

.png)
- Gattung Machinavirus
- Gattung Meadowvirus
- Spezies Meadowvirus AH04, mit Erwinia-Phage AH04
- Gattung Miamivirus
- Spezies Miamivirus miami, mit Klebsiella-Phage Miami
- Gattung Miltoncavirus abgetrennt von Phikzvirus
- Spezies Miltoncavirus PhiPA3 (Pseudomonas-Virus PhiPA3), mit Pseudomonas-Phage PhiPA3 (φPA3) – früher zu Phikzvirus[14]
- Gattung Moabitevirus
- Spezies Moabitevirus moabite (Serratia-Virus Moabite)
- Spezies Moabitevirus mv2050HW (Serratia-Virus 2050HW)
- Gattung Nimduovirus
- Spezies Nimduovirus N1M2, mit Klebsiella-Phage N1M2
- Gattung Noxifervirus
- Spezies Noxifervirus noxifer (Pseudomonas-Virus Noxifer), mit Pseudomonas-Phage Noxifer
- Gattung Pawinskivirus (abgetrennt von Phikzvirus)
- Spezies Pawinskivirus PS119XW, mit Pseudomonas-Phage vB_PaeM_PS119XW – früher zu Phikzvirus[14]
- Gattung Petsuvirus
- Spezies Petsuvirus pEtSU (Edwardsiella-Virus pEtSU), mit Edwardsiella-Phage pEt-SU
- Gattung Phabiovirus
- Spezies Phabiovirus phabio (Pseudomonas-Virus Phabio), mit Pseudomonas-Phage PhabioPhabio[14]
- Gattung Phikzvirus (früher Phikzlikevirus, PhiKZ-like viruses, PhiKZ-ähnliche Viren)[15]
- Gattung Ripduovirus
- Spezies Ripduovirus RP12 (Ralstonia-Virus RP12), mit Ralstonia-Phage RP12
- Gattung Risingsunvirus
- Spezies Risingsunvirus risingsun (Erwinia-Virus Risingsun), mit Erwinia-Phage vB_EamM_RisingSun
- Gattung Seoulvirus (früher Spn3virus), zu unterscheiden vom Seoul-Virus (Spezies Seoul orthohantavirus, Gattung Orthohantavirus)
- Gattung Serwervirus
- Spezies Serwervirus 201phi21 (Pseudomonas-Virus 201phi21), mit Pseudomonas-Phage 201phi2-1 alias Pseudomonas chlororaphis phage 201phi2-1 (201φ2-1)[16][17][18] – früher zu Phikzvirus[14]
- Gattung Siatvirus
- Spezies Siatvirus Lz245, mit Klebsiella-Phage KpLz-2_45
- Gattung Tepukevirus (abgetrennt von Phikzvirus)
- Spezies Tepukevirus Psa21 (Pseudomonas-Virus Psa21), mit Pseudomonas-Phage Psa21 – früher zu Phikzvirus[14]
- Gattung Wellingtonvirus
- Spezies Wellingtonvirus wellington (Erwinia-Virus Wellington), mit Erwinia-Phage Wellington
Etymologie
.jpg)
Die Bezeichnung der Familie Chimalliviridae verweist auf Chimallin, wichtigsten Kernhüllenprotein dieser Phagen, und bezieht sich damit auf den charakteristischen Replikationsmechanismus der Mitglieder der Familie. Dessen Name leitet sich ab von Nahuatl chīmalli (IPA ), dem traditionellen Schild der indigenen Ethnien Mexikos, insbes. der Azteken. Der Suffix ‚-viridae‘ kennzeichnet Virusfamilien.[2][4]
Die Bezeichnung der Unterfamilie Gorgonvirinae verweist auf die Gorgonen, Schreckgestalten der griechischen Mythologie; der Suffix ‚-virinae‘ kennzeichnet Virusunterfamilien.[19]
Einzelnachweise
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Order: Chimalliviridae .
- ↑ a b c Amy Prichard, Jina Lee, Thomas G. Laughlin, Amber Lee, Kyle P. Thomas, Annika E. Sy, Tara Spencer, Aileen Asavavimol, Allison Cafferata, Mia Cameron, Nicholas Chiu, Demyan Davydov, Isha Desai, Gabriel Diaz, Melissa Guereca, Kiley Hearst, Leyi Huang, Emily Jacobs, Annika Johnson, Samuel Kahn, Ryan Koch, Adamari Martinez, Meliné Norquist, Tyler Pau, Gino Prasad, Katrina Saam, Milan Sandhu, Angel Jose Sarabia, Siena Schumaker, Aaron Sonin, Ariya Uyeno, Alison Zhao, Kevin D. Corbett, Kit Pogliano, Justin Meyer, Julianne H. Grose, Elizabeth Villa1, Rachel Dutton, Joe Pogliano: Identifying the core genome of the nucleus-forming bacteriophage family and characterization of Erwinia phage RAY. In: Cell Reports, Band 42, Nr. 5, 30. Mai 2023, S. 112432; doi:10.1016/j.celrep.2023.112432, PMC 10299810 (freier Volltext), PMID 37120812 (englisch).
- ↑ a b c d e f g h i A. Prichard, J. I. Lee, Joe Pogliano: Create one new family (Chimalliviridae) in the class Caudoviricetes. Vorschlag 2023.015B (zip:docx). 4. Juni 2023.
- ↑ NCBI Taxonomy Browser: Serratia phage PCH45.
- ↑ Protein: Chimallin. Organism: Pseudomonas phage phiKZ. Auf UniProt.
- ↑ a b c d Lakshminarayan M. Iyer, Vivek Anantharaman, Arunkumar Krishnan, A. Maxwell Burroughs, L. Aravind: Jumbo Phages: A Comparative Genomic Overview of Core Functions and Adaptions for Biological Conflicts. In: MDPI: Viruses, Band 13, Nr. 1, Special Issue Giant or Jumbo Phages, 5. Januar 2021, S. 63; doi:10.3390/v13010063 (englisch). Siehe insbes. auch Supplement (Zip mit PDF und xlsx).
- ↑ ICTV: Master Species Lists (MSL).
- ↑ a b Victor Krylov, Maria Bourkaltseva, Elena Pleteneva, Olga Shaburova, Sergey Krylov, Alexander Karaulov, Sergey Zhavoronok, Oxana Svitich, Vitaly Zverev: Phage phiKZ—The First of Giants. In: Viruses. Band 13, Nr. 2. MDPI, 20. Januar 2021, 149, doi:10.3390/v13020149 (englisch).
- ↑ a b Victor Krylov, Olga Shaburova, Sergey Krylov, Elena Pleteneva: A Genetic Approach to the Development of New Therapeutic Phages to Fight Pseudomonas Aeruginosa in Wound Infections. In: MDPI Viruses, Band 5, Nr. 1, 21. Dezember 2012, Special Issue Recent Progress in Bacteriophage Research, S. 15–53; doi:10.3390/v5010015 (englisch).
- ↑ a b Jun Kwon et al.: Isolation and Characterization of Salmonella Jumbo-Phage pSal-SNUABM-04. In: Viruses, Band 13, Nr. 1, Giant or Jumbo Phages, 27, 25. Dezember 2020; doi:10.3390/v13010027 (englisch).
- ↑ a b c J. Bernard Heymann et al.: The Mottled Capsid of the Salmonella Giant Phage SPN3US, a Likely Maturation Intermediate with a Novel Internal Shell. In: Viruses, Band 12, Nr. 9, 19. August 2020, S. 910; doi:10.3390/v12090910 (englisch).
- ↑ Salmonella phage pSal-SNUABM-04 (species). NCBI
- ↑ a b c d e A. Millard, M. Łobocka, Igor Tolstoy, Dann Turner, Liliana Cristina Moraru, Andrew M. Kropinski: Create five new genera of Pseudomonas jumbo phages (Caudoviricetes). Vorschlag: 2022.066B (zip:docx). Dazu: Change one genus name that already existed. (2022).
- ↑ A. Cornelissen, S. C. Hardies, O. V. Shaburova, V. N. Krylov, W. Mattheus, A. M. Kropinski, R. Lavigne: Complete Genome Sequence of the Giant Virus OBP and Comparative Genome Analysis of the Diverse KZ-Related Phages. In: Journal of Virology. 86. Jahrgang, Nr. 3, 2011, S. 1844–1852, doi:10.1128/JVI.06330-11, PMID 22130535, PMC 3264338 (freier Volltext) – (englisch).
- ↑ Pseudomonas phage 201phi2-1, auf: Virus-Host DB, TAX:198110
- ↑ Proteomes - Pseudomonas phage 201phi2-1, auf: UniProt
- ↑ Pseudomonas phage 201phi2-1 (species). NCBI
- ↑ Evelian M. Adriaenssens, Igor Tolstoy, Andrew M. Kropinski, Liliana Cristina Moraru: Create one new subfamily (Gorgonvirinae) including two genera (Caudovirales: Myoviridae). Vorschlag 2020.065B. Juli 2020.