Asgard-Archaeen
| Asgard-Archaeen | ||||||||
|---|---|---|---|---|---|---|---|---|

Promethearchaeum syntrophicum, künstlerische Nachbildung in Plastilin. | ||||||||
| Systematik | ||||||||
| ||||||||
| Wissenschaftlicher Name | ||||||||
| Promethearchaeati | ||||||||
| Imachi et al. 2024 |
Das Reich (englisch kingdom, lateinisch regnum) der Asgard-Archaeen (auch Asgard-Gruppe, wissenschaftlich Promethearchaeati, ursprünglich auch als Phylum „Asgardarchaeota“ vorgeschlagen[1][2]) ist ein Taxon von Archaeen,[3][4] das derzeit monotypisch offiziell nur aus dem einzigen Phylum Promethearchaeota besteht.[3] Zu dessen Mitgliedern gehören insbesondere die Klasse Promethearchaeia (mit Promethearchaeum syntrophicum) und die Kandidaten-Klassen „Candidatus Asgardarchaeia“ (mit „Ca. Asgardarchaeum abyssi“), „Ca. Heimdallarchaeia“ (mit „Ca. Heimdallarchaeum aukensis“ und „Ca. H. endolithica“), „Ca. Odinarchaeia“ (mit „Ca. Odinarchaeum yellowstonii“), „Ca. Sifarchaeia“ (mit „Ca. Sifarchaeum marinarchaeum“ und „Ca. S. subterraneum“).
Zu den Asgard-Archaeen gehören per Vorschlag (inoffiziell) weitere Phyla (und kleinere Gruppen), darunter insbesondere „Ca. Thorarchaeota“.[5][6][7]
Die ursprünglichen Hinweise stammen alle nur aus Metagenom-Daten; inzwischen wurde aber erste Vertreter der Gruppe kultiviert (Promethearchaeum syntrophicum).[8]
Unter den Asgard-Archaeen befinden sich die nächsten prokaryotischen Verwandten der Eukaryoten,[9] die möglicherweise aus einer Vorfahrenlinie der Asgardarchaeota hervorgegangen sind, nachdem sie Bakterien durch den Prozess der Symbiogenese zu Mitochondrien oder mitochondrien-ähnliche Organellen (mitochondria-like organelles, MROs) assimiliert haben.[9][10]
Forschungsgeschichte
Im Sommer 2010 wurden Sedimente aus einem Bohrkern analysiert, der im Rifttal auf dem Knipowitsch-Rücken[A. 1] zwischen Grönland und Spitzbergen im Arktischen Ozean entnommen wurde. Der Entnahmeort war in der Nähe des Hydrothermalschlots namens Lokis Schloss[11] (Loki's Castle 73,55° N, 8,15° O), einem sog. „Schwarzen Raucher“. Weil vorherige Untersuchungen auf neuartige Archaeen-Linien hingedeutet hatten, wurden die Proben einer Metagenomanalyse unterzogen, die diese Vermutung bestätigten.[12][13]
Nach dem positiven Ergebnis der ersten Analysen wurden die Proben von einem Team unter Führung der Universität Uppsala einer phylogenetischen Analyse unterzogen, die eine Anzahl von hochkonservierter Protein-kodierender Gene zum Gegenstand hatte. Als Ergebnis schlug das Team im Jahr 2015 das neue Archaeenphylum „Lokiarchaeota“ für die aus der Metagenomik identifizierten Gensequenzen (Contigs) vor.[14]
Der Name ist ein Verweis auf den Schwarzen Raucher, von dem die erste Metagenomprobe stammte, und bezieht sich auf Loki, eine der vielschichtigsten und wandlungsfähigsten Gestalten des nordischen Pantheons.[15] Der mythologische Loki wurde beschrieben als „eine atemberaubend komplexe, verwirrende und ambivalente Figur, die Ursache unzähliger ungelöster wissenschaftlicher Kontroversen war“,[16] ganz analog zur Rolle der „Lokiarchaeota“ in den Debatten über den Ursprung der Eukaryoten.[14][17]
Im Jahr 2016 entdeckte ein anderes Team unter Leitung der University of Texas in Proben aus Sedimenten im Mündungsgebiet (Ästuarsedimenten) des White Oak River (34,8835° N, 77,2216° W) in North Carolina eine weitere, verwandte Gruppe von Archaeen, die „Thorarchaeota“ benannt wurde nach Thor, einem weiteren nordischen Gott.[18]
Weitere Proben von Lokis Schloss, dem Yellowstone-Nationalpark, der Aarhus-Bucht, einem Grundwasserleiter (Aquifer) in der Nähe des Colorado River, dem Radiata Pool in Neuseeland (Ngatamariki, bei der Stadt Taupō und dem gleichnamigen Supervulkan, Nordinsel),[19][20] Hydrothermalquellen in der Nähe der Taketomi-Insel, Japan, und der Mündung des White Oak River in den Vereinigten Staaten führten dazu, dass weitere verwandte Gruppen entdeckt wurden, „Ca. Odinarchaeota“ (heute „Ca. Odinarchaeia“) und „Ca. Heimdallarchaeota“ (heute „Ca. Heimdallarchaeia“),[6] und entsprechend der (unverbindlichen) Namenskonvention nach Odin bzw. Heimdall benannt wurden. Das Superphylum, das diese Mikroben enthält, bekam dann konsequenterweise den (provisorischen) Namen „Asgard“, nach dem Wohnort der Götter in der nordischen Mythologie.[6]
Eine vergleichende Analyse von 162 Asgard-Genomen durch Liu et al. erweiterte im Jahr 2021 die phylogenetische Vielfalt der Asgard-Archaeen erheblich und führte zum Vorschlag von sechs zusätzlichen Phyla, einschließlich einer basalen Klade, die vorläufig „Ca. Wukongarchaeota“ genannt wurde (heute „Ca. Wukongarchaeia“). In mehreren dieser Phyla wurden weitere Homologe von Proteinen entdeckt, die für Eukaryoten charakteristisch sind. Die deutet auf eine dynamische Evolution durch horizontalen Gentransfer (HGT), Genverlust und -verdopplung und sogar Cross-Domain-Shuffling hin. Die Studie erlaubte jedoch noch nicht, zwischen den beiden möglichen Positionen des letzten gemeinsamen Vorfahren der Eukaryoten (LECA) zu entscheiden: entweder einer Schwesterklade der Heimdallarchaeia-Wukongarchaeia-Klade innerhalb der Asgard-Archaeen (wie im Kladogramm unten) oder einer Schwesterklade derer selbst (innerhalb der Archaeen).[7]
Im Jahr 2023 kamen Laura Eme, Daniel Tamarit et al. aufgrund weiterer derartiger Analysen zum Schluss, dass die Eukaryoten ihren Ursprung tief in der Gruppe der Asgard-Archaeen haben. Sie identifizierten als Schwestergruppe der Eukaryota dabei die Ordnung „Ca. Hodarchaeales“ innerhalb der von ihnen als Kandidatenklasse angenommenen „Heimdallarchaeia“.[21]
Im Jahr 2024 wurden in den beiden Prokaryoten-Domänen der Bakterien und Archaeen erstmals offiziell Taxa im Rang eines Reichs (englisch kingdom, lateinisch regnum) eingeführt. Die Asgard-Archaeen wurden dabei mit der Bezeichnung Promethearchaeati (nach dem ersten kultivierten und gültig veröffentlichten Vertreter, Promethearchaeum syntrophicum) offiziell bestätigt.[3]
Beschreibung
Die Asgard-Mitglieder kodieren eine Vielzahl eukaryotischer Signaturproteine (ESPs),[22] darunter neuartige GTPasen, membranumbauende Proteine (en. membrane-remodelling proteins), wie ESCRT[23] und SNF7,[24][25] ein Ubiquitin-Modifizierungssystem und N-Glykosylierungspfad-Homologe.[6]
Asgard-Archaeen haben ein reguliertes Aktin-Zytoskelett, und die von ihnen verwendeten Profiline und Gelsoline können mit eukaryotischen Aktinen interagieren.[26][27][28] Sie scheinen auch Vesikel zu bilden, wie unter dem Kryoelektronenmikroskop (Kryo-EM) zu erkennen ist. Einige scheinen S-Layer-Proteine mit einer PKD-Domäne (en. polycystic kidney disease domain) zu haben.[8] Außerdem haben sie wie Eukaryoten in der größten Untereinheit der ribosomalen RNA, der LSU-rRNA (large subunit of ribosomal RNA), eine dreifache Erweiterung ES39 (expansion segment 39).[29][30]
Die Vielfalt an CRISPR-Cas-verwandten Systemen ist ein spezielles Merkmal der Asgard-Archaeen, das bei Eukaryoten nicht vorkommt.[31]
Mesophil sind folgende Untergruppen:[32]
Hyperthermophil sind dagegen:[32]
- Promethearchaeota: Freyarchaeia, Baldrarchaeia
- Idunnarchaeota
Stoffwechsel
-
![Stoffwechselwege der Asgard-Archaeen für einige der Phyla[33]](./Asgard_archaea_Phyla.png) Stoffwechselwege der Asgard-Archaeen für einige der Phyla[33]
Stoffwechselwege der Asgard-Archaeen für einige der Phyla[33] -
![Stoffwechselwege von Asgard-Archaeen, je nach Umgebung[33]](./Asgard_archaea_in_various_environments.png) Stoffwechselwege von Asgard-Archaeen, je nach Umgebung[33]
Stoffwechselwege von Asgard-Archaeen, je nach Umgebung[33]
Asgard-Archaeen sind obligate Anaerobier. Sie haben einen Wood-Ljungdahl-Weg und führen Glykolyse durch. Die Mitglieder dieser Gruppe können autotroph, heterotroph oder phototroph mit Heliorhodopsin sein.[33] Ein Mitglied, Candidatus Promethearchaeum syntrophicum, führt Syntrophie mit einem schwefelreduzierenden Proteobakterium und einem methanogenen Archaeon durch.[8]
Ihre RuBisCO-Versionen sind nicht kohlenstofffixierend, sondern werden wahrscheinlich für das Nukleosid-Salvage (en. nucleoside salvaging) verwendet.[33]
Ähnlichkeiten mit Eukaryoten in Untergruppen
Im Jahr 2017 wurde entdeckt, dass die Vertreter des vorgeschlagenen Phylums Heimdallarchaeota N-terminale Core-Histon-Arme (corehistone tails) haben, ein Merkmal, von dem man zuvor annahm, dass es ausschließlich bei Eukaryoten vorkommt. Bei zwei weiteren Archaeen-Phyla, die nicht Asgard angehören, wurde 2018 ebenfalls dieses Merkmal gefunden, und zwar in „Huberarchaeota“ (DPANN) und „Bathyarchaeota“ (beide im „TACK-Superphylum“, offiziell Reich Thermoproteati).[34]
Im Januar 2020 fanden Wissenschaftler die Archaeenspezies Promethearchaeum syntrophicum, ein Mitglied der Promethearchaeota (früher „Lokiarchaeota“), das eine Syntrophie mit zwei Bakterienarten eingeht. Dieser Befund zeigt, dass Asgard-Archaeen zu komplexen Syntrophien in der Lage sind, eine Voraussetzung für die von der Eozyten-Hypothese behauptete Entwicklung von den Archaeen hin zu komplexen eukaryotischen Mikroorganismen, wie sie vor etwa zwei Milliarden Jahren durch Symbiogenese entstanden sind.[35][8][A. 2]
Bedeutung in der Evolution
Versklavung des Cosymbionten

Promethearchaeum verbindet sich unter sauerstoffarmen (anaeroben) Bedingungen mit einem Alphaproteobakterium (beispielsweise Halodesulfovibrio), wobei die von Promethearchaeum durch Hydrolyse von Aminosäuren und Peptiden freigesetzten H+-Ionen (Protonen oder „Protien“) vom Alphaproteobakterium zur Synthese energiereicher Verbindungen genutzt werden, die dann wieder vom Asgard-Archaeon genutzt werden. Dabei entsteht aus dem H+-Ion (= „Protium“) von Promethearchaeum und dem Hydrogensulfid HS− von Halodesulfovibrio Schwefelwasserstoff H2S.
Ähnlich muss dann unter sauerstoffreicheren (aeroberen) Bedingungen ein Asgard-Archaeon Protonen an ein Alphaproteobakterium abgegeben haben, wobei aus dem Proton H+ des Asgard-Archaeons und dem Hydrogenoxid HO− (= Hydroxidion) des Alphaproteobakteriums Wasser H2O entstand, ähnlich wie dies auch heutzutage noch zwischen Zellplasma und Mitochondrien, genauer gesagt zwischen Intermembranraum und Innenraum der Mitochondrien, geschieht.
Nach der Umschling, Umhüll, Versklav Hypothese (engl. Entangle, Engulf, Enslave Hypothesis E3) nahmen noch nicht identifizierte Asgard-Archaeen
- zunächst Kontakt mit dem Symbionten auf,
- umschlangen ihn, um den Kontakt zu sichern,
- umhüllten ihn dann, um die Kontaktfläche zu vergrößern,
- verschlangen ihn anschließend, um ihn zu versklaven und
- sich zuletzt seine Gene allmählich einzuverleiben, sprich vom Symbionten ins Zellplasma zu transportieren.
Auf diese Weise entstanden so allmählich die Mitochondrien, die der Zelle als Kraftwerke dienen. Einige Proteine der Mitochondrien werden auch heute noch auf mitochondrialen Plasmiden mtDNA gespeichert und von mitochondrialen Ribosomen im hergestellt.
Als die Nachfahren dieser Archaeen dann zu einem Zellkern kamen, wurden diese Gene dann in den Zellkern transportiert. Archaeen selber besitzen keinen Zellkern.
Nach heutigem Stand sind die nächsten Vorfahren des Zellplasmas der Kernzeller die Heimdallarchaeen und die nächsten Vorfahren der Mitochondrien der Kernzeller die Alphaproteobakterien. Die Versklavung der Alphaproteobakterien durch Heimdallarchaeen ereignete sich vor rund 2,4 Milliarden Jahren im Huronium, als es durch die ständige Freisetzung von Sauerstoff durch Cyanobakterien zum Großen Sauerstoffanstieg kam.
E3-Modell der Eukaryogenese
Der Stamm (en. strain) MK-D1 von Promethearchaeum syntrophicum hat ebenso wie der 2022 kultivierte Stamm B-35 von Lokiarchaeum ossiferum[36] lange „Tentakel“, in denen Partnermikroben (Bakterien) nisten und die ihm womöglich als „Protomitochondrien“ verbesserte Überlebenschancen bei steigendem Sauerstoff ermöglichen. Nach dem E3-Modell der Eukaryogenese konnten so Bakterien als Vorfahren der Mitochondrien umschlossen und endogenisiert werden.[37][8][38][39]
Systematik
Der taxonomische Rang der Asgard-Archaeen und ihrer Mitgliedsgruppen ist derzeit (2019–2025) noch in Diskussion.[32]
Mit Stand 2. Juni 2025 ist die Systematik der Asgard-Archaeen nach der LPSN, ergänzt um die Phyla Heimdallarchaeota, Helarchaeota und Thorarchaeota gemäß der NCBI-Taxonomie wie folgt:[3][40][41][42]
Reich Promethearchaeati Imachi et al. 2024 [Asgard-Archaeen]
- Phylum Promethearchaeota Imachi et al. 2024 [„Ca. Lokiarchaeota“ corrig. Spang et al. 2015, bzw. „Ca. Lokiarchaeia“ Spang et al. 2015, „Ca. Lokiarchaeia“ Bulzu et al. 2019 — „Candidatus Baldrarchaeia“ Liu et al. 2021, „Ca. Borrarchaeota“ Liu et al. 2021, „Ca. Hermodarchaeota“ Liu et al. 2021, „Ca. Njordarchaeota“ Xie et al. 2022, „Ca. Odinarchaeota“ Zaremba-Niedzwiedzka et al. 2017, „Ca. Wukongarchaeota“ Liu et al. 2021]
- Klasse „Candidatus Asgardarchaeia“ Tamarit et al. 2024 (Asgard-Archaeen s. s.)
- Ordnung „Ca. Asgardarchaeales“ Tamarit et al. 2024
- Familie „Ca. Asgardarchaeaceae“ Tamarit et al. 2024
- Gattung „Ca. Asgardarchaeum“ Tamarit et al. 2024
- Spezies „Ca. Asgardarchaeum abyssi“ Tamarit et al. 2024, mit:
- Stämme AB3033_2; AB3033_2TS
- Spezies „Ca. Asgardarchaeum abyssi“ Tamarit et al. 2024, mit:
- Gattung „Ca. Asgardarchaeum“ Tamarit et al. 2024
- Familie „Ca. Asgardarchaeaceae“ Tamarit et al. 2024
- Ordnung „Ca. Asgardarchaeales“ Tamarit et al. 2024
- Klasse „Candidatus Asgardarchaeia“ Tamarit et al. 2024 (Asgard-Archaeen s. s.)
- Klasse „Candidatus Atabeyarchaeia“ Valentin-Alvarado et al. 2024
- Ordnung „Ca. Atabeyarchaeales“ Valentin-Alvarado et al. 2024
- Familie „Ca. Atabeyarchaeaceae“ Valentin-Alvarado et al. 2024
- Gattung „Ca. Atabeyarchaeum“ Valentin-Alvarado et al. 2024
- Spezies „Ca. Atabeyarchaeum deiterrae“ Valentin-Alvarado et al. 2024! mit
- Stamm SRVP18_Atabeyarchaeia-2 – Fundort: Metagenom aus Süßwasser-Sediment, Lake County, Kalifornien, USA
- Spezies „Ca. Atabeyarchaeum deiterrae“ Valentin-Alvarado et al. 2024! mit
- Gattung „Ca. Atabeyarchaeum“ Valentin-Alvarado et al. 2024
- Familie „Ca. Atabeyarchaeaceae“ Valentin-Alvarado et al. 2024
- Ordnung „Ca. Atabeyarchaeales“ Valentin-Alvarado et al. 2024
- Klasse „Candidatus Atabeyarchaeia“ Valentin-Alvarado et al. 2024
- Klasse „Candidatus Baldrarchaeia“ Liu et al. 2021
- Ordnung „Ca. Baldrarchaeales“ Liu et al. 2021
- Familie „Ca. Baldrarchaeaceae“ Liu et al. 2021
- Gattung „Ca. Baldrarchaeum“ Liu et al. 2021
- Spezies „Ca. Baldrarchaeum yapense“ corrig. Liu et al. 2021 [Spezies „Ca. Baldrarchaeum yapensis“ Liu et al. 2021], mit:
- Stamm As_130
- Familie „Ca. Baldrarchaeaceae“ Liu et al. 2021
- Ordnung „Ca. Baldrarchaeales“ Liu et al. 2021
- Klasse „Candidatus Baldrarchaeia“ Liu et al. 2021
- Klasse „Candidatus Borrarchaeia“ Liu et al. 2021
- Ordnung „Ca. Borrarchaeales“ Liu et al. 2021
- Familie „Ca. Borrarchaeaceae“ Liu et al. 2021
- Gattung „Ca. Borrarchaeum“ Liu et al. 2021
- Spezies „Ca. Borrarchaeum weybense“ Sun et al. 2021
- Spezies „Ca. Borrarchaeum yapense“ corrig. Liu et al. 2021 [„Ca. Borrarchaeum yapensis“ Liu et al. 2021], mit
- Stamm As_181
- Gattung „Ca. Borrarchaeum“ Liu et al. 2021
- Familie „Ca. Borrarchaeaceae“ Liu et al. 2021
- Ordnung „Ca. Borrarchaeales“ Liu et al. 2021
- Klasse „Candidatus Borrarchaeia“ Liu et al. 2021
- Klasse „Candidatus Freyarchaeia“ Valentin-Alvarado et al. 2024
- ohne Ordnungszuweisung.
- Familie „Ca. Freyarchaeaceae“ Valentin-Alvarado et al. 2024
- ohne Ordnungszuweisung.
- Klasse „Candidatus Freyarchaeia“ Valentin-Alvarado et al. 2024
- Klasse „Candidatus Heimdallarchaeia“ Rinke et al. 2021 [früher Uncultured Archaeal Phylum 3 (UAP3/AAG)]
- Klasse „Candidatus Hermodarchaeia“ Liu et al. 2021
- Ordnung „Ca. Hermodarchaeales“ Liu et al. 2021
- Familie „Ca. Hermodarchaeaceae“ Liu et al. 2021
- Gattung „Ca. Hermodarchaeum“ Liu et al. 2021
- Spezies „Ca. Hermodarchaeum yapense“ corrig. Liu et al. 2021 [„Ca. Hermodarchaeum yapensis“ Liu et al. 2021], mit
- Stamm Yap4.bin9.105
- Spezies „Ca. Hermodarchaeum yapense“ corrig. Liu et al. 2021 [„Ca. Hermodarchaeum yapensis“ Liu et al. 2021], mit
- Gattung „Ca. Hermodarchaeum“ Liu et al. 2021
- Familie „Ca. Hermodarchaeaceae“ Liu et al. 2021
- Ordnung „Ca. Hermodarchaeales“ Liu et al. 2021
- Klasse „Candidatus Hermodarchaeia“ Liu et al. 2021
- Klasse „Candidatus Jordarchaeia“ Sun et al. 2021 [Jordiarchaeia (GTDB)]
- Ordnung „Ca. Jordarchaeales“ Sun et al. 2021 [Jordiarchaeales (GTDB)]
- Familie „Ca. Jordarchaeaceae“ Sun et al. 2021 [Jordiarchaeaceae (GTDB)]
- Gattung „Ca. Jordarchaeum“ Sun et al. 2021 [Jordiarchaeum (GTDB)]
- Spezies „Ca. Jordarchaeum madagascariense“ Sun et al. 2021 [Jordiarchaeum madagascariense (GTDB)], mit
- Stamm EB_bin_7
- Spezies Jordiarchaeum sp018383815 (GTDB) [inkl. Ca. Jordarchaeia archaeon isolate QZM_A2_3],[43] mit
- Stamm QZM_A3
- Stamm QZM_A2_3 alias QZMA23B3 – Fundort: Metagenom aus Thermalquelle, Gemeinde Quchomo (alias Qǔzhuómù, QuZhuoMu) im Kreis Cona (Shannan) (alias CuoNa, Tshona), Tibet (siehe Asgardviren)
- Spezies „Ca. Jordarchaeum madagascariense“ Sun et al. 2021 [Jordiarchaeum madagascariense (GTDB)], mit
- Gattung „Ca. Jordarchaeum“ Sun et al. 2021 [Jordiarchaeum (GTDB)]
- Familie Freyrarchaeaceae (GTDB)
- Gattung Freyrarchaeum (GTDB)
- Spezies Ca. Freyrarchaeum guaymaensis (NCBI), mit
- Stamm GB_128 alias GB128 – Fundort: marines Metagenom vom Guaymas-Becken, Golf von Kalifornien
- Spezies Freyrarchaeum sp029258835 (GTDB), mit
- Stamm S9B3_HD70
- Spezies Ca. Freyrarchaeum guaymaensis (NCBI), mit
- Gattung Freyrarchaeum (GTDB)
- Familie „Ca. Jordarchaeaceae“ Sun et al. 2021 [Jordiarchaeaceae (GTDB)]
- Ordnung „Ca. Jordarchaeales“ Sun et al. 2021 [Jordiarchaeales (GTDB)]
- Klasse „Candidatus Jordarchaeia“ Sun et al. 2021 [Jordiarchaeia (GTDB)]
- Klasse „Candidatus Odinarchaeia“ Tamarit et al. 2022 [bzw. „Ca. Odinarchaeia“ Vogel et al. 2021]
- Klasse Promethearchaeia Imachi et al. 2024 [„Ca. Lokiarchaeia“ Spang et al. 2015[14], c__Lokiarchaeia (GTDB)], mit
- Spezies „Ca. Lokiarchaeum ossiferum“, Promethearchaeum syntrophicum
- Klasse „Candidatus Sifarchaeia“ Sun et al. 2021 [früher als Phylum „Ca. Sifarchaeota“[44]]
- Klasse „Candidatus Wukongarchaeia“ Liu et al. 2021
- Ordnung „Ca. Wukongarchaeales“ Liu et al. 2021
- Familia „Ca. Wukongarchaeaceae“ Liu et al. 2021
- Gattung „Ca. Wukongarchaeum“ Liu et al. 2021
- Spezies „Ca. Wukongarchaeum yapense“ corrig. Liu et al. 2021 [„Ca. Wukongarchaeum yapensis“ Liu et al. 2021], mit
- Stamm As_085
- Spezies „Ca. Wukongarchaeum yapense“ corrig. Liu et al. 2021 [„Ca. Wukongarchaeum yapensis“ Liu et al. 2021], mit
- Gattung „Ca. Wukongarchaeum“ Liu et al. 2021
- Familia „Ca. Wukongarchaeaceae“ Liu et al. 2021
- Ordnung „Ca. Wukongarchaeales“ Liu et al. 2021
- Klasse „Candidatus Wukongarchaeia“ Liu et al. 2021
- Klasse c__LC30 (früher in GTDB) – nur schwach belegt
- Ordnung o__LC30
- Familie f__LC30
- Gattung LC30
- Spezies Promethearchaeati archaeon isolate LC20 (NCBI) [früher LC30 sp019058495 (GTDB)], mit
- Stamm LC20 – Fundort: Lake Cootharaba,[45][46] Sunshine Coast, Australien[47]
- Spezies Promethearchaeati archaeon isolate LC30 (NCBI) [früher LC30 sp019058495 (GTDB)], mit
- Stamm LC30 – Fundort: Lake Cootharaba,[45][46] Sunshine Coast, Australien[47]
- Spezies Promethearchaeati archaeon isolate LC20 (NCBI) [früher LC30 sp019058495 (GTDB)], mit
- Gattung LC30
- Familie f__LC30
- Ordnung o__LC30
- Klasse c__LC30 (früher in GTDB) – nur schwach belegt
- ?Klasse „Candidatus Njordarchaeia“ Xie et al. 2022 – nach einer Studie aus dem Jahr 2025 gehören die Njordarchaeen zum „TACK-Superphylum“ (Reich Thermoproteati)[48]
Die folgenden Phyla sind nach der NCBI-Taxonomie Mitglieder der Promethearchaeati (Asgard-Archaeen), in der LPSN aber innerhalb der Domäne ohne Zuordnung. In der GTDB sind sie in der Regel mit niedrigerem taxonomischem Rang einer der obigen Klassen zugeordnet:
- Phylum „Ca. Hodarchaeota“ corrig. Liu et al. 2021 – in der GTDB als Ordnung Hodarchaeales der (Kandidaten-)Klasse „Ca. Heimdallarchaeia“ untergeordnet
- Phylum „Ca. Kariarchaeota“ Liu et al. 2020 – in der GTDB als Familie Kariarchaeaceae der (Kandidaten-)Ordnung Ca. Heimdallarchaeales in Ca. Heimdallarchaeia untergeordnet
- Phylum „Ca. Thorarchaeota“ corrig. Seitz et al. 2016 – in der GTDB als Klasse dem Phylum Asgardarchaeota untergeordnet, das im Umfang dem Reich Promethearchaeati entspricht.
- Phylum „Ca. Gerdarchaeota“ Cai et al. 2020 – in der LPSN unbekannt (alle Taxa der Form „Geradarch…“), es scheint aber eine nicht sehr gut belegte Übereinstimmung zwischen einer Ordnung Gerdarchaeales und der GTDB-Ordnung o__JABLTI01 in „Ca. Heimdallarchaeia“ zu geben (siehe dort).
- ?Phylum „Ca. Helarchaeota“ Seitz et al. 2019 – in der GTDB und bei Zhang et al. (2025) als Ordnung Helarchaeales der Klasse Lokiarchaeia (= Promethearchaeia) untergeordnet[48]
Nicht berücksichtigt wird hier der folgende verwaiste LPSN-Eintrag:
- ?Phylum „Ca. Heimdallarchaeota“ Zaremba-Niedzwiedzka et al. 2017 bzw. corrig. Bulzu et al. 2019 [„Ca. Heimdallarchaeia“ Bulzu et al. 2019] – Eintrag in der LPSN verwaist; der Eintrag zur Kandidaten-Klasse ist dem Phylum Promethearchaeota untergeordnet. In der GTDB (ohne den Rang „Reich“) ist die Klasse dem Phylum Asgardarchaeota unterstellt, das (als höchstrangiges Taxon unter der Domäne der Archaeen) dem Reich Promethearchaeati entspricht.
Anmerkungen:
- Die Tendenz war früher, neuen Kladen mit dem Rang eines Phylums (Endung „-archaota“) zu versehen. Die Taxonomie seit 2024 hat für Superphyla den Rang Reich (englisch kingdom) hinzugefügt (s. o.), aber gleichzeitig viele Kladen im Rang eines Phylums zu Klassen „degradiert“.
- Die vielen Synonyme der wissenschaftlichen Bezeichnung Promethearchaeati resultieren u. a. daher, dass einige Mitgliedskladen früher im Rang eines Phylums gesehen wurden, und ihr Bezeichner ein entsprechendes Suffix (‚-ota‘, meist ‚-archaeota‘) trug. Wenn diese Kladen heute als Klassen (mit einem entsprechenden neuen Suffix, etwa ‚-archaeia‘) im ein und demselben Phylum gesehen werden, dann werden die alten Bezeichner auf der Rang-Ebene der Phyla formal zu Synonymen für das heutige gemeinsame Phylum. Die inhaltliche Entsprechung ist aber die eines Subsets (z. B. Klasse). In der obigen Liste sind diese formalen Synonyme von inhaltlichen durch einen Bindestrich (‚—‘) abgesetzt.
Weitere als Mitglieder vorgeschlagene Kandidatengruppen
- „Ca. Gefionarchaeota“ Caceres 2019[32][51]
- „Ca. Friggarchaeota“ Caceres 2019[32][52]
- „Ca. Idunnarchaeota“ Caceres 2019[53] (Schwestergruppe der Heimdallarchaeota und möglicherweise die nächsten Verwandten der Eukaryoten)[32]
- „Ca. Freyrarchaeota“ Xie et al. 2022 mit Ordnung „Ca. Freyrarchaeales“ (in Klasse „Ca. Freyrarchaeia“) und darin die Spezies „Ca. Freyrarchaeum guaymaensis“" Xie et al. 2022 (Fundort: Guaymas-Becken)[49][54][55] In der GTDB ist die Familie Freyrarchaeaceae Mitglied der Ordnung Jord[i]archaeales, d. h. die Ordnung und Klasse der Frayrarchaeen sind nur Synonyme der entsprechenden Taxa der Jof[i]archaeen.
- „Ca. Jordarchaeia“ Sun et al. 2021[56][47] [Jodiarchaeia (GTDB)].[1]
- „Ca. Sigynarchaeota“ Xie et al. 2022 mit Ordnung „Ca. Sigynarchaeiales“ (in Klasse „Ca. Sigynarchaeia“) und darin die Spezies „Ca. Sigynarchaeum springense“" Xie et al. 2022 (Fundort: Thermalquelle der Tengchong-Vulkangruppe)[49][57]
Die Gruppe „Uncultured Archaeal Phylum 3 (UAP3/AAG)“ Parks et al. 2017[58] mit „Ancient Archaeal Group (AAG)“ Takai & Horikoshi 1999[59][60][61] beinhaltet (ursprünglich) nur eine vorgeschlagene Spezies „Archaeon UBA460“ (Fundort: Meeressediment vor Costa Rica).[62] Zwar ist diese Spezies in der NCBI-Taxonomie keiner bestimmten Archaeengruppe zugeordnet (Stand 3. Januar 2023),[63] in der Genome Taxonomy Database (GTDB) aber trägt diese Spezies die Bezeichnung UBA460 sp002505645 und ist den Heimdallarchaeia (syn. Heimdallarchaeota, s. u.) zugeordnet.[64] „Uncultured Archaeal Phylum 3 (UAP3/AAG)“ ist daher die frühere provisorische Bezeichnung für die Heimdallarchaeen.
Nach Xie et al. (2022) sollten die Sigynarchaeota Mitglied der durch Helarchaeota und Lokiarchaeota definierten Klade zu sein, das sie näher mit den Lokiarchaeota als den Helarchaeota verwandt erscheinen. Die Njordarchaeen stehen wie die Wukongarchaeen den Heimdallarchaeen nahe und gehören daher in dieselbe große Asgard-Klade.[49]
Nach Farag et al. (2021) scheinen die Sifarchaeen am nächsten mit den Thorarchaeen verwandt zu sein. Die Thorarchaeen sind bei diesen Autoren (mit den Sifarchaeen) aber im Asgard-Zweig der „Heimdallarchaeota“ angesiedelt, und nicht bei den Loki-Archaeen.[65]
Phylogenie
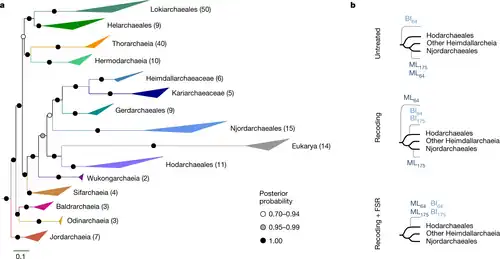
Die phylogenetischen Beziehungen innerhalb der Klade der Asgard-Archaeen sind größtenteils noch in der Diskussion.
Die Kandidaten-Klasse „Heimdallarchaeia“ (und ggf. ihnen nahestehende Gruppen) gelten als die am tiefsten verzweigten Asgard-Archaea.[8] Die Eukaryoten können eine Schwesterklade der Asgard-Archaeen als Ganzes oder der Klasse „Ca. Heimdallarchaeia“ bzw. den nahe verwandten Idunnarchaeen sein.[66][32] Ein bevorzugtes Szenario ist die Syntrophie, bei der ein Organismus auf die Ernährung des anderen angewiesen ist. In diesem Fall könnte die Syntrophie darauf zurückzuführen sein, dass die Asgard-Archaeen in eine unbekannte Bakterienart inkorporiert wurden und sich zum Zellkern entwickelten. Zudem wurde ein α-Proteobakterium wurde inkorporiert und entwickelte sich so zum Mitochondrium.[66] Ein anderes vorgeschlagenes Modell ist die virale Eukaryogenese.
Die seit Mitte der 2010er Jahre vorgeschlagenen phylogenetischen Bäume spiegeln jedoch die Erkenntnis wider, dass die DNA rezenter (heutiger) Asgard-Archaeen enger mit der DNA in den eukaryotischen Zellkerne verwandt ist als mit der DNA anderer Archaeen.[39][47][49]
In der 2023 veröffentlichten Studie von Laura Eme, Daniel Tamarit et al. plädieren die Autoren dafür, die Asgard-Gruppe im weiten Sinn aufzufassen, einschließlich der tief in dieser Gruppe wurzelnden Eukaryoten. Nach diesen Autoren sieht der phylogenetische Baum der „Eukaryomorpha“ etwa wie folgt aus:[21]
| Promethearchaeati[A. 3] |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Phylogenie nach der GTDB (Stand April 2025):[69]
| Promethearchaeati[A. 10] |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Anmerkungen:
- Als Schwesterklade der Eukaryoten wurde von Laura Eme et al. (2023) die Asgard-Ordnung „Hodarcheales“[21] mit Ca. Hodarchaeum mangrovi (Referenzstamm FT_5_011) identifiziert.[70][A. 11]
- Als Kandidaten für die Proto-Mitochondrien unter den α-Proteobacteria wurden früher die Rickettsiales gehandelt, neuerdings (seit 2023) werden die zwischenzeitlich gefundenen und mit den Rickettsiales weitläufig verwandten Iodidimonadales favorisiert.
- Im Gegensatz zu Eme et al. (2023) sehen Zhang et al. (2025) die Eukaryoten als Schwestergruppe der Heimdallarchaeen. Wie auch immer, den letzten gemeinsamen Vorfahren der heutigen Asgard-Archaeen und Eukaryoten nennen sie „LAECA“ (Last Asgard archaea and Eukaryote Common Ancestor).[48]
Sollten tatsächlich die Eukaryoten aus der Mitte der Asgard-Archaeen entspringen (so wie im Kladogramm), und will man diese Gruppe im engeren Sinn auffassen – dann ist sie möglicherweise paraphyletisch, da die Eukaryoten von ihrem letzten gemeinsamen Vorfahren (last common ancestor, LCA) (und den α─Proteobacteria) abstammen. Alternativ kann man diese Gruppen im weiteren Sinn auffassen – dann sind die Eukaryoten ebenfalls (sehr weit entwickelte) Asgard-Mitglieder. Als Konsequenz wurde folgende Bezeichnung vorgeschlagen:
- „Eukaryomorpha“ Fournier & Poole 2018[72]
Diese Bezeichnung umfasst explizit alle Asgard-Archaeen zusammen mit den Eukaryoten.
Die Problematik setzt sich unter dieser Annahme in den höheren taxonomischen Rängen fort, bis hin zur Domäne. Man kann die Bezeichnung Archaea (Archaeen) im weiten Sinn verstehen (inklusive Eukaryoten), dann wären diese monophyletisch. Es gäbe nur zwei Domänen: neben den so erweiterten Archaeen nur noch die Bacteria (Bakterien). Oder man versteht den Begriff Archaeen im engen Sinn und behält den bisherigen Sprachgebrauch bei, dann ist diese Gruppe paraphyletisch. Als taxonomischer Oberbegriff, der Archaeen und Eukaryoten umfasst, wurde 2020 von Cavalier-Smith und Chao die Bezeichnung „Neomura“ vorgeschlagen, dies wäre dann eine Schwestergruppe der Bacteria.[73][A. 12]
Als Ausweg stellt möglicherweise das vorgeschlagene Modell „Ring des Lebens“ die Bedeutung der bakteriellen Endosymbionten bei der Eukaryogenese in den Vordergrund und rechtfertigt damit die dritte Domäne der Eukaryoten (mit eigener Taxonomie): Infolge dieses Verschmelzungsprozesses sind Eukaryonten weit mehr als einfach nur sehr weit fortentwickelte Asgard-Archaeen; siehe Eukaryogenese §Letzter eukaryotischer gemeinsamer Ahn (LECA).
Namensherkunft
Namensherkunft:
- Promethearchaeati ⇒ Prometheus
――――
- „Baldrarchaeia“[7][75][32] ⇒ Balder
- „Borrarchaeia“[7][76] ⇒ Börr
- „Freyarchaeia“[32] ⇒ Freya
- „Freyrarchaeota“[49][54] ⇒ Freyr
- „Friggarchaeota“[32] ⇒ Frigg
- „Gefionarchaeota“[32] ⇒ Gefion
- „Gerdarchaeota“[77][77] ⇒ Gerda
- „Heimdallarchaeia“[78][79] ⇒ Heimdall
- „Helarchaeota“[80][81][82] ⇒ Hel
- „Hermodarchaeia“[7][83] ⇒ Hermodr
- „Hodarchaeota“[7][84] ⇒ Hödur
- „Idunnarchaeota“[53][32] ⇒ Idun
- „Kariarchaeota“[7][85] ⇒ Kari
- „Lokiarchaeota“[86][87] ⇒ Loki
- „Njordarchaeia“[49][50] ⇒ Njörðr
- „Odinarchaeia“[88][89] ⇒ Odin
- „Sifarchaeia“[65] ⇒ Sif
- „Sigynarchaeota“[49][57] ⇒ Sigyn
- „Thorarchaeota“[18][5][42] ⇒ Thor
――――
- „Wukongarchaeia“[7][90] ⇒ Sūn Wùkōng (chinesisch 孫悟空 / 孙悟空, W.-G. Sun Wu-k'ung)
- „Panguiarchaeaceae“[69] ⇒ Pángǔ (chinesisch 盤古 / 盘古, Pinyin Pángǔ, W.-G. P'an-ku)
Asgardviren
Die Viren der Asgard-Archaeen werden (nicht-taxonomisch) kurz als Asgardviren (en. Asgard viruses) klassifiziert. Bisher liegen nur Metagenomdaten vor, insbesondere aus dem CRISPR/Cas-Abwehrsystem, das Genomsequenzen des Virus zwecks Erkennung umfasst. Diese Asgardviren sind dsDNA-Viren, die gewisse Ähnlichkeiten sowohl zu anderen prokaryotischen dsDNA-Viren als auch zu eukaryotischen dsDNA-Viren zeigen; ein Umstand, der zur Abstammung der Eukaryoten aus dem Umfeld der Asgard-Archaeen (unter Einbeziehung eines endosymbiotischen Bakteriums) passt – ggf. unter Mithilfe von Viren (siehe Eukaryogenese, Eozyten-Hypothese). Im April 2023 hat das ICTV die ersten Vertreter von Asgardviren offiziell bestätigt. Für ihre Gesamtheit kann eine vorläufige Klassifizierung nach Wirten, Morphologie und Habitat erfolgen:[91]
- Wirte: Lokiarchaeia (=Promethearchaeia) und „Ca. Helarchaeia“:[92]
- Fenrir-Viren: Wirte: Lokiarchaeota, Fundort: Hydrothermalquellen im Guaymas-Becken (Golf von Kalifornien)
- Nidhogg-Viren: Wirte: Helarchaeota, Fundort: Guaymas-Becken[A. 13]
- Ratatoskr-Viren: Wirte: Lokiarchaeota, Fundort: Guaymas-Becken
- „Lokiarchaeota virus Ratatoskr Meg22_1012“[98]
- Sköll-Viren, Wirte: Lokiarchaeota, Fundort: Guaymas-Becken
- „Lokiarchaeota virus Skoll Meg22_1214“[99] mit Schreibvariante „Lokiarchaeota virus Sköll Meg22_1214“
- Wirte: „Ca. Heimdallarchaeia“, speziell „Ca. Heimdallarchaeum aukensis“: HeimV1 und HeimV2; Fundort: Hydrothermalfeld Auka im Pescadero-Becken, Golf von Kalifornien, Mexiko. Es handelt sich nach den Metagenomdaten vermutlich um Vertreter der Caudoviricetes vom Morphotyp der Siphoviren.[68]
- Wirte: „Ca. Odinarchaeum yellowstonii“ LCB_4, „Ca. Promethearchaeota archaeon isolate E29_bin63“ und Jordiarchaeum sp018383815 (GTDB) [Ca. Jordarchaeia archaeon isolate QZM_A2_3] Stamm QZMA23B3, sowie andere (Nitrososphaeria). Das Virusgenom deutet auf integrierte spindel- oder ellipsoidförmige Viren (vgl. Familien Bicaudaviridae, Fuselloviridae bzw. Ovaliviridae) hin. Fundort: Thermalquellen im Yellowstone-Nationalpark. Vorgeschlagene Namen „Muninnvirus“ (en. Muninn virus) und „Huginnvirus“ (en. Huginn virus). Ähnlichkeiten mit:[100]
- Sulfolobus ellipsoid virus 1 (SEV1, Gattung: Alphaovalivirus, Fam. Ovaliviridae),
- Icerudivirus SIRV2 (alias Sulfolobus islandicus rod-shaped virus 2, SIRV2, Fam. Rudiviridae),
- „Sulfolobus tengchongensis spindle-shaped virus 1“ (STSV1, Bicaudaviridae),
- Sulfolobus spindle-shaped virus 1 (SSV1, Fuselloviridae).
- Wirte: Asgard-Archaeen, Fundort: Tiefsee-Sedimente, z. B. bei der Shimokita-Halbinsel (Japan)[101]
- Verdandiviridae (Verdandiviren, en. verdandiviruses): Kopf-Schwanz-Aufbau (Klasse Caudoviricetes, Morphotyp Siphoviren):
- Skuldviridae (Skuldviren, en. skuldviruses): zum Reich Bamfordvirae, Phylum Preplasmiviricota, Klasse Tectiliviricetes, Ordnung Atroposvirales
- „Wyrdviren“ (en. “wyrdviruses”): zitronenförmige Gestalt (verwandt mit spindelförmigen Viren der Familie Halspiviridae bzw. Fuselloviridae):
- Spezies „Lokiarchaeota-Virus WyrdV1“ (zu Fuselloviridae, Wirte: Lokiarchaeota)[102]
- Spezies „Lokiarchaeota-Virus WyrdV2“ (Wirte: Lokiarchaeota)
- WyrdV3 – WyrdV7.
Namensherkunft der Viren
Namensherkunft nach der nordischen Mythologie:[91]
- Fenrir (wörtlich ‚der im Sumpf Lebende‘, auch Fenriswolf genannt): ein Wolf, Sohn des Loki
- Sköll (auch Skoll, Skøll oder Skalli): ein weiterer Wolf, Sohn des Fenrir
- Nidhogg (alias Nidhöggr): ein schlangenförmiger Drache, der im Weltenbaum Yggdrasil lebt
- Ratatoskr (alias Ratatöskr): das Eichhörnchen des Yggdrasil
- Huginn und Muninn (mit Schreibvariante Hugin respektive Munin), die beiden Raben Odins
- Verdandi (auch Werdandi), Skuld (vgl. de ‚Schuld‘) und Wyrd (auch Wurd, vgl. Urðr): drei Nornen (Schicksalsgöttinnen)
Anmerkungen
- ↑ benannt nach Nikolai Michailowitsch Knipowitsch
- ↑ Die Eozyten-Hypothese war in den 1980er Jahren – vor Entdeckung der ersten Asgard-Archaeen – aufgrund gewisser Übereinstimmungen der Crenarchaeota mit den Eukaryoten entwickelt worden. Diese gehören wir die Asgard-Archaeen den Proteoarchaeota an; „Eozyten“ ist eine veraltete Bezeichnung für die Crenarchaeota bzw. sensu lato ein Synonym für die Proteoarchaeota.
- ↑ = Asgard-Archaeen [„Asgardarchaeota“ (GTDB)]
- ↑ = „Asgardarchaeia“
- ↑ = Sigynarchaeales (GTDB) oder „Lokiarchaeales“
- ↑ a b = Lokiarchaeen
- ↑ a b = o__JABLTI01 (GTDB)
- ↑ = f__UBA460 (GTDB)
- ↑ = „Kariarchaeales“, o__UBA460 (GTDB)
- ↑ = Asgard-Archaeen [„Asgardarchaeota“ (GTDB)]
- ↑ Fundort: Mangroven des Futian Nature Reserve bei Shenzhen, China.[71]
- ↑ Der Begriff „Neomura“ wurde von Cavalier-Smith bereits 2002 vorgeschlagen, damals im Rahmen eines zur Eozyten-Hypothese alternativen Szenarios. Inzwischen räumt der Autor aufgrund der neuen Ergebnisse aber auch die Möglichkeit einer Abstammung der Eukaryoten aus einer Gruppe der Archaeen ein. Die Bezeichnung „Neomura“ als Oberbegriff für Archaeen s. s. und Bakterien bleibt davon aber weitgehend unberührt: Neomura Thomas Cavalier-Smith (2002).[74]
- ↑ Die Nidhogg-Asgardviren sind zu unterscheiden vom Mykobakterien-Virus „Mycobacterium phage Nidhogg“, Klasse Caudoviricetes, Morphotyp Myoviren (Vorschlag).[95]
Weblinks
- Traci Watson: The trickster microbes that are shaking up the tree of life, in: Nature, 14. Mai 2019
- NCBI: Asgard group (clade)
- OneZoom: Asgardarchaeota
- Pedro Leão, Mary E. Little, Kathryn E. Appler, Daphne Sahaya, Emily Aguilar-Pine, Kathryn Currie, Ilya J. Finkelstein, Valerie De Anda, Brett J. Baker: Asgard archaea defense systems and their roles in the origin of eukaryotic immunity. In: Nature Communications, Band 15, Nr. 6386, 31. Juli 2024; doi:10.1038/s41467-024-50195-2 (englisch). Dazu:
- Carly Cassella: Human Defenses Against Viruses First Evolved Billions of Years Ago, Study Suggests. Auf: sciencealert vom 8. September 2024.
- Masaru Nobu: Engineering history with Asgard archaea of the kingdom Promethearchaeati. In: Nature Microbiology, Band 9, Nr. 12, November 2024; doi:10.1038/s41564-024-01866-9, ResearchGate:386004321 (englisch).
- Wen-Cong Huang, Maraike Probst, Zheng-Shuang Hua, Lénárd L. Szánthó, Gergely J. Szöllősi, Thijs J. G. Ettema, Christian Rinke, Tom A. Williams, Anja Spang: Phylogenomic analyses reveal that Panguiarchaeum is a clade of genome-reduced Asgard archaea. In: bioRχiv, 17. Februar 2025; doi:10.1101/2025.02.13.637844 (Preprint, englisch).
Einzelnachweise
- ↑ a b GTDB: Phylum Asgardarchaeota
- ↑ Violette Da Cunha, Morgan Gaia, Daniele Gadelle, Arshan Nasir, Patrick Forterre: Lokiarchaea are close relatives of Euryarchaeota, not bridging the gap between prokaryotes and eukaryotes. In: PLoS Genet., Band 13, Nr. 6, 12. Juni 2017, e1006810, doi:10.1371/journal.pgen.1006810.
- ↑ a b c d
LPSN: Kingdom Promethearchaeati Imachi et al. 2024.
Phylum Promethearchaeota Imachi et al. 2024. - ↑ NCBI: Promethearchaeati, Details: Promethearchaeati Imachi et al. 2024 (kingdom), heterotypic synonym: Candidatus Asgardarchaeota, …; graphisch: Promethearchaeati (Asgard group), auf: Lifemap, NCBI Version.
- ↑ a b
LPSN: Phylum "Candidatus Thorarchaeota" corrig. Seitz et al. 2016.
Phylum "Candidatus Thorarchaeia" Seitz et al. 2016. - ↑ a b c d Katarzyna Zaremba-Niedzwiedzka, Eva F. Cáceres, Jimmy H. Saw, Disa Bäckström, Lina Juzokaite, Emmelien Vancaester, Kiley W. Seitz, Karthik Anantharaman, Piotr Starnawski, Kasper U. Kjeldsen, Matthew B. Stott, Takuro Nunoura, Jillian F. Banfield, Andreas Schramm, Brett J. Baker, Anja Spang, Thijs J. G. Ettema: Asgard archaea illuminate the origin of eukaryotic cellular complexity. In: Nature. 541. Jahrgang, 11. Januar 2017, ISSN 1476-4687, ResearchGate:312324762, PDF (umn.edu), S. 353–358, doi:10.1038/nature21031, PMID 28077874, bibcode:2017Natur.541..353Z (englisch).
- ↑ a b c d e f g h Yang Liu, Kira S. Makarova, Wen-Cong Huang, Yuri I. Wolf, Anastasia Nikolskaya, Xinxu Zhang, Mingwei Cai, Cui-Jing Zhang, Wei Xu, Zhuhua Luo, Lei Cheng, Eugene V. Koonin, Meng Li: Expanded diversity of Asgard archaea and their relationships with eukaryotes. In: Nature, Band 593, 28. April 2021, S. 553–557; doi:10.1038/s41586-021-03494-3, PMC 11165668 (freier Volltext), PMID 33911286 (englisch).
- ↑ a b c d e f g
Hiroyuki Imachi, Masaru K. Nobu, Nozomi Nakahara, Yuki Morono, Miyuki Ogawara, Yoshihiro Takaki, Yoshinori Takano, Katsuyuki Uematsu, Tetsuro Ikuta, Motoo Ito, Yohei Matsui, Masayuki Miyazaki, Kazuyoshi Murata, Yumi Saito, Sanae Sakai, Chihong Song, Eiji Tasumi, Yuko Yamanaka, Takashi Yamaguchi, Yoichi Kamagata, Hideyuki Tamaki, Ken Takai: Isolation of an archaeon at the prokaryote–eukaryote interface. In: Nature. Band 577, Nr. 7791, 23. Januar 2020, ISSN 1476-4687, S. 519–525, doi:10.1038/s41586-019-1916-6, PMID 31942073, PMC 7015854 (freier Volltext), bibcode:2020Natur.577..519I (englisch, nature.com). Dazu:
- Yasemin Saplakoglu: Out of Deep-Sea Mud, a Strange Blob May Hold Secrets to the Origins of Complex Life. Auf: LiveScience vom 17. Januar 2020.
- ↑ a b Laura Eme, Anja Spang, Jonathan Lombard, Courtney W. Stairs, Thijs J. G. Ettema: Archaea and the origin of eukaryotes. In: Nature Reviews Microbiology. 15. Jahrgang, Nr. 12, 10. November 2017, ISSN 1740-1534, S. 711–723, doi:10.1038/nrmicro.2017.133, PMID 29123225 (englisch).
- ↑ Tom A. Williams, Cymon J. Cox, Peter G. Foster, Gergely J. Szöllősi, T. Martin Embley: Phylogenomics provides robust support for a two-domains tree of life. In: Nature Ecology & Evolution. 4. Jahrgang, Nr. 1, 9. Dezember 2019, ISSN 2397-334X, S. 138–147, doi:10.1038/s41559-019-1040-x, PMID 31819234, PMC 6942926 (freier Volltext) – (englisch).
- ↑ Scientists Break Record By Finding Northernmost Hydrothermal Vent Field. 24. Juli 2008, abgerufen am 30. April 2021 (englisch). Auf: ScienceDaily vom 4. Juli 2008.
- ↑ Steffen Leth Jørgensen, Bjarte Hannisdal, Anders Lanzen, Tamara Baumberger, Kristin Flesland, Rita Fonseca, Lise Øvreås, Ida H. Steen, Ingunn H. Thorseth, Rolf B. Pedersen, Christa Schleper: Correlating microbial community profiles with geochemical data in highly stratified sediments from the Arctic Mid-Ocean Ridge. In: PNAS. 109. Jahrgang, Nr. 42, 5. September 2012, S. E2846–55, doi:10.1073/pnas.1207574109, PMID 23027979, PMC 3479504 (freier Volltext) – (englisch).
- ↑ Steffen Leth Jørgensen, Ingunn H. Thorseth, Rolf B. Pedersen, Tamara Baumberger, Christa Schleper: Quantitative and phylogenetic study of the Deep Sea Archaeal Group in sediments of the Arctic mid-ocean spreading ridge. In: Frontiers in Microbiology. 4. Jahrgang, 4. Oktober 2013, S. 299, doi:10.3389/fmicb.2013.00299, PMID 24109477, PMC 3790079 (freier Volltext) – (englisch, frontiersin.org).
- ↑ a b c Anja Spang, Jimmy H. Saw, Steffen L. Jørgensen, Katarzyna Zaremba-Niedzwiedzka, Joran Martijn, Anders E. Lind, Roel van Eijk, Christa Schleper, Lionel Guy, Thijs J. G. Ettema: Complex archaea that bridge the gap between prokaryotes and eukaryotes. In: Nature. 521. Jahrgang, Nr. 7551, Mai 2015, ISSN 1476-4687, S. 173–179, doi:10.1038/nature14447, PMID 25945739, PMC 4444528 (freier Volltext), bibcode:2015Natur.521..173S (englisch).
- ↑ Ed Yong: Break in the Search for the Origin of Complex Life In: The Atlantic. Abgerufen am 30. April 2021 (amerikanisches Englisch).
- ↑ Stefanie von Schnurbein: The Function of Loki in Snorri Sturluson's "Edda". In: History of Religions. 40. Jahrgang, Nr. 2, Oktober 2000, S. 109–124, doi:10.1086/463618 (englisch).
- ↑ Anja Spang, Laura Eme, Jimmy H. Saw, Eva F. Caceres, Katarzyna Zaremba-Niedzwiedzka, Jonathan Lombard, Lionel Guy, Thijs J. G. Ettema, Antonis Rokas: Asgard archaea are the closest prokaryotic relatives of eukaryotes. In: PLOS Genetics. 14. Jahrgang, Nr. 3, 18. März 2018, S. e1007080, doi:10.1371/journal.pgen.1007080, PMID 29596421, PMC 5875740 (freier Volltext) – (englisch).
- ↑ a b Kiley W. Seitz, Cassandre S. Lazar, Kai-Uwe Hinrichs, Andreas P. Teske, Brett J. Baker: Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction. In: The ISME Journal. 10. Jahrgang, Nr. 7, 29. Januar 2016, ISSN 1751-7370, S. 1696–1705, doi:10.1038/ismej.2015.233, PMID 26824177, PMC 4918440 (freier Volltext) – (englisch).
- ↑ Matthew Stott, Craig Cary, Melissa Climo: The 1000 Project – A microbial inventory of geothermal ecosystems. New Zealand Geothermal Workshop, The 1000 Project – A microbial inventory of geothermal ecosystems, GNS Science. 20.–22. November 2012; Radiata Pool.
- ↑ Nukki Macdonald: What lives in New Zealand's hot and hostile volcanic springs?. Auf: stuff vom 7. Mai 2015.
- ↑ a b c d
Laura Eme, Daniel Tamarit, Eva F. Cáceres, Courtney W. Stairs, Valerie De Anda, Max E. Schön, Kiley W. Seitz, Nina Dombrowski, William H. Lewis, Felix Homa, Jimmy H. Saw, Jonathan Lombard, Takuro Nunoura, Wen-Jun Li, Zheng-Shuang Hua, Lin-Xing Chen, Jillian F. Banfield, Emily St. John, Anna-Louise Reysenbach, Matthew B. Stott, Andreas Schramm, Kasper U. Kjeldsen, Andreas P. Teske, Brett J. Baker, Thijs J. G. Ettema: Inference and reconstruction of the heimdallarchaeial ancestry of eukaryotes. In: Nature, Band 618, S. 992–999; doi:10.1038/s41586-023-06186-2. Dazu:
- Nadja Podbregar: Wir sind alle „Asgardianer“ – Eukaryoten entstanden aus einer Untergruppe der Asgard-Archaeen. Auf: scinexx.de vom 26. Juni 2023.
- Asgard Archaea and All Eukaryotes Share Common Ancestor, Study Says. Auf Sci.News vom 26. Juni 2023.
- ”We’re All Asgardians”: Scientists Discover New Clues About the Origin of Complex Life. Auf: SciTechDaily vom 28. Juli 2023.
- Mysterious Microorganisms Unveiled as Key to the Origin of Complex Life. Auf: SciTechDaily vom 6. August 2023.
- ↑ eukaryotic signature proteins (ESPs), ein Satz von Proteinen, die Verwandtschaft zu Eukaryoten anzeigen kann.
- ↑ Dikla Nachmias, Béla P. Frohn, Carsten Sachse, Itzhak Mizrahi, Natalie Elia: ESCRTs – a multi-purpose membrane remodeling device encoded in all life forms. In: Trends in Microbiology, Band 33, Nr. 6, Juni 2025, S. 665–687; doi:10.1016/j.tim.2025.01.009 (englisch).
- ↑ UniProt: Q9H444 · CHM4B_HUMAN.
- ↑ UniProt: P39929 · SNF7_YEAST.
- ↑ Caner Akıl, Robert C. Robinson: Genomes of Asgard archaea encode profilins that regulate actin. In: Nature. 562. Jahrgang, Nr. 7727, 3. Oktober 2018, S. 439–443, doi:10.1038/s41586-018-0548-6, PMID 30283132, bibcode:2018Natur.562..439A (englisch).
- ↑ Caner Akıl, Linh T. Tran, Magali Orhant-Prioux, Yohendran Baskaran, Edward Manser, Laurent Blanchoin, Robert C. Robinson: Complex eukaryotic-like actin regulation systems from Asgard archaea, bioRxiv 10.1101/768580, 14. September 2019, doi:10.1101/768580.
- ↑ Caner Akıl, Linh T. Tran, Magali Orhant-Prioux, Yohendran Baskaran, Edward Manser, Laurent Blanchoin, Robert C. Robinson: Insights into the evolution of regulated actin dynamics via characterization of primitive gelsolin/cofilin proteins from Asgard archaea. In: PNAS. 117. Jahrgang, Nr. 33, 18. August 2020, S. 19904–19913, doi:10.1073/pnas.2009167117, PMID 32747565 (englisch).
- ↑ Odd Nygård, Gunnar Alkemar, Sofia L. Larsson: Analysis of the secondary structure of expansion segment 39 in ribosomes from fungi, plants and mammals. In: J Mol Biol, Band 357, Nr. 3, 31. März 2006, S. 904–916; doi:10.1016/j.jmb.2006.01.043, PMID 16473366.
- ↑ Petar I. Penev, Sara Fakhretaha-Aval, Vaishnavi J. Patel, Jamie J. Cannone, Robin R. Gutell, Anton S. Petrov, Loren Dean Williams, Jennifer B. Glass: Supersized Ribosomal RNA Expansion Segments in Asgard Archaea. In: Genome Biology and Evolution. 12. Jahrgang, Nr. 10, 1. Oktober 2020, S. 1694–1710, doi:10.1093/gbe/evaa170 (englisch).
- ↑ Kira S. Makarova, Yuri I. Wolf, Sergey A. Shmakov, Yang Liu, Meng Li, Eugene V. Koonin: Unprecedented Diversity of Unique CRISPR-Cas-Related Systems and Cas1 Homologs in Asgard Archaea. In: The CRISPR Journal Band 3, Nr. 3, 1. Juni 2020, S. 156–163; doi:10.1089/crispr.2020.0012, ISSN 2573-1599, PMC 7307682 (freier Volltext).
- ↑ a b c d e f g h i j k l Eva F. Cáceres: Genomic and evolutionary exploration of Asgard archaea, Doctoral thesis, Uppsala University, Disciplinary Domain of Science and Technology, Biology, Department of Cell and Molecular Biology, 12. November 2019.
- ↑ a b c d Fraser MacLeod, Gareth S. Kindler, Hon Lun Wong, Ray Chen, Brendan P. Burns: Asgard archaea: Diversity, function, and evolutionary implications in a range of microbiomes. In: AIMS Microbiology. 5. Jahrgang, Nr. 1, 2019, ISSN 2471-1888, S. 48–61, doi:10.3934/microbiol.2019.1.48, PMID 31384702, PMC 6646929 (freier Volltext) – (englisch).
- ↑ Bram Henneman, Clara van Emmerik, Hugo van Ingen, Remus T. Dame: Structure and function of archaeal histones. In: PLOS Genetics. 14. Jahrgang, Nr. 9, September 2018, S. e1007582, doi:10.1371/journal.pgen.1007582, PMID 30212449, PMC 6136690 (freier Volltext), bibcode:2018BpJ...114..446H (englisch).
- ↑ Carl Zimmer: This Strange Microbe May Mark One of Life's Great Leaps - A organism living in ocean muck offers clues to the origins of the complex cells of all animals and plants. In: The New York Times, 15. Januar 2020. Abgerufen am 20. Januar 2020 (englisch).
- ↑ Elizabeth Pennisi: Strange, tentacled microbe may resemble ancestor of complex life. In: Science, 21. Dezember 2022; Nadja Podbregar: Bindeglied der frühen Evolution kultiviert, scinexx.de vom 22. Dezember 2022. Quelle: doi:10.1038/s41586-022-05550-y Actin cytoskeleton and complex cell architecture in an Asgard archaeon.
- ↑ Carl Zimmer: Under the Sea, a Missing Link in the Evolution of Complex Cells. In: The New York Times. 6. Mai 2015, ISSN 0362-4331 (nytimes.com).
- ↑ Jonathan Lambert: Scientists glimpse oddball microbe that could help explain rise of complex life. In: Nature. Band 572, 2019, S. 294 (nature.com).
- ↑ a b
Daniel B. Mills, Richard A. Boyle, Stuart J. Daines, Erik A. Sperling, Davide Pisani, Philip C. J. Donoghue, Timothy M. Lenton: Eukaryogenesis and oxygen in Earth history. In: Nature Ecology & Evolution, 21. April 2022; doi:10.1038/s41559-022-01733-y. Dazu:
- Neue Studie legt nahe, dass eukaryotische Zellen in anoxischer Umgebung entstanden sind. Auf: Aus Der Welt (Max) vom 27. April 2022.
- Eukaryotic Cells Emerged in Anoxic Environment, New Study Suggests. Auf: sci-news vom 27. April 2022. Im Bild: Prometheoarchaeum syntrophicum MK-D1.
- ↑ NCBI Taxonomy Browser: Candidatus Heimdallarchaeota, Details: "Candidatus Heimdallarchaeota" Zaremba-Niedzwiedzka et al. 2017 (phylum).
- ↑ NCBI Taxonomy Browser: Candidatus Helarchaeota (phylum).
- ↑ a b NCBI Taxonomy Browser: „Candidatus Thorarchaeota“ corrig. Seitz et al. 2016 (phylum); homotypic synonym: "Candidatus Thorarchaeia" Seitz et al. 2016.
- ↑ NCBI Genome assembly: ASM1838389v1. Submitted GenBank assembly: GCA_018383895.1, Taxon: Candidatus Jordarchaeia archaeon, Isolate: QZM_A2_3. 17. Mai 2021.
- ↑ LPSN: Phylum "Candidatus Sifarchaeota" Farag et al. 2021.
- ↑ a b Katie D.: Lake Cootharaba | Sunshine Coast's hidden oasis, Escape Parks.
- ↑ a b Lake Cootharaba, auf: Webseite von Noosa Shire, Queensland (noosa.com).
- ↑ a b c d Jiarui Sun, Paul N. Evans, Emma J. Gagen, Ben J. Woodcroft, Brian P. Hedlund, Tanja Woyke, Philip Hugenholtz: Recoding of stop codons expands the metabolic potential of two novel Asgardarchaeota lineages. In: Nature, ISME Commun. Band 1, Nr. 30, 28. Juni 2021; doi:10.1038/s43705-021-00032-0.
- ↑ a b c
Jiawei Zhang, Xiaoyuan Feng, Meng Li, Yang Liu, Min Liu, Li-Jun Hou, Hong-Po Dong: Deep origin of eukaryotes outside Heimdallarchaeia within Asgardarchaeota. In: Nature, Print-ISSN 0028-0836; doi:10.1038/s41586-025-08955-7, PMID 40335687, Online-ISSN 1476-4687, Epub 7. Mai 2025 (englisch). Siehe insbes. Fig. 3 und Ext. Data Fig. 1. Dazu:
- Eukaryotes’ Deep Origin Outside Heimdallarchaeia. Auf: Bioengineer (bioengineer.org) vom 8. Mai 2025.
- ↑ a b c d e f g h i
Ruize Xie, Yinzhao Wang, Danyue Huang, Jialin Hou, Liuyang Li, Haining Hu, Xiaoxiao Zhao, Fengping Wang: Expanding Asgard members in the domain of Archaea sheds new light on the origin of eukaryotes. In: Science China Life Sciences, Band 65, S. 818–829, April 2022; doi:10.1007/s11427-021-1969-6, PMID 34378142, Epub 6. August 2021. Dazu:
- Njordarchaeota, a new candidate for a sister group to eukaryotes. Auf: EurekAlert vom 20. Oktober 2021.
- ↑ a b LPSN: Phylum "Candidatus Njordarchaeota" Xie et al. 2022.
- ↑ NCBI Taxonomy Browser: Candidatus Gefionarchaeota.
- ↑ NCBI Taxonomy Browser: Candidatus Friggarchaeota.
- ↑ a b NCBI Taxonomy Browser: "Candidatus Idunnarchaeota" Caceres 2019 (phylum)
- ↑ a b LPSN: Phylum "Candidatus Freyrarchaeota" Xie et al. 2022.
- ↑ NCBI: "Candidatus Freyarchaeota" Caceres 2019 (phylum). Wie man sieht, wirft das NCBI Frey[a] und Freyr zusammen. Das LPSN folgt dem nicht.
- ↑ NCBI Taxonomy Browser: "Candidatus Jordarchaeia" Sun et al. 2021 (class)
- ↑ a b LPSN: Phylum "Candidatus Sigynarchaeota" Xie et al. 2022.
- ↑ DRA: Sample Candidatus UAP3 archaeon UBA460 ( vom 1. Mai 2021 im Internet Archive)
- ↑ Alla L. Lapidus, Anton I. Korobeynikov: Metagenomic Data Assembly – The Way of Decoding Unknown Microorganisms. In: Frontiers in Microbiology Band 12, 23. März 2021, S. 653, ISSN 1664-302X; doi:10.3389/fmicb.2021.613791 (englisch).
- ↑ Nina Dombrowski, Tom A. Williams, Jiarui Sun, Benjamin J. Woodcroft, Jun-Hoe Lee, Bui Quang Minh, Christian Rinke, Anja Spang: Undinarchaeota illuminate DPANN phylogeny and the impact of gene transfer on archaeal evolution, in: Nature Communications, Band 11, Nr. 3939, 7. August 2020, doi:10.1038/s41467-020-17408-w.
- ↑ Taxonomicon: Paraphyletic taxon: Candidate superphylum Asgard.
- ↑ Donovan H. Parks, Christian Rinke, Maria Chuvochina, Pierre-Alain Chaumeil, Ben J. Woodcroft, Paul N. Evans, Philip Hugenholtz, Gene W. Tyson: Recovery of nearly 8,000 metagenome-assembled genomes substantially expands the tree of life. In: Nature Microbiology, Band 2, S. 1533–1542, 11. September 2017; doi:10.1038/s41564-017-0012-7, mit Korrektur vom 12. Dezember 2017, doi:10.1038/s41564-017-0083-5. Siehe insbesondere Supplement Tabelle 14.
- ↑ NCBI Taxonomy Browser: archaeon UBA460 (species).
- ↑ GTDB: UBA460 (genus) – Phylogenetischer Baum; (species) – Details.
- ↑ a b
Ibrahim F. Farag, Rui Zhao, Jennifer F. Biddle: “Sifarchaeota,” a Novel Asgard Phylum from Costa Rican Sediment Capable of Polysaccharide Degradation and Anaerobic Methylotrophy, in: ASM Appl Environ Microbiol 87:e02584-20, Epub 13. April 2021, doi:10.1128/AEM.02584-20, PMID 33608286.
Preprint: “Sifarchaeota” a novel Asgard phylum capable of polysaccharide degradation and anaerobic methylotrophy, auf: CSH bioRχiv vom 15. Oktober 2020, doi:10.1101/2020.10.14.339440, ResearchGate, PDF (14. Oktober 2020). Siehe insbes. Fig. 1 ( vom 3. Mai 2021 im Internet Archive) (Kladogramm). - ↑ a b Purificación López-García, David Moreira: Eukaryogenesis, a syntrophy affair. In: Nature Microbiology. 4. Jahrgang, Nr. 7, 1. Juli 2019, ISSN 2058-5276, S. 1068–1070, doi:10.1038/s41564-019-0495-5, PMID 31222170, PMC 6684364 (freier Volltext) – (englisch).
- ↑ Charlotte D. Vavourakis, Maliheh Mehrshad, Cherel Balkema, Rutger van Hall, Adrian-Ştefan Andrei, Rohit Ghai, Dimitry Y. Sorokin, Gerard Muyzer: Metagenomes and metatranscriptomes shed new light on the microbial-mediated sulfur cycle in a Siberian soda lake. In: BMC Biology, Band 17, Nr. 69, 22. August 2019; doi:10.1186/s12915-019-0688-7, PMID 31438955, PMC 6704655 (freier Volltext). Siehe Additional File 4 (xlsx).
- ↑ a b Fabai Wu, Daan R. Speth, Alon Philosof, Antoine Crémière, Aditi Narayanan, Roman A. Barco, Stephanie A. Connon, Jan P. Amend, Igor A. Antoshechkin, Victoria J. Orphan: Unique mobile elements and scalable gene flow at the prokaryote–eukaryote boundary revealed by circularized Asgard archaea genomes. In: Nature Microbiology, Band 7, S. 200–212, 13. Januar 2022; doi:10.1038/s41564-021-01039-y, PMID 35027677, PMC 8813620 (freier Volltext).
- ↑ a b
Genome Taxonomy Database (GTDB) Release 10-RS226:
- About. In: Genome Taxonomy Database. Abgerufen am 1. Mai 2025 (englisch).
- Tree Data (ar53_r226.sp_labels). In: GTDB. Abgerufen am 1. Mai 2025.
- Taxon History. In: GTDB. Abgerufen am 1. Mai 2025 (englisch).
- ↑ NCBI Taxonomy Browser: "Candidatus Hodarchaeum mangrovi" Liu et al. 2020, Nucleotide: MAG: Candidatus Hodarchaeum mangrovi isolate FT_5_011, …
- ↑ Futian Mangrove Nature Reserve. Auf Google Maps.
- ↑ Gregory P. Fournier, Anthony M. Poole: A Briefly Argued Case That Asgard Archaea Are Part of the Eukaryote Tree. In: Front. Microbiol. 9. Jahrgang, 2018, S. 1896, doi:10.3389/fmicb.2018.01896, PMID 30158917, PMC 6104171 (freier Volltext) – (englisch).
- ↑ Thomas Cavalier-Smith, Ema E-Yung Chao: Multidomain ribosomal protein trees and the planctobacterial origin of neomura (eukaryotes, archaebacteria), in: Protoplasma Band 257, S. 621–753, 3. Januar 2020, doi:10.1007/s00709-019-01442-7.
- ↑ Thomas Cavalier-Smith: The phagotrophic origin of eukaryotes and phylogenetic classification of Protozoa, in: Int. J. Syst. Evol. Microbiol. Band 52, Nr. Pt2, März 2002, S. 297–354; doi:10.1099/00207713-52-2-297, PMID 11931142.
- ↑ NCBI: Candidatus Baldrarchaeota (phylum)
- ↑ NCBI: Candidatus Borrarchaeota (phylum)
- ↑ a b
NCBI Taxonomy Browser: Candidatus Gerdarchaeota (phylum). Details: Candidatus Gerdarchaeota archaeon (species), MAGs:
- Nucleotide: MAG: Candidatus Gerdarchaeota archaeon isolate …
- ↑ LPSN: Phylum "Candidatus Heimdallarchaeota"
- ↑ NCBI: Candidatus Heimdallarchaeota (phylum)
- ↑ Kiley W. Seitz, Nina Dombrowski, Laura Eme, Anja Spang, Jonathan Lombard, Jessica R. Sieber, Andreas P. Teske, Thijs J. G. Ettema, Brett J. Baker: Asgard archaea capable of anaerobic hydrocarbon cycling, in: Nature Communications, 23. April 2019, doi:10.1038/s41467-019-09364-x.
- ↑ NCBI: Candidatus Helarchaeota (phylum)
- ↑ LPSN: Phylum "Candidatus Helarchaeota"
- ↑ NCBI: Candidatus Hermodarchaeota (phylum)
- ↑ NCBI: Candidatus Hodarchaeota (phylum)
- ↑ NCBI: Candidatus Kariarchaeota (phylum)
- ↑ LPSN: Phylum "Candidatus Lokiarchaeota"
- ↑ NCBI: Candidatus Lokiarchaeota
- ↑ LPSN: Phylum "Candidatus Odinarchaeota"
- ↑ NCBI: Candidatus Odinarchaeota (phylum)
- ↑ NCBI: Candidatus Wukongarchaeota (phylum)
- ↑ a b
Tomas Alarcón-Schumacher, Susanne Erdmann: A trove of Asgard archaeal viruses. In: Nature Microbiology. Band 7, 27. Juni 2022, S. 931–932; doi:10.1038/s41564-022-01148-2. Dazu:
- Nicoletta Lanese: These Newly Discovered Viruses May Have Shaped The Rise of Complex Life on Earth. Auf: sciencealert vom 2. Juli 2022. Quelle: LiveScience.
- ↑
Ian M. Rambo, Marguerite V. Langwig, Pedro Leão, Valerie De Anda. Brett J. Baker: Genomes of six viruses that infect Asgard archaea from deep-sea sediments. In: Nature Microbiology. S. 953–961, Juli 2022; doi:10.1038/s41564-022-01150-8, Volltext (E-Book), Epub 27. Juni 2022. Dazu:
- Ermöglichten Viren komplexes Leben? Auf: wissenschaft.de vom 27. Juni 2022.
- Tiffany Winfrey: New Microbiology Research Provides Evidence Virus Plays a Role in Evolution of Asgard Archaeans. Auf: The Science Times, 27. Juni 2022. Anm.: Asgard ist keine class of bacteria, richtiger wäre etwa group of archaea.
- Incredible Virus Discovery Offers Clues About the Origins of Complex Life. Auf: SciTechDaily vom 27. Juni 2022.
- Mysterious Asgard Archaea-Infecting Viruses Discovered. Auf: sci-news.com vom 30. Juni 2022.
- Ian M. Rambo, Valerie de Anda, Marguerite V. Langwig, Brett J. Baker: Unique viruses that infect Archaea related to eukaryotes. Preprint auf: CSH bioRxiv, 29. Juli 2021; doi:10.1101/2021.07.29.454249, ResearchGate.
- ↑ NCBI: Lokiarchaeota virus Fenrir Meg22_1012 (species)
- ↑ NCBI: Lokiarchaeota virus Fenrir Meg22_1012 (species)
- ↑ NCBI: Mycobacterium phage Nidhogg (species).
- ↑ NCBI: Helarchaeota virus Nidhogg Meg22_1012 (species)
- ↑ NCBI: Helarchaeota virus Nidhogg Meg22_1012 (species)
- ↑ NCBI: Lokiarchaeota virus Ratatoskr Meg22_1012 (species)
- ↑ NCBI: Lokiarchaeota virus Skoll Meg22_1214 (species)
- ↑
Daniel Tamarit, Eva F. Cáceres, Mart Krupovic, Reindert Nijland, Laura Eme, Nicholas P. Robinson, Thijs J. G. Ettema: A closed Candidatus Odinarchaeum chromosome exposes Asgard archaeal viruses. In: Nature Microbiology. Band 7, 27. Juni 2022; doi:10.1038/s41564-022-01122-y. Dazu:
- HAL. Preprint. 6. Oktober 2021.
- ↑
Sofia Medvedeva, Jiarui Sun, Natalya Yutin, Eugene V. Koonin, Takuro Nunoura, Christian Rinke, Mart Krupovic: Three families of Asgard archaeal viruses identified in metagenome-assembled genomes. In: Nature Microbiology. Band 7, S. 962–973962–973, 27. Juni 2022, doi:10.1038/s41564-022-01144-6, PMID 35760839, Volltext. Dazu:
- Viruses of Asgard archaea. Preprint auf: CSH bioRxiv, 30. Juli 2021; doi:10.1101/2021.07.29.453957.
- ↑ NCBI Taxonomy Browser: Lokiarchaeota virus WyrdV1 (species).