Pflanzliche Immunantwort
Die pflanzliche Immunantwort ist ein natürlicher Schutzmechanismus von Pflanzen, analog zur Immunantwort bei Tieren. Da Pflanzen keine durch Antikörper vermittelte adaptive Immunität haben, zählt die pflanzliche Immunantwort vollständig zur angeborenen Immunantwort. Dass diese Zuordnung auch evolutionsbiologisch korrekt ist, zeigt das Vorhandensein pathogenassoziierter molekularer Muster bei Pflanzen und Tieren.
Pflanzen haben im Gegensatz zu Wirbeltieren keine Antikörper. Dennoch sind sie in der Regel gegenüber Krankheiten, die durch Pathogene wie Bakterien, Nematoden, Pilze oder Viren verursacht werden können, resistent. Verschiedene Abwehrmechanismen kommen hierbei zum Einsatz. Saponine, eine Gruppe von Triterpenen, werden von den Pflanzen bereits vor Infektionen gebildet. Sie bieten Schutz vor Pilzen, indem sie an die Sterole in deren Plasmamembran binden und diese dadurch zerstören. Andere Abwehrmaßnahmen setzen erst während der Infektion ein.
Induzierte Abwehr
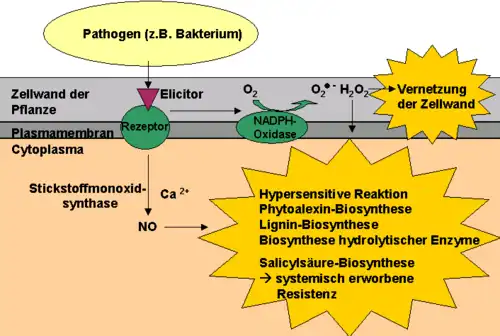
Im Rahmen der induzierten Abwehr binden Moleküle aus dem Pathogen (z. B. Proteine, Sterole, Polysaccharid-Fragmente) an ein Rezeptorprotein in der pflanzlichen Plasmamembran. Durch die Bindung eines solchen Elicitors wird der Rezeptor aktiviert und setzt zwei Signalkaskaden in Gang. Zum einen wird die ebenfalls in der Plasmamembran befindliche NADPH-Oxidase aktiviert, so dass Luftsauerstoff zu Superoxidanionen reduziert wird. Superoxidanionen werden in der Folge zu Hydroxyl-Radikalen und Wasserstoffperoxid umgesetzt. Diese drei reaktiven Sauerstoffspezies starten radikalische Kettenreaktionen mit organischen Molekülen, aus denen Lipidperoxidation, Enzym-Inaktivierung und Abbau von Nukleinsäuren resultieren, wovon Pathogen und befallene Pflanzenzellen betroffen sind. Die zweite vom pflanzlichen Rezeptor eingeleitete Signalkaskade führt zur calciumabhängigen Aktivierung der Stickstoffmonoxid-Synthase und somit zur Bildung von Stickstoffmonoxid, das gemeinsam mit Wasserstoffperoxid (H2O2) folgende pflanzenart- und pathogenabhängige Abwehrmechanismen auslösen kann:
Hypersensitive Reaktion
Im Rahmen der hypersensitiven Reaktion sterben beschleunigt Zellen ab, die die Infektionsstelle umgeben, so dass dem Pathogen mit dem Entzug von Nährstoffen keine Ausbreitungsmöglichkeit mehr gegeben wird. Die abgestorbenen Zellen sind makroskopisch als nekrotische Läsionen erkennbar. Die hypersensitive Reaktion ist eine Form des programmierten Zelltods, der u. a. zur gesteigerten Bildung von Nukleasen und Proteasen führt, die Nukleinsäuren bzw. Proteine hydrolytisch abbauen. Viele Pflanzenarten reagieren auf Pathogenbefall mit der Synthese von Lignin oder Callose, die eine physikalische Barriere gegen die Pathogen-Ausbreitung darstellen. Eine ähnliche Reaktion ist eine durch Wasserstoffperoxid bedingte Quervernetzung prolinreicher Proteine in der Zellwand, die zu deren Verdichtung führt. Infizierende Pilze können auch mit der Synthese von Chitinasen abgewehrt werden, wobei die Enzyme Chitin als Bestandteil der pilzlichen Zellwand hydrolysieren. Zusätzlich kann die Expression von Glukanasen oder anderer pilzangreifender Enzyme gesteigert werden. Die Synthese der antimikrobiell wirkenden Phytoalexinen ist an verschiedenen Pflanzen eingehend erforscht worden. Bei diesen Sekundärmetaboliten handelt es sich um unterschiedliche Substanzen wie z. B. Isoflavone im Falle von Leguminosen oder Sesquiterpene im Falle von Nachtschattengewächsen. Nach einer Pathogen-Infektion kann die Pflanze gegenüber einer Bandbreite verschiedener Pathogene eine erhöhte Resistenz aufweisen, und zwar auch an nicht zuvor befallenen Stellen. Diese systemisch erworbene Resistenz basiert u. a. auf den bereits angesprochenen hydrolytischen Enzymen. Salicylsäure führt hierbei wahrscheinlich zur Synthese eines Lipids (oder Lipidderivats). Das Signalmolekül wird im Phloem transportiert und bedingt die Resistenz in noch nicht befallenen Pflanzenteilen. Die methylierte Form der Salicylsäure (Methylsalicylat) ist flüchtig, so dass eine erhöhte Resistenz auch in benachbarten Pflanzen induziert werden kann.
Zwei Ebenen der pflanzlichen Immunität
Pflanzen besitzen ein hochentwickeltes, mehrstufiges Immunsystem, das sie gegen eine Vielzahl von Krankheitserregern wie Bakterien, Pilze, Viren und Nematoden schützt. Dieses System lässt sich in zwei Hauptkomponenten unterteilen: Pattern-Triggered Immunity (PTI) und Effector-Triggered Immunity (ETI). Beide Systeme arbeiten zusammen, um Pflanzen gegen eine Vielzahl von Pathogenen zu schützen.
Pattern-Triggered Immunity (PTI)
Die Pattern-Triggered Immunity stellt die erste Verteidigungslinie der pflanzlichen Immunantwort dar. Sie basiert auf der Erkennung konservierter molekularer Strukturen, die bei vielen Mikroorganismen vorkommen. Diese sogenannten Pathogen-Associated Molecular Patterns (PAMPs) oder Microbe-Associated Molecular Patterns (MAMPs) umfassen unter anderem Flagellin (ein Bestandteil bakterieller Geißeln), Chitin (ein Strukturpolysaccharid in Pilzzellwänden) und Lipopolysaccharide (Bestandteile der äußeren Membran Gram-negativer Bakterien).
Diese PAMPs werden von Pattern Recognition Receptors (PRRs) erkannt, die sich auf der Plasmamembran pflanzlicher Zellen befinden. Die Bindung eines PAMPs an einen PRR löst eine intrazelluläre Signalkaskade aus, die zur Aktivierung verschiedener Abwehrmechanismen führt. Dazu gehören die Produktion reaktiver Sauerstoffspezies (ROS), die Einlagerung von Callose zur Verstärkung der Zellwand, die Aktivierung von Mitogen-aktivierten Proteinkinasen (MAP-Kinasen) sowie die Transkription von Genen, die antimikrobielle Proteine und Enzyme codieren. Diese Reaktionen dienen dazu, die Ausbreitung des Pathogens zu begrenzen und eine systemische Immunantwort vorzubereiten.[1]
Effector-Triggered Immunity (ETI)
Einige Pathogene haben im Laufe der Koevolution mit Pflanzen spezifische Effektoren entwickelt – Proteine, die in pflanzliche Zellen eingeschleust werden, um die PTI gezielt zu unterdrücken und die pflanzliche Abwehr zu umgehen. Als Reaktion darauf haben Pflanzen Resistenzgene (R-Gene) entwickelt, deren Genprodukte – meist NLR-Proteine (Nucleotide-binding Leucine-rich Repeat Proteins) – in der Lage sind, diese Effektoren direkt oder indirekt zu erkennen.
Die durch Effektorerkennung ausgelöste Effector-Triggered Immunity ist in der Regel stärker und spezifischer als PTI. Sie führt häufig zu einer Hypersensitiven Reaktion (HR), bei der infizierte Zellen gezielt absterben, um die Ausbreitung des Pathogens zu verhindern. Darüber hinaus kann ETI eine Systemische Erworbene Resistenz (SAR) induzieren, die eine langanhaltende Immunität in nicht infizierten Pflanzenteilen vermittelt. Auch die Expression von Abwehrgenen wird im Rahmen der ETI deutlich verstärkt, was zu einer erhöhten Produktion antimikrobieller Substanzen führt.[2]
Wirkungsweise der NLR-Proteine
NLR-Proteine (Nucleotide-binding Leucine-rich Repeat Proteins) sind zentrale Komponenten der ETI und fungieren als intrazelluläre Immunrezeptoren. Sie bestehen typischerweise aus drei funktionellen Domänen:
- N-terminaler Bereich (z. B. TIR- oder CC-Domäne): beteiligt an der Signalweiterleitung nach Aktivierung.
- NB-ARC-Domäne (Nucleotide-binding domain): bindet und hydrolysiert ATP oder ADP, was zur Konformationsänderung des Proteins führt.
- Leucinreiche Wiederholungen (LRR): verantwortlich für die Erkennung spezifischer Effektoren oder effektorinduzierter Veränderungen zellulärer Zielproteine.
Die Aktivierung eines NLR-Proteins erfolgt entweder durch direkte Bindung eines Effektors an die LRR-Domäne oder durch indirekte Erkennung von Effektorwirkungen auf Wirtsproteine (sogenanntes „Guard-Modell“ oder „Decoy-Modell“). Nach der Erkennung verändert das NLR-Protein seine Konformation, oligomerisiert und bildet häufig sogenannte Resistosome – Proteinkomplexe, die als Signalplattformen dienen und zelluläre Abwehrreaktionen auslösen, einschließlich der hypersensitiven Reaktion.[3]
Zusammenspiel von PTI und ETI
PTI und ETI sind keine isolierten Systeme, sondern wirken synergistisch. Während PTI eine breit angelegte, basal wirksame Immunantwort darstellt, bietet ETI eine gezielte und oft intensivere Reaktion auf spezifische Pathogenstämme. Beide Systeme aktivieren ähnliche Abwehrmechanismen, unterscheiden sich jedoch in der Art der Erkennung und in der Stärke der ausgelösten Reaktionen. Die Interaktion zwischen Pathogenen und Pflanzen ist durch ein ständiges evolutionäres Wettrüsten geprägt: Pathogene entwickeln neue Effektoren, um die pflanzliche Abwehr zu umgehen, während Pflanzen neue R-Gene entwickeln, um diese Effektoren zu erkennen.
Siehe auch
Literatur
- Jonathan D. G. Jones, Jeffery L. Dangl: The plant immune system. In: Nature. Bd. 444, Nr. 7117, November 2006, S. 323–329, doi:10.1038/nature05286, PMID 17108957 (Review), Digitalisat (PDF; 517 kB) ( vom 17. Juni 2010 im Internet Archive).
- Bob B. Buchanan, Wilhelm Gruissem, Russell L. Jones: Biochemistry & molecular biology of plants. American Society of Plant Physiologists, Rockville MD 2000, ISBN 0-943088-37-2.
- Leendert C. van Loon (Hrsg.): Plant Innate Immunity (= Advances in Botanical Research. Bd. 51). Academic Press, Amsterdam u. a. 2009, ISBN 978-0-12-374834-8.
- Peter Schopfer, Axel Brennecke: Pflanzenphysiologie. Elsevier – Spektrum Akademischer Verlag, Heidelberg 2006, ISBN 3-8274-1561-6.
- Lincoln Taiz, Eduardo Zeiger: Plant physiology. 4th edition. Sinauer, Sunderland MA 2006, ISBN 0-87893-856-7.
Einzelnachweise
- ↑ Minhang Yuan, Bruno Pok Man Ngou, Pingtao Ding, Xiu-Fang Xin: PTI-ETI crosstalk: an integrative view of plant immunity. In: Current Opinion in Plant Biology. Band 62, August 2021, S. 102030, doi:10.1016/j.pbi.2021.102030 (elsevier.com [abgerufen am 22. Mai 2025]).
- ↑ Minhang Yuan, Bruno Pok Man Ngou, Pingtao Ding, Xiu-Fang Xin: PTI-ETI crosstalk: an integrative view of plant immunity. In: Current Opinion in Plant Biology. Band 62, August 2021, S. 102030, doi:10.1016/j.pbi.2021.102030 (elsevier.com [abgerufen am 22. Mai 2025]).
- ↑ Isabel M. L. Saur, Ralph Panstruga, Paul Schulze-Lefert: NOD-like receptor-mediated plant immunity: from structure to cell death. In: Nature Reviews Immunology. Band 21, Nr. 5, Mai 2021, ISSN 1474-1741, S. 305–318, doi:10.1038/s41577-020-00473-z (nature.com [abgerufen am 27. Mai 2025]).