Orlajensenviridae
| Orlajensenviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
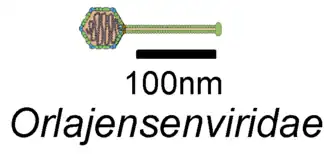
Schemazeichnung (Virion im Querschnitt) | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Orlajensenviridae | ||||||||||||||
| Links | ||||||||||||||
|
Orlajensenviridae ist eine im März 2022 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp C (Podoviren). Ihre Wirte sind Bakterien der Gattung Microbakterium,[1] was sie als Bakteriophagen klassifiziert.[2] Die Familie besteht monotypisch aus der einzigen Unterfamilie Pelczarvirinae.[3][4]
Beschreibung
Mit der Einrichtung der Familie Orlajensenviridae hat das ICTV die Klassifizierung des Microbacterium-Phagen-Clusters EE in der Actinobacteriophage Database (PhagesDB) formalisiert. ViPTree-Analysen hatten gezeigt, dass diese Gruppe von Viren sehr tief verwurzelt ist, sodass der taxonomische Rang einer Familie angebracht erschien. Innerhalb dieser gehören alle drei ursprünglichen Gattungen (Gründungsgattungen) zu einer einzigen Unterfamilie, deren Mitglieder im Durchschnitt ein Genom mit einer Länge von 17,43 kbp (Kilobasenpaare) haben bei einem GC-Gehalt von durchschnittlich 68,43 mol%G. Das Genom kodiert für (fast immer genau) 26 Proteine, aber keine tRNAs. Mithilfe von CoreGenes 5.0[5] wurden 20 gemeinsame Homologe dieser Gruppe von Phagen (Übereinstimmung 76,9 %) gefunden. Zu diesen Homologen gehören: Terminase der großen Untereinheit,[6] Portalprotein, Hauptkapsidprotein (major capsid protein, MCP) und Protease-Fusionsprotein, Haupt-Schwanz-Protein (major tail protein, MTP), zwei kleinere Schwanz-Proteine (minor tail proteins, mTPs), zwei Chaperone für die Montage von der Schwänze, drei DNA-bindende Proteine, etc.[2]
Der lytische Microbacterium-Phage PaoPu wurde 2016 von Acacia Eleri aus dem Boden von Germantown (Maryland), USA, isoliert, wobei als Wirt der Stamm NRRL B-24224 von Microbacterium foliorum diente. Das Genom dieses Phagen ist mit einem 9 nt (Nukleotide) langen kohäsiven 3'-Terminus (Klebeende, „Sticky End“) abgeschlossen (CCCGCCCCA). Er wird in der Actinobacteriophage Database (PhagesDB) – wie alle anderen Mitglieder dieser Familie – ins Cluster EE klassifiziert.[7][2]
Die erwähnte ViPTree-Analyse legte nahe, auch den Microbacterium-Phagen Min1 (Spezies Minunavirus Min1)[8][9][10] der Familie hinzuzufügen. Da aber seine Genomgröße fast dreimal so groß ist wie die der hier beschriebenen Phagen, wurde dies wieder fallengelassen. In der PhagesDB wird er entsprechend als Singleton geführt (Stand April 2025).[2]
Genomeigenschaften einiger Phagen der Orlajensenviridae:[2]
Phagenname Zugriffsnr. Gr. (Kbp) GC (%) Proteine Microbacterium phage PaoPu MH045561.1 17.36 68.5 25 Microbacterium phage Azizam MN585990.1 17.39 68.7 25 Microbacterium phage Bri160 MT521990.1 17.44 68.6 25 Microbacterium phage Kaijohn MN369745.1 17.43 68.8 26 Microbacterium phage Nobel MT310864.1 17.05 69.0 25 Microbacterium phage Noelani MH399783.1 17.35 68.2 25 Microbacterium phage PoRanda MT498065.1 17.51 69.1 25 Microbacterium phage Quaker MH371111.1 17.45 68.6 25 Microbacterium phage Scamander MH576963.1 17.45 68.7 25
Systematik
Die Systematik der Familie Orlajensenviridae ist nach International Committee on Taxonomy of Viruses (ICTV) – MSL#40v1; erstgenannt sind die exemplarischen Phagen der jeweiligen Spezies; in Klammern finden sich die Zugriffsnummern – mit Stand 16. April 2025 wie folgt:[3][11][2][12]
Familie Orlajensenviridae (PhagesDB-Cluster EE)
- Unterfamilie Pelczarvirinae
- Gattung Bonaevitaevirus
- Gattung Efekovirus
- Gattung Paopuvirus
- Spezies Paopuvirus azizam (im Vorschlag auch Paopuvirus Azizam),[B. 3] mit
- Microbacterium-Phage Azizam (MN585990)
- Microbacterium-Phage JRok (MT639647.1)
- Spezies Paopuvirus bri160, mit
- Microbacterium-Phage Bri160 (MT521990)[15]
- Spezies Paopuvirus kaijohn, mit
- Microbacterium-Phage Kaijohn (MN369745)
- Spezies Paopuvirus nobel (im Vorschlag auch Paopuvirus Nobel),[B. 4] mit
- Microbacterium-Phage Nobel (MT310864)
- Microbacterium-Phage Bullzi2019 (MT498067.1)
- Microbacterium-Phage HarperAnne (MN586010.1)
- Microbacterium-Phage Miaurora (MH779512.1)
- Microbacterium-Phage TinyTruffula (MT498047.1)
- Spezies Paopuvirus noelani, mit
- Microbacterium-Phage Noelani (MH399783)
- Spezies Paopuvirus paopu (im Vorschlag auch Paopuvirus PaoPu),[B. 5] mit
- Microbacterium-Phage PaoPu (MH045561)[16]
- Microbacterium-Phage Bradley2 (MN284892.1)
- Microbacterium-Phage Dongwon (MH744416.1)
- Microbacterium-Phage Hernandez44 (MT498046.1)
- Microbacterium-Phage Hulk (MK878899.1)
- Microbacterium-Phage Jahseh (MT498051.1)
- Microbacterium-Phage Minima (MH65118.1)
- Microbacterium-Phage YertPhresh (MT639642.1)
- Spezies Paopuvirus poranda,[B. 6] mit
- Microbacterium-Phage PoRanda (MT498065)
- Spezies Paopuvirus quaker (im Vorschlag auch Paopuvirus Quaker),[B. 7] mit
- Microbacterium-Phage Quaker (MH371111)
- Microbacterium-Phage Belthelas (MK801730.1)
- Microbacterium-Phage KayPaulus (MH371118.1)
- Microbacterium-Phage Livingwater (MT498040.1)
- Microbacterium-Phage McShie (MT316458.1)
- Spezies Paopuvirus Scamander,[B. 8] mit
- Microbacterium-Phage Scamander (MH576963)
- Microbacterium-Phage BurtonThePup (MH045557.1)
- Microbacterium-Phage Ciel (MN010759.1)
- Microbacterium-Phage Danno (MT316462.1)
- Microbacterium-Phage LaviMo (MK894434.1)
- Microbacterium-Phage Luxx (MT498038.1)
- Microbacterium-Phage Majesty (MT776807.1)
- Microbacterium-Phage Otwor (MT316459.1)
- Microbacterium-Phage Owens (MN428064.1)
- Microbacterium-Phage Rhysand (MK620897.1)
- Microbacterium-Phage Scrunchy (MN586008.1)
- Microbacterium-Phage Slentz (MN010755.1)
- Microbacterium-Phage TeddyBoy (MN428062.1)
- Microbacterium-Phage TimoTea (MK524502.1)
- Microbacterium-Phage Vanisius (MN329679.1)
- Microbacterium-Phage VitulaEligans (MH371124.1)
- Spezies Paopuvirus azizam (im Vorschlag auch Paopuvirus Azizam),[B. 3] mit
Namensherkunft (Etymologie)
Mit dem Familiennamen Orlajensenviridae wird der dänischen Chemiker und Molkereibakteriologe Sigurd Orla-Jensen[17] geehrt. Er war maßgeblich für die Isolierung und Taxonomie der Bakterien der Gattung Microbacterium[1] verantwortlich. Darüber hinaus hat er die Gattung Propionibacterium und die das Joghurt-Bakterium Lactobacillus delbrueckii subsp. bulgaricus (unter dem Namen Thermobacterium bulgaricum) erstbeschrieben und war Mitglied der Königlich Dänischen Akademie der Wissenschaften. Der Suffix ‚-viridae‘ kennzeichnet Virusfamilien.[4][2]
Die Unterfamilie Pelczarvirinae ist nach Michael Joseph Pelczar[18] benannt, der Suffix ‚-virinae‘ kennzeichnet Virusunterfamilien.[19]
Weblinks
Bildergalerie: EM-Aufnahmen einiger Virionen aus dem Cluster EE in der PhagesDB:
Einzelnachweise
- ↑ a b LPSN: Genus Microbacterium Orla-Jensen 1919.
- ↑ a b c d e f g Liliana Cristina Moraru, Dann Turner, Igor Tolstoy, Evelien M. Adriaenssens, Andrew M. Kropinski: Createo one new family (Orlajensenviridae) including three new genera (Caudoviricetes). Vorschlag 2021.060B (zip:docx), Filelist.
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Family: Orlajensenviridae .
- ↑ CoreGenes 5.0 (ngrok.io).
- ↑ UniProt: M9NUS7 - Terminase, large subunit.
- ↑ PhagesDB: Microbacterium Phage PaoPu.
- ↑ PhagesDB: Microbacterium phage Min1, Singleton.
- ↑ NCBI Taxonomy Browser: Microbacterium phage Min1.
- ↑ ICTV: Species: Minunavirus Min1.
- ↑ ICTV: Master Species Lists (MSL).
- ↑ NCBI Taxonomy Browser: Orlajensenviridae, Details: Orlajensenviridae (family).
- ↑ NCBI: Microbacterium-Phage BonaeVitae (species).
- ↑ NCBI: Microbacterium-Phage Efeko (species).
- ↑ NCBI: Microbacterium-Phage Bri160 (species).
- ↑ NCBI: Microbacterium-Phage PaoPu (species).
- ↑ Wikidata: Sigurd Orla-Jensen (Q12335261).
- ↑ Michael Joseph Pelczar, Jr. (poles.org).
- ↑ Dann Turner, Andrey N. Shkoporov, Evelien M. Adriaenssens et al.: Abolishment of morphology-based taxa and change to binomial species names: 2022 taxonomy update of the ICTV bacterial viruses subcommittee. In: Archives of Virology, Nr. 2, 01. Februar 2023; doi:10.1007/s00705-022-05694-2, PMC 9868039 (freier Volltext), PMID 36683075 (englisch).