Obscuriviridae
| Obscuriviridae[1][2] | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
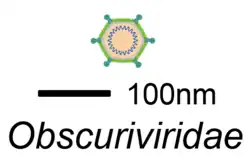
Schemazeichnung eines | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Obscuriviridae[1][2] | ||||||||||||||
| Links | ||||||||||||||
|
Obscuriviridae ist eine Familie von Viren, deren Wirte Bakterien der marinen Gattung Cellulophaga (Familie Flavobacteriaceae) sind, was sie als Bakteriophagen und speziell Flavophagen klassifiziert.
Beschreibung
Die Familie Obscuriviridae wurde vom International Committee on Taxonomy of Viruses (ICTV) im März 2025 eingerichtet, um einigen neuen Vier, die als Bakteriophagen Bakterien der Gattung Cellulophaga infizieren, in die offizielle Virus-Taxonomie aufzunehmen. Bei den betreffenden Phagen handelt es sich um Omtje_1, dessen Isolierung und Charakterisierung von Nina Bartlau et al. 2021/2022 beschrieben wurde, sowie einige teilweise kultivierte Verwandte.[3][3][4]
Im Vorschlag von Nina Bartlau an das ICTV werden die Vertreter der neuen Familie Obscuriviridae auch als Flavophagen bezeichnet, was daher rührt, dass die Wirtsgattung Cellulophaga der Familie Flavobacteriaceae in der Ordnung Flavobacteriales angehört.[4] Diese Bezeichnung ist nicht zu verwechseln mit den Flaviviren, einer umgangssprachlichen Bezeichnung für die ssRNA-Viren der Familie Flaviviridae (und speziell der Gattung Orthoflavivirus – früher Flavivirus), zu der u. a. der Erreger des Gelbfiebers gehört.
Die innere Systematik der Familie basiert auf der Analyse nach sechs verschiedenen Methoden:
- Nukleotid-basierte Ähnlichkeiten zwischen der verschiedenen Genomen, berechnet per VIRIDIC Web-Service[5]
- Protein-basiertes hierarchische Cluster-Analyse, berechnet mit VirClust[6]
- Aminosäure-basierte phylogenetische Bäume basierend auf dem Ganzgenom, berechnet mit Viral Proteomic Tree (ViPTree)
- Aminosäure-basierte phylogenetische Bäume basierend auf dem Ganzgenom, berechnet mit der Virus Classification and Tree Building Online Resource (VICTOR)
- Kernprotein-basierte Bäume mit IQ-Trees
- Sequenzverwandtschaft nach GRAViTy.
Die innerfamiliäre Struktur und die Beziehungen wurden wurde mit VirClust berechnet, ein Alignement mit MUSCLE (v3.8.425) vorgenommen.
Gattung Omtjevirus

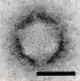
Zusammen mit dem Cellulophaga-Stamm HaHaR_3_176 (alias DSM 111152) wurde 2017 bis 2018 vor dem Dünehafen der Insel Düne (Helgoland) ein Satz von fünf eng verwandten Phagen aus der Nordsee isoliert, die als Omtje_1 bis Omtje_5 bezeichnet werden und untereinander eine Sequenzidentität von mehr als 99,9 % besitzen. Sie werden alle zur Spezies Omtjevirus Omtje klassifiziert, mit Omtje_1 als exemplarischen Repräsentanten. Ihre Viruspartikel (Virionen) haben einen Kapsiddurchmesser von 52,3 ± 4,6 nm, im Gegensatz zu den Bakteriophagen der Klasse Caudoviricetes fehlt ihnen der „Schwanz“. Dünnschnitte der Virionen zeigen eine mögliche Lipidschicht im Inneren des Kapsids. Analyse und Abbau des DNA-Genoms ergab, dass es sich um ssDNA-Viren handelt (Baltimore-Gruppe 2). Ihr kleines zirkuläres Genom von 6.558 nt (Nukleotiden oder Basen) mit einem GC-Gehalt von 31,2 % unterstützt die Klassifizierung als schwanzlosen ssDNA-Phagen. Die Vorhersage der Offenen Leserahmen (ORFs) ergab 13 Gene, die hauptsächlich für Strukturproteine kodieren. Darüber hinaus wurden ein Replikationsinitiationsfaktor (REP) und ein Lyseprotein (N-Acylmuramoyl-L-Alanin-Amidase) identifiziert.
Gattung Cebaduodecimvirus
Das exempalrische Virus der Spezies Cebaduodecimvirus phi12duo, der Cellulophaga-Phage phi12:2 (ϕ12:2), wurde im Jahr 2005 mit seinem Wirt Cellulophaga sp. #12 (synonym Cellulophaga baltica #12) aus der Meeresenge Öresund isoliert. Die Virionen haben ein ikosaedrisches Kapsid, sind ebenfalls ohne Schwanz und ein haben ssDNA-Genom mit eine Länge von 6.453 nt. Die Omtje-Cellulophaga-Phagen (d. h. exemplarisch Omtje_1) ähneln zu 57,6 % dem Cellulophaga-Phagen phi12:2.
Ebenfalls aus dem Öresund isdoliert wurde Cebaduodecimvirus phi12auna mit dem exemplarischen Cellulophaga-Phagen phi12a:1 (ϕ12a:1) und den zwei weiteren Vertretern, phi18:4 (ϕ18:4) und phi48:1 (ϕ48:1).
Der Obscuriviridae-Vertreter Cellulophaga-Phage phi12a:1 (ϕ12a:1) ist nicht zu verwechseln mit dem Cellulophaga-Phagen phi12:1 (ϕ12:1), dem exemplarischen Vertreter der Spezies Helsingorvirus Cba121, zu der auch Cellulophaga-Phage phi12:3 (ϕ12:3) gehört.[7] Die Gattung Helsingorvirus umfasst zwei weitere Spezies, Helsingorvirus Cba171 (mit Cellulophaga-Phage phi17:1) und Helsingorvirus Cba181 (mit Cellulophaga-Phage phi18:1 und phi18:2); auch diese Phagen stammen von derselben Lokalität. Die Virionen dieser Gattung sind jedoch vom Morphotyp der Siphoviren mit langem „Schwanz“ und haben ein dsDNA-Genom. Sie gehören damit der Klasse Caudoviricetes an.[2]
Systematik
Äußere Systematik
Möglicherweise aufgrund des gleichen Fundorts und derselben Wirtsgattung (oder gar -spezies) lautete der Vorschlag zur Familie Obscuriviridae ursprünglich wie bei den Assiduviridae ebenfalls auf Zugehörigkeit zur Klasse Caudoviricetes (Titel!),[4] was aber zurückgenommen wurde, so dass die Familie jetzt offiziell incertae sedis ist.[4][2]
Die Taxonomie des National Center for Biotechnology Information (NCBI) hatte die Vertreter der Obscuriviridae – wohl aufgrund des kleinen zirkulären ssDNA-Genoms – ursprünglich der Gattung Microviridae zugeordnet.[8][9][10] Karin Holmfeldt et al. hatten diese Zuordnung 2013 bereits für Cellulophaga-Phage phi18:4 und die ihn enthaltene Gattung vorgeschlagen.[11] Ein Replikationsinitiationsfaktor (REP) ist typisch für Viren des Phylums Cressdnaviricota (CressDNA-Viren, circular REP-encoding ssDNA viruses). Microviridae und Cressdnaviricota gehören beide dem Realm Monodnaviria an. Nach einer phylogenetischen Analyse von Nina Bartlau et al. (2022) könnte allerdings ein anderer Flavophage, der der Flavobacterium-Phage FLiP (Familie Finnlakeviridae, Realm Varidnaviria) der nächste bekannte Verwandte sein.[3]
Innere Systematik
Innere Systematik der Familie (Stand Anfang April 2025):[1][2][12]
Familie Obscuriviridae (früher Cellulophaga-Phage-Gruppe 11[11])
- Gattung Cebaduodecimvirus (früher Cba184likevirus[11])
- Spezies Cebaduodecimvirus phi12auna (früher zu Microviridae, NCBI Taxid: 1327987),[8] mit
- Cellulophaga-Phage phi12a:1 (ϕ12a:1, Zugriffsnr.: KC821623)[11]
- – Fundort: Öresund, Südl. Kattegat, Dänemark im 1 Meter Tiefe, Probennahme am 31. Mai 2005.
- – Wirt: Cellulophaga baltica OL12a
- Cellulophaga phage phi18:4 (ϕ18:4, Zugriffsnr.: KC821628)
- – Fundort: Öresund, Südl. Kattegat, Dänemark im 1 Meter Tiefe, Probennahme am 31. Mai 2005.
- – Wirt: Cellulophaga baltica #18 [syn. Cellulophaga sp. #18]
- Cellulophaga phage phi48:1 (ϕ48:1, Zugriffsnr.: KC821631)
- – Fundort: Öresund, Südl. Kattegat, Dänemark im 1 Meter Tiefe, Probennahme am 31. Mai 2005.
- – Wirt: Cellulophaga baltica NN016048
- Spezies Cebaduodecimvirus phi12duo (früher zu Microviridae, NCBI Taxid: 1327969),[9] mit
- Cellulophaga-Phage phi12:2 (ϕ12:2, Zugriffsnr.: KC821606)[11]
- – Fundort: Öresund, Südl. Kattegat, Dänemark im 1 Meter Tiefe, Probennahme am 31. Mai 2005.
- – Wirt: Cellulophaga baltica #12 [syn. Cellulophaga sp. #12]
- Spezies Cebaduodecimvirus phi12auna (früher zu Microviridae, NCBI Taxid: 1327987),[8] mit
- Gattung Omtjevirus
- Spezies Omtjevirus Omtje (früher zu Microviridae, NCBI Taxid: 2745694),[10] mit
- Cellulophaga-Phage Omtje_1 (Zugriffsnr.: MT732445)[3]
- – Fundort: SW vom Dünenhafen, Insel Düne, Helgoland, Nordsee. Probennahme am 14. März 2017.
- – Wirt: Cellulophaga sp. HaHaR_3_176 DSM 111152
- – Fundort/-datum und Wirt: wie Omtje_1
- – Fundort und Wirt: wie Omtje_1, Probennentnahme jedoch 2017/2018
- – Fundort/-datum und Wirt: wie Omtje_1
- – Fundort/-datum und Wirt: wie Omtje_1
- Spezies Omtjevirus Omtje (früher zu Microviridae, NCBI Taxid: 2745694),[10] mit
Die Akronyme folgen Karin Holmfeldt et al. (2013).[11]
Namensherkunft (Etymologie)
Der Name der Familie Obscuriviridae kommt von lateinisch obscurus ‚unsicher‘, ‚mehrdeutig‘ und bezieht sich auf den ungeklärten Status der Familie (Stand März 2025) und weil die Bedeutung dieser Familie noch nicht geklärt ist (Stand September 2024); der Suffix ‚-viridae‘ kennzeichnet Virusfamilien.[14][4]
Der Gattungsname Omtjevirus stammt vom exemplarischen Virus, dem Cellulophaga-Phagen Omtje_1. Omtje ist ein Vorname friesischen Ursprungs, der Sprache, die auf Helgoland gesprochen wird, wo das Virus in den umliegenden Meeresgewässern gefunden wurde.[4]
Der Name der Gattung Cebaduodecimvirus stammt von dem Wirt Cellulophaga baltica und verweist auf sein zwölftes Virus, lateinisch duodecim ‚zwölf‘.[4]
Einzelnachweise
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b c d ICTV: Master Species Lists (MSL).
- ↑ a b c d e f g h Nina Bartlau, Antje Wichels, Georg Krohne, Evelien M. Adriaenssens, Anneke Heins, Bernhard M Fuchs, Rudolf Amann, Cristina Moraru: Highly diverse flavobacterial phages isolated from North Sea spring blooms. In: Oxford Academic: The ISME Journal, Band 16, Nr. 2, Februar 2022, S. 555–568; doi:10.1038/s41396-021-01097-4, PMC 8776804 (freier Volltext), PMID 34475519, Epub 2. September 2021 (englisch).
- ↑ a b c d e f g h i j k Nina Bartlau, Cristina Moraru, Antje Wichels, Karin Holmfeldt, R. I. Amann: Create a new family, Obscuriviridae (Class: Caudoviricetes). Vorschlag 2024.025B (zip:docx), Filelist. 21. Juni 2024. Stand: 30. September 2024.
- ↑ VIRIDIC Web-Service (viridic.icbm.de).
- ↑ VirClust (virclust.icbm.de).
- ↑ NCBI Taxonomy Browser: Cellulophaga phage phi12:1 (als Spezies). Details: Cellulophaga phage phi12:1, Linieage: … unclassified Microviridae.
- ↑ a b NCBI Taxonomy Browser: Cellulophaga phage phi12a:1 (als Spezies). Details: Cellulophaga phage phi12a:1, Linieage: … Helsingorvirus Cba121.
- ↑ a b NCBI Taxonomy Browser: Cellulophaga phage phi12:2, Linieage: … unclassified Microviridae.
- ↑ a b NCBI Taxonomy Browser: Cellulophaga phage Omtje_1, Linieage: … unclassified Microviridae.
- ↑ a b c d e f Karin Holmfeldt, Natalie Solonenko, Manesh Shah, Kristen Corrier, Lasse Riemann, Nathan C. Verberkmoes, Matthew B. Sullivan: Twelve previously unknown phage genera are ubiquitous in global oceans, In: PNAS, Band 110, Nr. 31, 30. Juli 2013, S. 12798–12803; doi:10.1073/pnas.1305956110, PMC 3732932 (freier Volltext), PMID 23858439, ResearchGate:249649168, Epub 15. Juli 2013 (englisch).
- ↑ NCBI Nucleotide: txid1327987[Organism:exp] OR txid1327987[Organism:exp] OR txid2745694[Organism:exp].
- ↑ DSMZ: Cellulophaga phage Omtje_1 DSM 111240.
- ↑ ICTV: Family: Obscuriviridae.