Lindbergviridae
| Lindbergviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
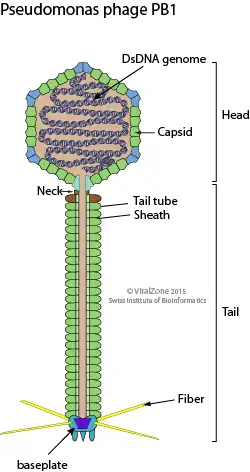
Gattung Pbunavirus (Schemazeichnung Querschnitt). | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Lindbergviridae | ||||||||||||||
| Links | ||||||||||||||
Lindbergviridae ist eine im Mãrz 2025 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp A (Myoviren). Ihre Wirte sind Bakterien, was sie als Bakteriophagen klassifiziert.[1][2]
Beschreibung
Bis zur Einrichtung der Familie im März 2025 wurden PB1-ähnliche Phagen in die folgenden Gattungen eingeteilt: Kylevirus, Tabernariusvirus, Bcepfunavirus, Pbunavirus, Wifcevirus, Myosmarvirus und Carpasinavirus. Dabei handelt es sich um Myoviren, die Mitglieder der Betaproteobakterien und Gammaproteobakterien infizieren. Mit der Schaffung der Familie Lindbergviridae wurden neben diesen Gattungen zusätzlich als neue Gattungen Kylevirus, Tabernariusvirus und Bcepfunavirus mit in sie aufgenommen. Genom- und Proteom-Analysen hatten gezeigt, dass die zuvor etablierten Gattungen zusammen mit den drei neuen zu einer neuen Familie gehören. Diese Bakteriophagen haben 12 gemeinsame Proteine.[3]
Die Viren dieser Familie infizieren bakterielle Wirte verschiedener Gattungen zumeist der Gammaproteobacteria:[4]
- Ordnung Enterobacterales
- Pantoea[5] (Erwiniaceae)
- Serratia (Yersiniaceae)
- Escherichia (Enterobacteriaceae)
- Ordnung Lysobacterales[6]
- Xanthomonas (Lysobacteraceae)
- Luteibacter[7] (Rhodanobacteraceae)
- Ordnung Pseudomonadales
- Pseudomonas (Pseudomonadaceae)
Lediglich die Mitglieder der Gattung Bcepfunavirus infizieren Betaproteobakterien, nämlich die der Gattung Burkholderia (Burkholderiales; Burkholderiaceae).[4]
Da unter diesen Wirtsbakterien etliche Krankheitserreger von Wirbeltieren, insbesondere des Menschen, vorkommen, sind deren Phagen Kandidaten für eine mögliche Phagentherapie – vorausgesetzt, sie sind lytisch, d. h. wenn sie ihre Wirtsbakterien zerstören. Beispiele für solche lytischen Phagen sind die derzeit (Stand März 2025) vom ICTV noch nicht bestätigten Kandidaten Pseudomonas-Phage AA20 („Pseudomonas-Phage vB_PaeM_HTN3“) und AC20 („Pseudomonas-Phage vB_PaeM_HTN4“), deren Wirt Pseudomonas aeruginosa mit zu den Verursachern schwerer Verläufe der Mukoviszidose (englisch cystic fibrosis, CF) gehört und sich oft durch Resistenz gegenüber Antibiotika auszeichnet. Zusammen mit diesen beiden Kandidaten wurden als lytische Phagen von P. aeruginosa noch gefunden: U17 „Pseudomonas-Phage vB_PaeP_HTN1“ (Bruynoghevirus, Podoviren) und AA17 „Pseudomonas-Phage vB_PaeS_HTN2“ (Iggyvirus, Siphoviren).[8]
Infektionsmechanismus

Genom

Systematik
Die Systematik der Lindbergviridae ist nach International Committee on Taxonomy of Viruses (ICTV), ergänzt um einige Vorschläge nach der Taxonomie des National Center for Biotechnology Information (NCBI) in Hochkommata mit Stand 1. April 2025 (MSL#40v1) wie folgt:[1][4]
Familie Lindbergviridae (früher PB1-ähnliche Phagen)
- ohne Zuordnung einer Unterfamilie
- Gattung Bcepfunavirus
- Spezies Bcepfunavirus bcepF1 (Burkholderia virus BcepF1), mit Burkholderia-phage BcepF1
- Gattung Carpasinavirus (Xanthomonas-Phagen)
- Spezies Carpasinavirus carpasina, mit Xanthomonas-Phage Carpasina
- Spezies Carpasinavirus FoX6, mit Xanthomonas-Phage FoX6
- Spezies Carpasinavirus XcP1 (Xanthomonas-Virus Carpasina), mit Xanthomonas-Phage XcP1
- Gattung Gladiolivirus (abgetrennt von Bcepfunavirus)
- Spezies Gladiolivirus maja, mit Burkholderia-Phage Maja – unterscheide Arthrobacter-Phage Maja (Majavirus maja)
- Gattung Irusalimvirus
- Spezies Irusalimvirus BCSR52, mit Burkholderia-Phage BCSR52
- Gattung Kylevirus
- Spezies Kylevirus kyle (Panteoa-Virus Kyle), mit Panteoa phage Kyle (sic!) – Wirt: Pantoea sp. (Buchstaben-Dreher)
- Gattung Myosmarvirus (Serratio-Phagen, 3 Spezies)
- Spezies Myosmarvirus MTx, mit Serratia-Phage MTx
- Spezies Myosmarvirus myosmar (Serratia-Virus MyoSmar), mit Serratia-Phage MyoSmar
- Spezies Myosmarvirus SMP, mit Serratia-Phage SMP
- Gattung Bcepfunavirus
- Gattung Pbunavirus (früher Pbunalikevirus, PB1likevirus, Pseudomonas-Phagen, Myoviren, 45 Spezies)[10]
- Spezies Pbunavirus Ab28, mit Pseudomonas-Phage vB_PaeM_C1-14_Ab28
- Spezies Pbunavirus antinowhere, mit Pseudomonasbbnantinowhere
- Spezies Pbunavirus crassa, mit Pseudomonas-Phage crassa
- Spezies Pbunavirus datas, mit Pseudomonas-Phage datas
- Spezies Pbunavirus E217, mit Pseudomonas-Phage vB_PaeM_E217 alias E217
- Spezies Pbunavirus F8, mit Pseudomonas-Phage F8
- Spezies Pbunavirus NH4, mit Pseudomonas-Phage NH-4
- Spezies Pbunavirus PA01, mit Pseudomonas-Phage PA01
- Spezies Pbunavirus Pa193, mit Pseudomonas-Virus Pa193 (ICTV) alias Pseudomonas-Phage Pa193 (Iglesias et&mbsp;al., 2024)[11]
- Spezies Pbunavirus PA5, mit Pseudomonas-Phage PA5
- Spezies Pbunavirus PA8P1, mit Pseudomonas-Phage PA8P1 und Pseudomonas-Phage vB_PaeM_SMS29 alias SMS29
- Spezies Pbunavirus PB1 (Pseudomonas-Virus PB1), mit Pseudomonas-Phage PB1
- Spezies Pbunavirus PS44, mit Pseudomonas-Phage vB_Pae_PS44
- Spezies Pbunavirus S1, mit Pseudomonas-Phage vB_PaeM_SCUT-S1
- Spezies Pbunavirus SN (Pseudomonas-Virus SN), mit Pseudomonas-Phage SN[12]
- Spezies Pbunavirus TH15, mit Pseudomonas-Phage TH15
- Spezies Pbunavirus USP1, mit Pseudomonas-Phage vB_PaeM_USP_1
- Spezies Pbunavirus victoria, mit Pseudomonas-Phage Victoria
- Spezies …
- Spezies „Pseudomonas-Phage vB_PaeM_HTN3“, mit Pseudomonas-Phage AA20[8]
- Spezies „Pseudomonas-Phage vB_PaeM_HTN4“, mit Pseudomonas-Phage AC20[8]
- Gattung Pbunavirus (früher Pbunalikevirus, PB1likevirus, Pseudomonas-Phagen, Myoviren, 45 Spezies)[10]
- Gattung Plutovirus
- Spezies Plutovirus pluto, mit Luteibacter-Phage vB_LflM-Pluto
- Gattung Tabernariusvirus
- Spezies Tabernariusvirus tabernarius (Pseudomonas-Virus tabernarius), mit Pseudomonas-Phage tabernarius
- Gattung Wifcevirus (Escherichia-Phagen), 10 Spezies
- Spezies Wifcevirus AV128, mit Escherichia-Phage AV128
- Spezies Wifcevirus EC150, mit Escherichia-Phage EC150
- Spezies Wifcevirus ECML117, mit Escherichia-Phage ECML-117
- Spezies Wifcevirus ECO71P1, mit Escherichia-Phage ECO71P1
- Spezies Wifcevirus FEC19, mit Escherichia-Phage FEC19
- Spezies Wifcevirus mansfield, mit Escherichia-Phage Mansfield
- Spezies Wifcevirus Ro157lw, mit Escherichia-Phage vB_EcoM-Ro157lw
- Spezies Wifcevirus SP13, mit Escherichia-Phage vB_EcoM_SP13
- Spezies Wifcevirus WFC (Escherichia-Virus WFC), mit Escherichia-Phage vB_EcoM_WFC
- Spezies Wifcevirus WFH, mit Escherichia-Phage vB_EcoM_WFH
- Gattung Plutovirus
Bildergalerie
-
-
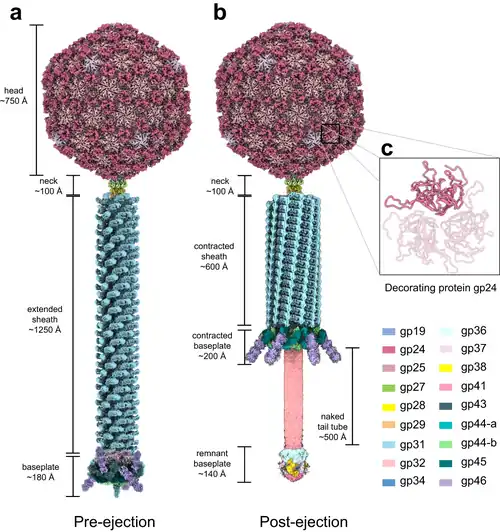 Cryo-EM von Pseudomonas-Phage E217 (Pbunavirus E217), in der Konformation vor (a) und nach (b) dem DNA-Ausstoß. (c) Zoom-Ansicht des trimeren Kapsidproteins gp25
Cryo-EM von Pseudomonas-Phage E217 (Pbunavirus E217), in der Konformation vor (a) und nach (b) dem DNA-Ausstoß. (c) Zoom-Ansicht des trimeren Kapsidproteins gp25 -

-
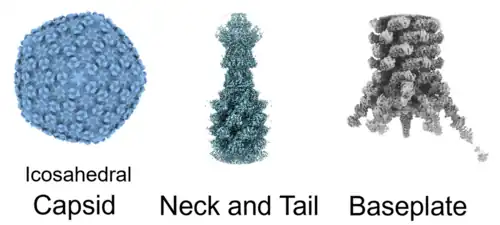
![Cryo-EM von Pseudomonas-Phage Pa193 (Pbunavirus Pa193), Virion mit gestrecktem Schwanz.[A. 1]](./42003_2024_6985_Fig1a.jpg)
-
-
![Cryo-EM von Pseudomonas-Phage Pa193 (Pbunavirus Pa193), Kapsid: höhere Auflösung, zu sehen 3 fünffach-Vertices.[A. 2]](./42003_2024_6985_Fig3ab.png)
-
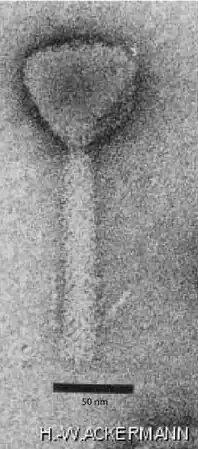
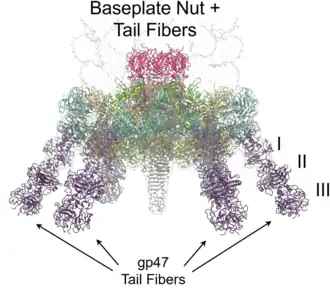 Basisplatte und Schwanzfibern von Pseudomonas-Phage Pa193 (Pbunavirus Pa193) im Detail
Basisplatte und Schwanzfibern von Pseudomonas-Phage Pa193 (Pbunavirus Pa193) im Detail
Namensherkunft (Etymologie)
Mit dem Familiennamen Lindbergviridae wird der schwedische Arzt und Wissenschaftler Alf Erik Anton Lindberg[13] (1939–2021[14]) geehrt; der Suffix ‚-viridae‘ kennzeichnet Virusfamilien.[2][3] Alf Erik Anton Lindberg promovierte 1971 am Karolinska Institutet[15] und war als Professor für klinische Bakteriologie am Huddinge-Krankenhaus (dem Karolinska-Universitätskrankenhaus) und bei der Nobel-Stiftung tätig. Seit 1989 war er Mitglied der Schwedischen Akademie der Wissenschaften. Von 1991 bis 1992 war er Sekretär des Nobelkomitees am Karolinska-Institut.[16] Eine Liste seiner Veröffentlichungen findet sich bei ResearchGate.[17]
Anmerkungen
- ↑ Man erkennt drei der fünffach-Vertices, an einem weiteren unten ist der Schwanz befestigt.
- ↑ Der Schwanz ist an einem solchen Vertex befestigt.
Einzelnachweise
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Family: Lindbergviridae .
- ↑ a b Liliana Cristina Moraru, Igor Tolstoy, Andrew M. Kropinski: Create a new family, Lindbergviridae, for PB1-like phages (Class: Caudoviricetes). Vorschlag 2024.020B (zip:docx), Filelist. 27. April 2024, Stand: 30. September 2024.
- ↑ a b c ICTV: Master Species Lists (MSL).
- ↑ LPSN: Genus Pantoea Gavini et al. 1989.
- ↑ LPSN: Order Lysobacterales Christensen and Cook 1978.
- ↑ LPSN: Genus Luteibacter Johansen et al. 2005.
- ↑ a b c d e Hanzada T. Nour El-Din, Maryam Kettal, José C. Granados Maciel, Greg Beaudoin, Umut Oktay, Sabahudin Hrapovic, Subash Sad, Jonathan J. Dennis, Danielle L. Peters, Wangxue Chen: Isolation, Characterization, and Genomic Analysis of Bacteriophages Against Pseudomonas aeruginosa Clinical Isolates from Early and Chronic Cystic Fibrosis Patients for Potential Phage Therapy. In: MDPI: Microorganisms, Band 13, Nr.&nbwp;3, 26. Februar 2025, S. 511; doi:10.3390/microorganisms13030511 (englisch).
- ↑ Yangyijun Guo, Ping Chen, Zhanglin Lin, Tingting Wang: Characterization of Two Pseudomonas aeruginosa Viruses vB_PaeM_SCUT-S1 and vB_PaeM_SCUT-S2. In: MDPI: Viruses, Band 11, Nr. 4, 1. April 2019, S. 318; doi:10.3390/v11040318 (englisch).
- ↑ SIB: Pbunavirus. Auf: ViralZone.
- ↑ Stephano M. Iglesias, Chun-Feng David Hou, Johnny Reid, Evan Schauer, Renae Geier, Angela Soriaga, Lucy Sim, Lucy Gao, Julian Whitelegge, Pierre Kyme, Deborah Birx, Sebastien Lemire, Gino Cingolani: Cryo-EM analysis of Pseudomonas phage Pa193 structural components. In: Nature: Communications Biology, Band 7, 6. Oktober 2024, S. 1275; doi:10.1038/s42003-024-06985-x (englisch).
- ↑
Ramon Sanchez-Rosario, Jesus Garcia, Vivian Rodriguez, Kevin A. Schug, Zacariah L. Hildenbrand, Ricardo A. Bernal: Using Bacteriophages to Treat Resilient Bacteria Found in Produced Water. In: MDPI Water, Band 16, Nr. 6; 7. März 2024; doi:10.3390/w16060797 (englisch). Dazu:
- A virus could help save billions of gallons of wastewater produced by fracking. Auf: EurekAlert! vom 30. April 2024.
- ↑ Wikidata: Alf A. Lindberg (Q5953343).
- ↑ Nachruf im Svenska Dagbladet, 23. Mai 2021, S. 76 (schwedisch).
- ↑ Alf A. Lindberg: Bacteriophage attachment and inactivation in relation to chemical composition of Salmonella lipopolysaccharides. Stockholm, 1971. Libris:8081422 (englisch).
- ↑ Königlich Schwedische Akademie der Wissenschaften, Matrikel 1991, ISSN 0302-6558, S. 66.
- ↑ ResearchGate: Alf A. Lindberg’s research while affiliated with Karolinska Institutet and other places (englisch).
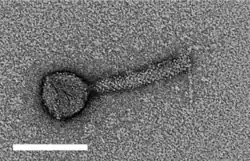
![TEM-Aufnahme der lytischen Pseudomonas-Phagen AA20 („Pseudomonas-Phage vB_PaeM_HTN3“)[8]](./Microorganisms-13-00511-g003c-AA20.png)
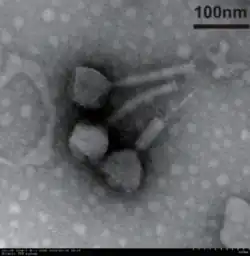
![TEM-Aufnahme der lytischen Pseudomonas-Phagen AC20 („Pseudomonas-Phage vB_PaeM_HTN4“)[8]](./Microorganisms-13-00511-g003d-AC20.png)