α-Ketoglutarat-Dehydrogenase-Komplex
Der α-Ketoglutarat-Dehydrogenase-Komplex oder 2-Oxoglutarat-Dehydrogenase-Komplex (OGDC) ist ein Proteinkomplex in Eukaryoten und manchen Bakterien, der die oxidative Decarboxylierung von α-Ketoglutarat und die nachfolgende Umsetzung mit Coenzym A zum Succinyl-CoA katalysiert. Diese Reaktion ist Teil des Citratzyklus. Die Gesamtreaktion lautet:
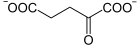 + CoA-SH + NAD+
+ CoA-SH + NAD+ 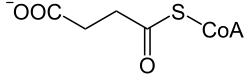 + CO2 + NADH
+ CO2 + NADH
| Übergeordnet |
|---|
| Mitochondrium |
| Gene Ontology |
| QuickGO |
Aufbau
Am α-Ketoglutarat-Dehydrogenase-Komplex sind drei Enzyme beteiligt:
| Untereinheit | EC-Nummer | Name | Gen | Cofaktor |
|---|---|---|---|---|
| E1 | EC 1.2.4.2 | α-Ketoglutarat-Dehydrogenase | OGDH | Thiaminpyrophosphat (TPP) |
| E2 | EC 2.3.1.61 | Dihydrolipoamid-Succinyltransferase | DLST | Liponsäure, Coenzym A |
| E3 | EC 1.8.1.4 | Dihydrolipoamid-Dehydrogenase | DLD | FAD, NAD |
Der α-Ketoglutarat-Dehydrogenase-Komplex bildet zusammen mit dem Pyruvat-Dehydrogenase-Komplex, dem 2-Oxoadipat-Dehydrogenase-Komplex (OADHC) und dem verzweigtkettigen α-Ketosäure-Dehydrogenase-Komplex (BCKDC) eine Familie von Multienzymkomplexen sogenannter α-Ketosäure-Dehydrogenase-Komplexe.[1] Alle vier Komplexe verwenden dieselbe E3-Untereinheit und dieselben Cofaktoren.[1] OGDC und OADHC nutzen sogar noch die gleiche E2-Untereinheit (DLST).[2][3] Auch zum Glycinspaltungssystem besteht eine biochemische Verwandtschaft, da es ebenfalls dieselbe E3-Untereinheit mit den zugehörigen Cofaktoren sowie zusätzlich den gemeinsamen Cofaktor Liponsäure nutzt.[4]
| Untereinheiten OGDC | Untereinheiten OADHC | Untereinheiten PDC |
|---|---|---|
| α-Ketoglutarat-Dehydrogenase (E1) | 2-Oxoadipat-Dehydrogenase (E1) | Pyruvatdehydrogenase (E1) |
| Dihydrolipoamid-Succinyltransferase (E2) | Dihydrolipoyl-Transacetylase (E2) | |
| Dihydrolipoamid-Dehydrogenase (E3) | ||
Im aktiven Multienzymkomplex ist die Succinyltransferase zu hochsymmetrischen Multimeren aggregiert (Tetracosamer der Punktgruppe 432, Hexacontamer der Punktgruppe 532), an welche die verschiedenen Dehydrogenasen gebunden sind.
In Eukaryoten wurde eine einzige Variante des Komplexes in Mitochondrien identifiziert.
Reaktion

OGDC und PDC nutzen die gleichen Coenzyme und katalysieren auch eine analoge Reaktion, auch der Reaktionsmechanismus ist bei beiden Komplexen sehr ähnlich.[5] α-Ketoglutarat wird oxidativ zu Succinyl-CoA decarboxyliert, dabei entsteht NADH. Im Citratzyklus wird dieses in die Atmungskette eingespeist und dient zur Energieerzeugung.
Klinische Relevanz
Bei der Stoffwechselkrankheit der kombinierten Malon- und Methylmalonazidurie (CMAMMA) aufgrund ACSF3-Mangels ist die mitochondriale Fettsäuresynthese (mtFAS) gestört, die die Vorläuferreaktion der Liponsäurebiosynthese darstellt.[6] Die Folge ist ein verminderter Lipoylierungsgrad von wichtigen mitochondrialen Enzymen, wie unter anderem des α-Ketoglutarat-Dehydrogenase-Komplexes (OGDC) und des Pyruvat-Dehydrogenase-Komplexes.[6]
Bildung von Succinat in Prokaryoten
Veränderte Citratzyklus-Stoffwechselwege, in denen ein Teilschritt fehlt, sind bei Bakterien der Normalfall (13 von 17 untersuchte). Der fehlende Schritt kann durch andere Reaktionsschritte ersetzt sein oder auch nicht. Tatsächlich sind nur von drei Bakterienarten Enzyme mit Ketoglutarat-Dehydrogenase-Aktivität (KDH) bekannt: Euglena gracilis, Bacillus japonicum und Escherichia coli.- Das Bakterium Escherichia coli fährt unter aeroben Bedingungen den kompletten Citratzyklus wie beschrieben. Unter anaeroben Bedingungen ist es in der Lage, die KDH zu deaktivieren. Die Stoffwechselwege, die vorher einen Kreis bildeten, sind nun baumstrukturartig verbunden. M. tuberculosis hingegen kann zwischen zwei verschiedenen Citratzyklen umschalten, die beide vom eukaryotischen Weg verschieden sind.[7]
Archaeen, aber auch manche Bakterien, wie Helicobacter pylori, das unter microaerophilen Bedingungen wächst, katalysieren die Umwandlung von α-Ketoglutarat zu Succinyl-CoA mittels einer oxidationsempfindlichen 2-Oxoglutarat:Ferredoxin-Oxidoreduktase (OGOR, EC 1.2.7.3). Im Gegensatz zur OGDC enthält diese Eisen-Schwefel-Cluster; es fehlen das Flavin und das Liponsäureamid. Anstatt NADH wird Ferredoxin als Reduktionsmittel genutzt. Auch Mycobacterium tuberculosis enthält ein CoA-abhängiges Enzym, das dagegen auch unter aeroben Bedingungen stabil ist.[8][9][10]
Bei verschiedenen Mycobakterien (darunter auch Mycobacterium tuberculosis) ist die E1-Untereinheit der Ketoglutarat-Dehydrogenase durch eine Ketoglutarat-Decarboxylase ersetzt, die unabhängig von Coenzym A zunächst Succinat-Semialdehyd produziert, welches von einer NADP+-abhängigen Succinat-Semialdehyd-Dehydrogenase zu Succinat dehydriert wird.[11]
Literatur
- Bunik VI, Fernie AR: Metabolic control exerted by the 2-oxoglutarate dehydrogenase reaction: a cross-kingdom comparison of the crossroad between energy production and nitrogen assimilation. In: Biochem. J. 422. Jahrgang, Nr. 3, September 2009, S. 405–21, doi:10.1042/BJ20090722, PMID 19698086.
- Kabysheva MS, Storozhevykh TP, Pinelis VG, Bunik VI: Synthetic regulators of the 2-oxoglutarate oxidative decarboxylation alleviate the glutamate excitotoxicity in cerebellar granule neurons. In: Biochem. Pharmacol. 77. Jahrgang, Nr. 9, Mai 2009, S. 1531–40, doi:10.1016/j.bcp.2009.02.001, PMID 19426691.
- Bunik VI, Raddatz G, Wanders RJ, Reiser G: Brain pyruvate and 2-oxoglutarate dehydrogenase complexes are mitochondrial targets of the CoA ester of the Refsum disease marker phytanic acid. In: FEBS Lett. 580. Jahrgang, Nr. 14, Juni 2006, S. 3551–7, doi:10.1016/j.febslet.2006.05.040, PMID 16737698.
Einzelnachweise
- ↑ a b Ryan J. Mailloux: The emerging importance of the α-keto acid dehydrogenase complexes in serving as intracellular and intercellular signaling platforms for the regulation of metabolism. In: Redox Biology. Band 72, Juni 2024, S. 103155, doi:10.1016/j.redox.2024.103155, PMC 11021975 (freier Volltext) – (elsevier.com).
- ↑ Natalia S. Nemeria, Gary Gerfen, Pradeep Reddy Nareddy, Luying Yang, Xu Zhang, Michal Szostak, Frank Jordan: The mitochondrial 2-oxoadipate and 2-oxoglutarate dehydrogenase complexes share their E2 and E3 components for their function and both generate reactive oxygen species. In: Free Radical Biology and Medicine. Band 115, Februar 2018, S. 136–145, doi:10.1016/j.freeradbiomed.2017.11.018 (elsevier.com).
- ↑ Natalia S. Nemeria, Gary Gerfen, Luying Yang, Xu Zhang, Frank Jordan: Evidence for functional and regulatory cross-talk between the tricarboxylic acid cycle 2-oxoglutarate dehydrogenase complex and 2-oxoadipate dehydrogenase on the l-lysine, l-hydroxylysine and l-tryptophan degradation pathways from studies in vitro. In: Biochimica et Biophysica Acta (BBA) - Bioenergetics. Band 1859, Nr. 9, September 2018, S. 932–939, doi:10.1016/j.bbabio.2018.05.001 (elsevier.com).
- ↑ Parith Wongkittichote, Sanmati R. Cuddapah, Stephen R. Master, Dorothy K. Grange, Dennis Dietzen, Stephen M. Roper, Rebecca D. Ganetzky: Biochemical characterization of patients with dihydrolipoamide dehydrogenase deficiency. In: JIMD Reports. Band 64, Nr. 5, September 2023, ISSN 2192-8312, S. 367–374, doi:10.1002/jmd2.12382, PMC 10494496 (freier Volltext) – (wiley.com).
- ↑ H. Robert Horton, Laurence A. Moran, K. Gray Scrimgeour, Marc D. Perry, J. David Rawn und Carsten Biele (Übersetzer): Biochemie. Pearson Studium; 4. aktualisierte Auflage 2008; ISBN 978-3-8273-7312-0; S. 536
- ↑ a b Zeinab Wehbe, Sidney Behringer, Khaled Alatibi, David Watkins, David Rosenblatt, Ute Spiekerkoetter, Sara Tucci: The emerging role of the mitochondrial fatty-acid synthase (mtFASII) in the regulation of energy metabolism. In: Biochimica et Biophysica Acta (BBA) - Molecular and Cell Biology of Lipids. Band 1864, Nr. 11, November 2019, S. 1629–1643, doi:10.1016/j.bbalip.2019.07.012 (elsevier.com).
- ↑ Cordwell SJ: Microbial genomes and "missing" enzymes: redefining biochemical pathways. In: Arch. Microbiol. 172. Jahrgang, Nr. 5, November 1999, S. 269–79, PMID 10550468.
- ↑ Mai, X. und Adams, MW. (1996): Characterization of a fourth type of 2-keto acid-oxidizing enzyme from a hyperthermophilic archaeon: 2-ketoglutarate ferredoxin oxidoreductase from Thermococcus litoralis. In: Journal of bacteriology. Band 178, Nummer 20, Oktober 1996, S. 5890–5896, PMID 8830683, PMC 178443 (freier Volltext).
- ↑ Pitson SM, Mendz GL, Srinivasan S, Hazell SL: The tricarboxylic acid cycle of Helicobacter pylori. In: Eur J Biochem. 260. Jahrgang, Nr. 1, Februar 1999, S. 258–67, PMID 10091606.
- ↑ Baughn AD, Garforth SJ, Vilchèze C, Jacobs WR: An anaerobic-type alpha-ketoglutarate ferredoxin oxidoreductase completes the oxidative tricarboxylic acid cycle of Mycobacterium tuberculosis. In: PLoS Pathog. 5. Jahrgang, Nr. 11, November 2009, S. e1000662, doi:10.1371/journal.ppat.1000662, PMID 19936047, PMC 2773412 (freier Volltext).
- ↑ Tian J, Bryk R, Itoh M, Suematsu M, Nathan C: Variant tricarboxylic acid cycle in Mycobacterium tuberculosis: identification of alpha-ketoglutarate decarboxylase. In: Proc. Natl. Acad. Sci. USA. 102. Jahrgang, Nr. 30, Juli 2005, S. 10670–5, doi:10.1073/pnas.0501605102, PMID 16027371, PMC 1180764 (freier Volltext).