Andersonviridae
| Andersonviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
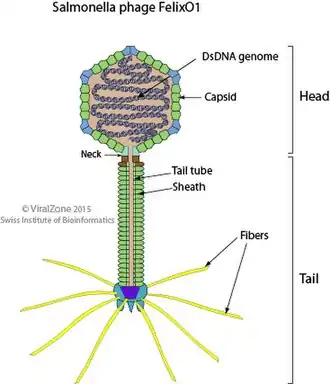
Schemazeichnung eines Virions von Salmonella-Phage FelixO1 (Felixounavirus felixO1), Querschnitt | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Andersonviridae | ||||||||||||||
| Links | ||||||||||||||
|
Andersonviridae ist eine im März 2025 als Familie eingerichtete Gruppe von Viren mit Kopf-Schwanz-Aufbau vom Morphotyp A (Myoviren). Ihre Wirte sind Bakterien, was sie als Bakteriophagen klassifiziert.[1][2]
Beschreibung
Salmonella-Phage FelixO1 wurde im Jahr 2010 sequenziert.[3]
Die Einrichtung der Familie Andersonviridae war das Resultat einer Untersuchung der evolutionären Beziehungen von 123 Bakteriophagen. Die Analyse der konservierten Gene dieser Phagen ergab, dass die FelixO1-ähnlichen Phagen eine tief verzweigte monophyletische Klade bilden, deren Abstand zu andren Phagen der Klasse Caudoviricetes die Einrichtung einer neuen Familie rechtfertigt. Im Übrigen konnten dieser Familie die bereits existierenden und bis dato innerhalb dieser Klasse noch nicht weiter klassifizierten Gattungen Felixounavirus, Kolesnikvirus und Mooglevirus (inklusive Suspvirus) zugeordnet werden.
Die Familie ist in den wichtigsten proteombasierten Clustering-Tools (VirClust, ViPTree, GRAViTy-Dendrogramm, vConTACT2-Netzwerk) durch eine zusammenhängende und monophyletische Gruppe vertreten. Die Gründungsmitglieder der Familie haben eine beträchtliche Anzahl von orthologen Genen gemeinsam; die genaue Anzahl hängt jedoch von der Genomgröße und der Anzahl der kodierenden Sequenzen der Familienmitglieder ab.[4]
Systematik
Die Systematik der Andersonviridae ist nach International Committee on Taxonomy of Viruses (ICTV) – (MSL#40v1) mit Stand 16. April 2025 wie folgt:[1][5]
Familie Andersonviridae (früher „FelixO1-ähnliche Phagen“, „Felix-O1-ähnliche Phagen“)
- Unterfamilie Ounavirinae (ursprünglich vorgeschlagen als Felixounavirinae [6]), ehem. Myoviridae
- Gattung Felixounavirus (97 Spezies, früher Felixo1virus, Felixounalikevirus[6])[7]
- Spezies Felixounavirus adrianh, mit Escherichia-Phage adrianh
- Spezies Felixounavirus Alf5, mit Escherichia-Phage vB_EcoM_Alf5
- Spezies Felixounavirus allfine, mit Escherichia-Phage allfine
- Spezies Felixounavirus andreotti, mit Escherichia-Phage Andreotti
- Spezies Felixounavirus ASO1A, mit Escherichia-Phage vB_EcoM_ASO1A
- Spezies Felixounavirus AYO145A, mit Escherichia-Phage vB_EcoM_AYO145A
- Spezies Felixounavirus barry, mit Escherichia-Phage Barry
- Spezies Felixounavirus BPS15Q2, mit Salmonella-Phage BPS15Q2
- Spezies Felixounavirus BPS17L1, mit Salmonella-Phage BPS17L1
- Spezies Felixounavirus BPS17W1, mit Salmonella-Phage BPS17W1
- Spezies Felixounavirus BPSELC1, mit Salmonella-Phage BPSELC
- Spezies Felixounavirus bumzen, mit Escherichia-Phage bumzen
- Spezies Felixounavirus CapYZU01, mit Salmonella-Phage SapYZU01
- Spezies Felixounavirus dune, mit Escherichia-Phage dune
- Spezies Felixounavirus ekra, mit Escherichia-Phage ekra
- Spezies Felixounavirus felixO1 (Salmonella-Virus FelixO1), mit Salmonella-Phage FelixO1 (alias Felix O1, Felix-O1),[3] Salmonella-Phage Felix O1 VT1 und Salmonella-Phage FO1a
- Spezies Felixounavirus finno, mit Escherichia-Phage finno
- Spezies Felixounavirus heid, mit Escherichia-Phage heid
- Spezies Felixounavirus humlepung, mit Escherichia-Phage humlepung
- Spezies Felixounavirus johannrwettstein, mit Escherichia-Phage JohannRWettstein
- Spezies Felixounavirus meda, mit Salmonella-Phage Meda
- Spezies Felixounavirus mio, mit Escherichia-Phage mio
- Spezies Felixounavirus momo, mit Escherichia-Phage momo
- Spezies Felixounavirus mushroom (Salmonella-Virus Mushroom), mit Salmonella-Phage Mushroom
- Spezies Felixounavirus nataliec, mit Escherichia-Phage nataliec
- Spezies Felixounavirus shy, mit Escherichia-Phage vB_EcoM_Shy
- Spezies Felixounavirus tribble, mit Escherichia-Phage vB_Eco_Tribble
- Spezies Felixounavirus VpaE1, mit Escherichia-Phage vB_EcoM-VpaE1 alias VpaE1[8]
- Spezies Felixounavirus warpig, mit Escherichia-Phage warpig
- Spezies Felixounavirus wV8, mit Escherichia-Phage wV8[9]
- Spezies …
- Gattung Felixounavirus (97 Spezies, früher Felixo1virus, Felixounalikevirus[6])[7]
- Gattung Kolesnikvirus (früher Ea214virus)
- Spezies Kolesnikvirus Ea214 (Erwinia-Virus Ea214), mit Erwinia-Phage phiEa21-4
- Spezies Kolesnikvirus M7 (Erwinia-Virus M7), mit Erwinia-Phage vB_EamM-M7
- Spezies Kolesnikvirus SE5, mit Salmonella-Phage SE5
- Gattung Kolesnikvirus (früher Ea214virus)
- Gattung Mooglevirus (inklusive der früheren Gattung Suspvirus)
- Spezies Mooglevirus CHB7, mit Enterobacteria-Phage CHB7
- Spezies Mooglevirus EP1, mit Escherichia-Phage EP1
- Spezies Mooglevirus Henu11, mit Shigella-Phage Henu11
- Spezies Mooglevirus KMM2, mit Citrobacter-Phage BSwM KMM2
- Spezies Mooglevirus KMM4, mit Citrobacter-Phage BSwM KMM4
- Spezies Mooglevirus M7196WT1, mit Escherichia-Phage pEC-M719-6WT.1
- Spezies Mooglevirus mistaenkt, mit Escherichia-Phage mistaenkt
- Spezies Mooglevirus moogle (Citrobacter-Virus Moogle), mit Citrobacter-Phage Moogle
- Spezies Mooglevirus mordin (Citrobacter-Virus Mordin), mit Citrobacter-Phage Mordin
- Spezies Mooglevirus PC7913, mit Salmonella-Phage PC79-13
- Spezies Mooglevirus Sf13, mit Shigella-Phage Sf13
- Spezies Mooglevirus Sf14, mit Shigella-Phage Sf14
- Spezies Mooglevirus Sf17, mit Shigella-Phage Sf17
- Spezies Mooglevirus SFPB, mit Shigella-Phage SFPB
- Spezies Mooglevirus silverhawkium, mit Shigella-Phage Silverhawkium
- Spezies Mooglevirus susp1 (früher Suspvirus SUSP1, Escherichia-Virus SUSP1), mit Escherichia-Phage phiSUSP1
- Spezies Mooglevirus susp2 (früher Suspvirus SUSP2, Escherichia-Virus SUSP2), mit Escherichia-Phage phiSUSP2
- Gattung Mooglevirus (inklusive der früheren Gattung Suspvirus)
- ohne Zuweisung einer Unterfamilie
- Gattung Arnovirus
- Spezies Arnovirus arno162, mit Pectobacterium-Phage Arno162
- Spezies Arnovirus arno18, mit Pectobacterium-Phage Arno18
- Spezies Arnovirus Wc4, mit Pectobacterium-Phage Wc4
- Gattung Arnovirus
- Gattung Daniellevirus
- Spezies Daniellevirus danielle, mit Hafnia-Phage vB_HpaM_SarahDanielle
- Spezies Daniellevirus Zyzzx, mit Hafnia-Phage vB_HpaM_Zyzzx
- Gattung Daniellevirus
Namensherkunft (Etymologie)
Die Benennung der Familie Andersonviridae ehrt den amerikanischen biophysikalischen Chemiker und Genetiker Thomas Foxen Anderson (1911–1991),[10][11][12] der Suffix ‚-viridae‘ kennzeichnet Virusfamilien.[2][4]
Die Unterfamilie Ounavirinae verweist wie auch der Gattungsname Felixounavirus auf den Salmonella-Phagen FelixO1 (italienisch una ‚eins‘).
Bildergalerie
-
![Elektronenmikroskop-Aufnahme von Partikeln des Escherichia-Phagen vB_EcoM-VpaE1 (Felixounavirus VpaE1).[8]](./Viruses-07-02932-g002A-VpaE1.png) Elektronenmikroskop-Aufnahme von Partikeln des Escherichia-Phagen vB_EcoM-VpaE1 (Felixounavirus VpaE1).[8]
Elektronenmikroskop-Aufnahme von Partikeln des Escherichia-Phagen vB_EcoM-VpaE1 (Felixounavirus VpaE1).[8] -
![Elektronenmikroskop-Aufnahme von Partikeln der Escherichia-Phagen T4(Tequatrovirus T4, Straboviridae, 1) und VpaE1 (2).[8]](./Viruses-07-02932-g002B-T4.png) Elektronenmikroskop-Aufnahme von Partikeln der Escherichia-Phagen T4(Tequatrovirus T4, Straboviridae, 1) und VpaE1 (2).[8]
Elektronenmikroskop-Aufnahme von Partikeln der Escherichia-Phagen T4(Tequatrovirus T4, Straboviridae, 1) und VpaE1 (2).[8] -
![Elektronenmikroskop-Aufnahme von Partikeln von VpaE1 mit gestrecktem (1) und kontrahiertem (2) Schwanz.[8]](./Viruses-07-02932-g002C-VpaE1.png) Elektronenmikroskop-Aufnahme von Partikeln von VpaE1 mit gestrecktem (1) und kontrahiertem (2) Schwanz.[8]
Elektronenmikroskop-Aufnahme von Partikeln von VpaE1 mit gestrecktem (1) und kontrahiertem (2) Schwanz.[8] -
![EM-Nahaufnahme eines Partikels von Escherichia-Phage vB_EcoM-VpaE1 (Felixounavirus VpaE1) mit Schwanzfibern.[8]](./Viruses-07-02932-g002D-VpaE1.png)
-
![EM-Aufnahmen des Escherichia-Phagen wV8 (Felixounavirus wV8) zeigen die typische Morphologie der Myoviren. Offene Pfeilspitzen in B weisen auf verlängerte Schwanzfasern hin, während gefüllte Pfeilspitzen auf gefaltete Schwanzfasern hinweisen. Bei mehreren Partikeln sind Kragen und Hals zu erkennen.[9]](./10.1186-1743-422X-6-41-fig1-wV8.jpg) EM-Aufnahmen des Escherichia-Phagen wV8 (Felixounavirus wV8) zeigen die typische Morphologie der Myoviren. Offene Pfeilspitzen in B weisen auf verlängerte Schwanzfasern hin, während gefüllte Pfeilspitzen auf gefaltete Schwanzfasern hinweisen. Bei mehreren Partikeln sind Kragen und Hals zu erkennen.[9]
EM-Aufnahmen des Escherichia-Phagen wV8 (Felixounavirus wV8) zeigen die typische Morphologie der Myoviren. Offene Pfeilspitzen in B weisen auf verlängerte Schwanzfasern hin, während gefüllte Pfeilspitzen auf gefaltete Schwanzfasern hinweisen. Bei mehreren Partikeln sind Kragen und Hals zu erkennen.[9]
Einzelnachweise
- ↑ a b ICTV: MSL #40.v1, 3. März 2025.
- ↑ a b ICTV: Family: Andersonviridae.
- ↑ a b c Jean M. Whichard, Lee A. Weigt, Douglas J. Borris, Ling Ling Li, Qing Zhang, Vivek Kapur, F. William Pierson, Erika J. Lingohr, Yi-Min She, Andrew M. Kropinski, Nammalwar Sriranganathan: Complete Genomic Sequence of Bacteriophage Felix O1. In: MDPI: Viruses, Band 2, Nr. 3, 9. März 2010, S. 710–730; doi:10.3390/v2030710 (englisch).
- ↑ a b Liliana Cristina Moraru, Igor Tolstoy, Andrew M. Kropinski: Create a new family, Andersonviridae for the FelixO1-like phages (Class: Caudoviricetes). Vorschlag 2024.002B (zip:docx), Filelist. 1. Juni 2024. Stand 30. September 2024.
- ↑ ICTV: Master Species Lists (MSL).
- ↑ a b Marine Henry, Louis-Marie Bobay, Anne Chevallereau, Emilie Saussereau, Pieter-Jan Ceyssens, Laurent Debarbieux: The Search for Therapeutic Bacteriophages Uncovers One New Subfamily and Two New Genera of Pseudomonas-Infecting Myoviridae. In: PLOS One, Band 10, Nr. 1, 28. Januar 2ß15, S. e0117163; doi:10.1371/journal.pone.0117163, ResearchGate:271597978 (englisch).
- ↑ SIB: Felixounavirus. Auf: ViralZone.
- ↑ a b c d e Eugenijus Šimoliūnas, Monika Vilkaitytė, Laura Kaliniene, Aurelija Zajančkauskaitė, Algirdas Kaupinis, Juozas Staniulis, Mindaugas Valius, Rolandas Meškys, Lidija Truncaitė: Incomplete LPS Core-Specific Felix01-Like Virus vB_EcoM_VpaE1. In: MDPI: Viruses, Section Bacterial Viruses, Band 7, Nr. 12, 27. November 2015, S. 6163–6181; doi:10.3390/v7122932 (englisch).
- ↑ a b Andre Villegas, Yi-Min She, Andrew M. Kropinski, Erika J. Lingohr, Amanda Mazzocco, Shivani Ojha, Thomas E. Waddell, Hans-Wolfgang Ackermann, Dianne M. Moyles, Rafiq Ahmed, Roger P. Johnson: The genome and proteome of a virulent Escherichia coli O157:H7 bacteriophage closely resembling Salmonella phage Felix O1. In: BMC: Viology Journal, Band 6, Nr. 41, 20. April 2009; doi:10.1186/1743-422X-6-41 (englisch).
- ↑ Wikidata: Thomas F. Anderson (Q7789424).
- ↑ Robert P. Perry: THOMAS FOXEN ANDERSON. In: National Academies of Sciences, Engineering, and Medicine, Biographical Memoirs, Band 87, Kapitel 4, 2005; doi:10.17226/11522 (englisch).
- ↑ Thomas Foxen Anderson Papers. American Philosophical Society Manuscript Collections Search.